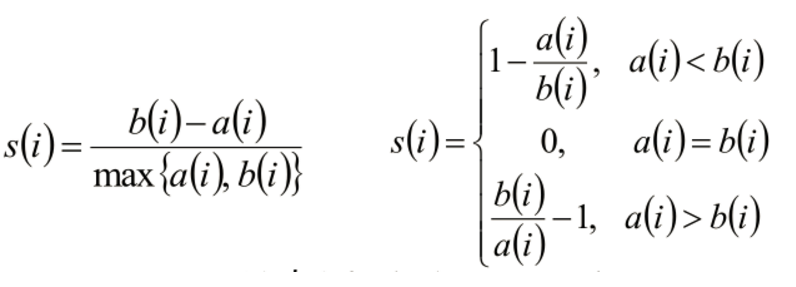基础知识:
【转载】:深入理解K-Means聚类算法 _ 转自:CSDNyqtaowhu
【转载】:第十三篇:K-Means 聚类算法原理分析与代码实现 转自:花名穆晨
【转载】:聚类算法——python实现密度聚类(DBSCAN)
Github地址:https://github.com/yjfiejd/k-means_2
k-means伪代码:
1 创建 k 个点作为起始质心 (随机选择): 2 当任意一个点的簇分配结果发生改变的时候: 3 对数据集中的每个数据点: 4 对每个质心: 5 计算质心与数据点之间的距离 6 将数据点分配到距其最近的簇 7 对每一个簇: 8 求出均值并将其更新为质心
DBSCAN伪代码:

k-means: sklearn 调包法代码
# -*- coding:utf8 -*-
# @TIME : 2018/4/11 下午10:06
# @Author : yjfiejd
# @File : Kmeans&Dbscan.py
# 1)调用sklearn调用,导入数据
import pandas as pd
import numpy as np
import matplotlib.pyplot as plt
f = open('/Users/a1/Desktop/算法实战/K-means/K-means_2/data.txt')
beer = pd.read_csv(f, sep=' ')
print(beer)
print("***********************************")
#X为所有的聚类特征,无标签数据
X = beer[["calories", "sodium", "alcohol", "cost"]]
# 2导入KMeans包,先分成N簇,然后第一轮fit后分类
from sklearn.cluster import KMeans
km = KMeans(n_clusters=3).fit(X)
km2 = KMeans(n_clusters=2).fit(X)
print("This is km.labels_:",km.labels_) #告诉你第几个数据属于哪个类别,这里共3种类别0、1、2
print("This is km2.labels_:", km2.labels_)
print("***********************************")
#新建两列在pandas中,把第一轮分类结果,扩展在pandas表中
beer['cluster1'] = km.labels_
beer['cluster2'] = km2.labels_
beer.sort_values('cluster1')
beer.sort_values('cluster2')
print(beer)
print("***********************************")
# 3)看三堆中到平均值,找下一波质心到位置
from pandas.tools.plotting import scatter_matrix
cluster_centers_1 = km.cluster_centers_
cluster_centers_2 = km2.cluster_centers_
print(beer.groupby("cluster1").mean())
print(beer.groupby("cluster2").mean())
print("***********************************")
# 4)把中心点拿出来,准备画图
centers = beer.groupby("cluster1").mean().reset_index()
plt.rcParams['font.size'] = 14
colors = np.array(['red', 'green', 'blue'])
#这里先挑选两2中数据,进行画图
#先绘制数据点,让颜色(红,绿,蓝)与cluster1中的(0类,1类,2类)对应起来
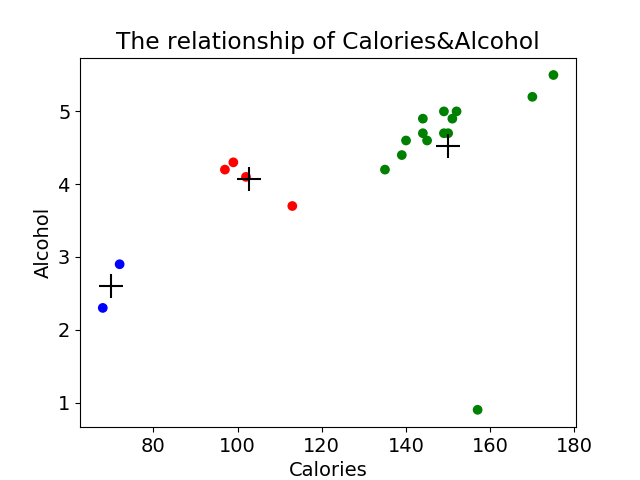
plt.scatter(beer["calories"], beer["alcohol"], c=colors[beer["cluster1"]])
#再绘制数据点中的中心点位置
plt.scatter(centers.calories, centers.alcohol, linewidths=3, marker='+', s=300, c='black')
plt.xlabel("Calories")
plt.ylabel("Alcohol")
plt.title("The relationship of Calories&Alcohol")
plt.show()
# 5)但是我们数据分别有4个维度,我们希望看俩俩之间的关系
# 先看簇1
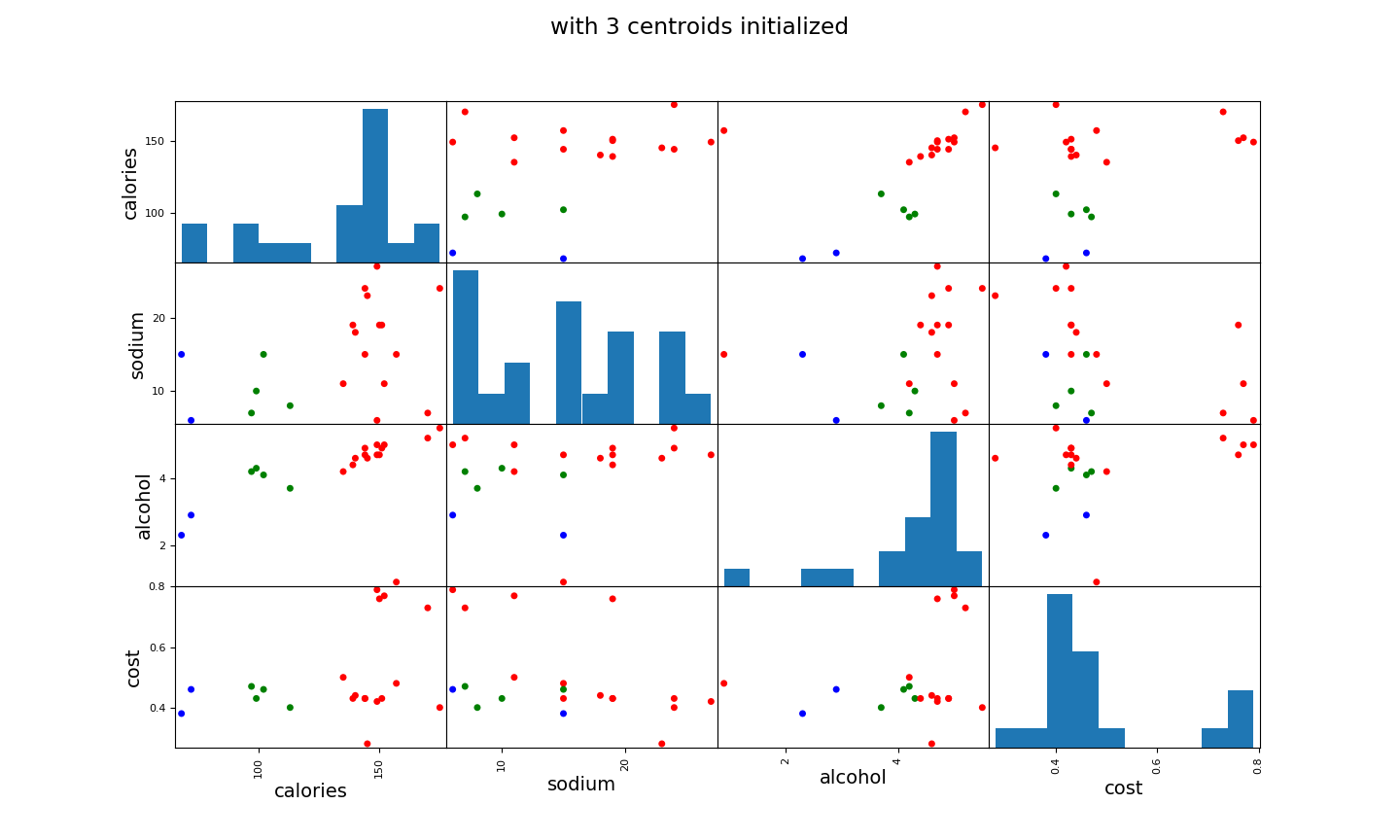
scatter_matrix(beer[["calories", "sodium", "alcohol", "cost"]], s=100, alpha=1, c=colors[beer["cluster1"]], figsize=(10, 10))
plt.suptitle("with 3 centroids initialized")
plt.show()
# 再看簇2
#scatter_matrix(beer[["calories", "sodium", "alcohol", "cost"]], s=100, alpha=1, c=colors[beer["cluster2"]], figsize=(10, 10))
#plt.suptitle("with 2 centroids initialized")
#plt.show()
print("***********************************")
# 6)Scaled data 数据预处理得先标准化一波,一般来说
# 导入标准化的包
from sklearn.preprocessing import StandardScaler
scaler = StandardScaler()
X_scaled = scaler.fit_transform(X)
print(X_scaled)
print("***********************************")
# 用KMeans的fit一下
km = KMeans(n_clusters=3).fit(X_scaled)
# pandas新建一列
beer["scaled_cluster"] = km.labels_
print(beer.sort_values("scaled_cluster"))
# 求质心均值
beer.groupby("scaled_cluster").mean()
# 画出标准化后的图像
#pd.scatter_matrix(X, c=colors[beer.scaled_cluster], alpha=1, figsize=(10,10), s=100)
# 8)聚类评估:采用轮廓系数:
## 聚类评估:轮廓系数(Silhouette Coefficient )
#<img src="1.png" alt="FAO" width="490">
#- 计算样本i到同簇其他样本的平均距离ai。ai 越小,说明样本i越应该被聚类到该簇。将ai 称为样本i的簇内不相似度。
#- 计算样本i到其他某簇Cj 的所有样本的平均距离bij,称为样本i与簇Cj 的不相似度。定义为样本i的簇间不相似度:bi =min{bi1, bi2, ..., bik}
#* si接近1,则说明样本i聚类合理
#* si接近-1,则说明样本i更应该分类到另外的簇
#* 若si 近似为0,则说明样本i在两个簇的边界上。
from sklearn import metrics
#做了标准化后的结果
score_scaled = metrics.silhouette_score(X, beer.scaled_cluster)
#没做标准化的结果
score = metrics.silhouette_score(X, beer.cluster1)
print(score_scaled, score)
print("***********************************")
# 9)遍历K值,看哪个K值合适
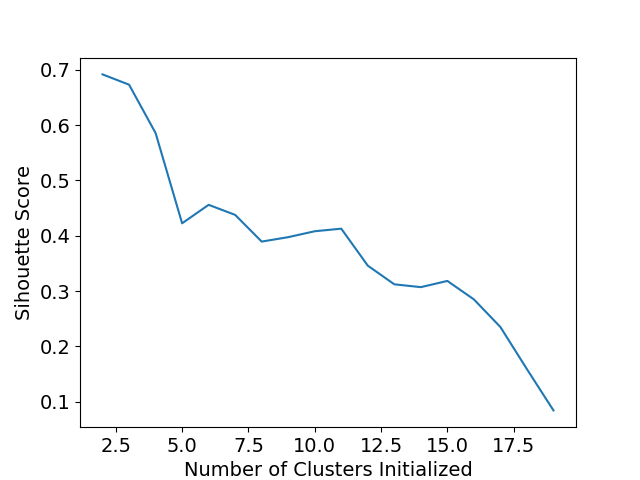
scores = []
for k in range(2,20):
labels = KMeans(n_clusters=k).fit(X).labels_
score = metrics.silhouette_score(X, labels)
scores.append(score)
print(scores)
plt.plot(list(range(2,20)), scores)
plt.xlabel("Number of Clusters Initialized")
plt.ylabel("Sihouette Score")
plt.show()
# DBSCAN clustering 算法后续补上
其中图片1:显示卡路里与酒精的关系:
plt.scatter(centers.calories, centers.alcohol, linewidths=3, marker='+', s=300, c='black')
图片2: 数据中4个维度,每两个之间的关系 (此处没有标准数据) , 后期要标准化,当然不是所有的数据标准化后结果都会变好,这个例子就是反例。
scatter_matrix(beer[["calories", "sodium", "alcohol", "cost"]], s=100, alpha=1, c=colors[beer["cluster1"]], figsize=(10, 10))
图3:聚类评价标准
- #- 计算样本i到同簇其他样本的平均距离ai。ai 越小,说明样本i越应该被聚类到该簇。将ai 称为样本i的簇内不相似度。
#- 计算样本i到其他某簇Cj 的所有样本的平均距离bij,称为样本i与簇Cj 的不相似度。定义为样本i的簇间不相似度:bi =min{bi1, bi2, ..., bik}
#* si接近1,则说明样本i聚类合理
#* si接近-1,则说明样本i更应该分类到另外的簇
#* 若si 近似为0,则说明样本i在两个簇的边界上。 - 简单来说:分子代表簇内某个点到簇内其他店的距离 a(i); 簇内某个点到其他簇样本的平均距离为b(i), 当然希望簇内点距离越小越好,簇内点到其他簇的平均距离越大越好
图片4: 因为Kmeans 非常受初始随机值影响,k的个数也需要多次实验才能选择最好的