1.在VAE生成模型中,它包括推断和生成过程,那么如果通过一个训练集训练好了整个模型,是否可以将生成模型单独使用?也就是z随机生成,隐空间随机,那么生成的数据是可用的吗?生成的数据是有意义的吗?
最简单的办法就是使用mnist生成数据,看生成的是否有规律了。
这个是下列代码生成的结果:
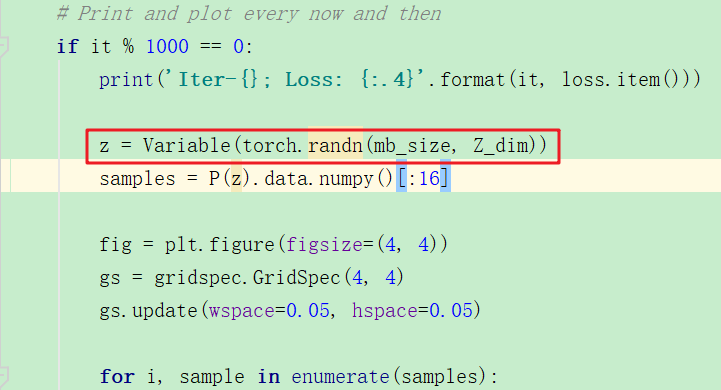
z是通过随机取样获得的,而不是在手写数据中学习到的,结果如下:

觉得效果很一般啊。
想起来那篇论文是使用生成的数据,我来看一下,

它是给定不同参数生成的数据集的。反正这里就是只要有模型参数,那么就能生成。那么在VAE中,有了模型参数,不就可以随机生成了吗?但是其实图像和RNA-seq是不同的?当然是ok的,但是这样的话就没有什么评价标准了啊。嗯是的。
下面是训练好了模型之后,随机生成z然后进行生成过程:

结果如下:

2.如果用数据集A生成了B和C,那么都是可以的吗?B和C等会因为太过相似而只能选择其中一个吗?
在splat中尝试使用同一个真实数据集,生成两次,使用不同的seed,看是否会生成同样的。
http://www.bioconductor.org/packages/release/bioc/vignettes/splatter/inst/doc/splatter.html
#我都在想些什么,,好像这没啥用啊。
> set.seed(1) > sce <- mockSCE() > sce #我尝试使用同样的sce那么,生成的数据是一样一样的。 > c0[1:5,1:5] Cell1 Cell2 Cell3 Cell4 Cell5 Gene1 27 54 141 26 107 Gene2 1 25 0 1 0 Gene3 782 738 386 334 130 Gene4 58 16 21 86 6 Gene5 11 24 18 20 34 > c1[1:5,1:5] Cell1 Cell2 Cell3 Cell4 Cell5 Gene1 27 54 141 26 107 Gene2 1 25 0 1 0 Gene3 782 738 386 334 130 Gene4 58 16 21 86 6 Gene5 11 24 18 20 34
当我设置不同的seed时,设置seed为20,然后mock生成的数据集的参数有差异,但是差异不是特别大,
> c2[1:5,1:5] Cell_001 Cell_002 Cell_003 Cell_004 Cell_005 Gene_0001 0 5 7 276 50 Gene_0002 12 0 0 0 0 Gene_0003 97 292 58 64 541 Gene_0004 0 0 0 170 19 Gene_0005 105 123 174 565 1061 > c0[1:5,1:5] Cell1 Cell2 Cell3 Cell4 Cell5 Gene1 27 54 141 26 107 Gene2 1 25 0 1 0 Gene3 782 738 386 334 130 Gene4 58 16 21 86 6 Gene5 11 24 18 20 34
看来生成的数据还是有较大差异的,因为给的参数不同。
3.由1我想到,那么scvi这么复杂,它是怎么训练的呢?训练过程是怎样的?
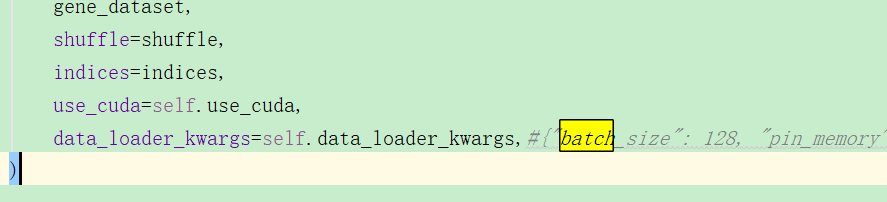
batch_size为128,然后epoch默认为400,每一次epoch都会完整地训练一次数据:
