在前一篇文章中,我们介绍了随机森林,本文我们将着重介绍其R语言的实现。
使用randomForest包中的randomForest函数
数据简介
本文数据选择了红酒质量分类数据集,这是一个很经典的数据集,原数据集中“质量”这一变量取值有{3,4,5,6,7,8}。为了实现二分类问题,我们添加一个变量“等级”,并将“质量”为{3,4,5}的观测划分在等级0中,“质量”为{6,7,8}的观测划分在等级1中。
因变量:等级
自变量:非挥发性酸性、挥发性酸性、柠檬酸、剩余糖分、氯化物、游离二氧化硫、二氧化硫总量、浓度、pH、硫酸盐、酒精
library(openxlsx)
wine = read.xlsx("C:/Users/Mr.Reliable/Desktop/classification/winequality-red.xlsx")
#将数据集分为训练集和测试集,比例为7:3
train_sub = sample(nrow(wine),7/10*nrow(wine))
train_data = wine[train_sub,]
test_data = wine[-train_sub,]
随机森林的实现
R包下载
install.packages('randomForest')
实现随机森林
randomForest函数的重要参数:
| 参数 | 意义 |
|---|---|
| formula | ~ ,确定自变量和因变量 |
| data | 使用的数据集 |
| ntree | 在森林中树的个数,默认是500 |
| mtry | 每棵树使用的特征个数 |
| importance | 是否计算变量的特征重要性,默认为False |
| proximity | 是否计算各个观测之间的相似性 |
randomForest函数的参数非常多,这里只列举了几个常用以及会对模型结果造成影响的参数。其全部参数请参考:随机森林参数
library(pROC) #绘制ROC曲线
library(randomForest)
#数据预处理
train_data$等级 = as.factor(train_data$等级)
test_data$等级 = as.factor(test_data$等级)
wine_randomforest <- randomForest(等级 ~ 非挥发性酸性+挥发性酸性+柠檬酸+
剩余糖分+氯化物+游离二氧化硫+
二氧化硫总量+浓度+pH+硫酸盐+酒精,
data = train_data,
ntree =500,
mtry=3,
importance=TRUE ,
proximity=TRUE)
这样我们就实现了随机森林算法
查看变量的重要性
#查看变量的重要性
wine_randomforest$importance
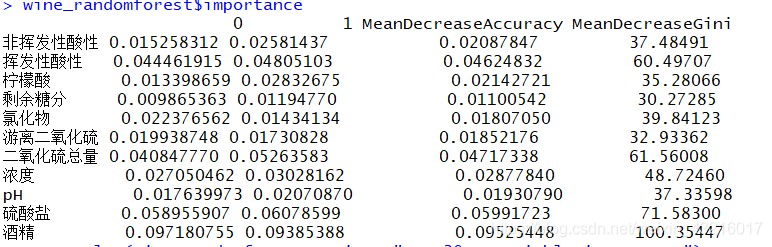 解释: 在随机森林中变量的重要性计算时通过将相应变量替换成一列随机的数后,计算模型准确率或者GINI系数的降低。上图的意思是:0:表示变量替换后对分类为0的数据的影响;1:表示表示变量替换后对分类为0的数据的影响;MeanDecreaseAccuracy:表示变量替换后准确率的下降;MeanDecreaseGini:表示变量替换后GINI系数的降低。数值越大表示变量越重要。
解释: 在随机森林中变量的重要性计算时通过将相应变量替换成一列随机的数后,计算模型准确率或者GINI系数的降低。上图的意思是:0:表示变量替换后对分类为0的数据的影响;1:表示表示变量替换后对分类为0的数据的影响;MeanDecreaseAccuracy:表示变量替换后准确率的下降;MeanDecreaseGini:表示变量替换后GINI系数的降低。数值越大表示变量越重要。
对于变量重要性的判断,也可以通过画图的方式直观显现:
varImpPlot(wine_randomforest, main = "variable importance")

ROC曲线和AUC值
#对测试集进行预测
pre_ran <- predict(wine_randomforest,newdata=test_data)
#将真实值和预测值整合到一起
obs_p_ran = data.frame(prob=pre_ran,obs=test_data$等级)
#输出混淆矩阵
table(test_data$等级,pre_ran,dnn=c("真实值","预测值"))
#绘制ROC曲线
ran_roc <- roc(test_data$等级,as.numeric(pre_ran))
plot(ran_roc, print.auc=TRUE, auc.polygon=TRUE, grid=c(0.1, 0.2),grid.col=c("green", "red"), max.auc.polygon=TRUE,auc.polygon.col="skyblue", print.thres=TRUE,main='随机森林模型ROC曲线,mtry=3,ntree=500')
1.当mtry=3,ntree=500时: AUC = 0.811
 2.当mtry=7,ntree=500时: AUC = 0.814
2.当mtry=7,ntree=500时: AUC = 0.814
 3.当mtry=3,ntree=1000时: AUC = 0.809
3.当mtry=3,ntree=1000时: AUC = 0.809

由此可见,mtry和ntree两个参数会影响模型的结果,但是其影响程度都不是很高。而且,由于行列的随机性,并不是说mtry越大(一棵CART树选择的特征越多)效果就越好,ntree越大(森林中的树越多)效果就越好。