【寻优算法】遗传算法(Genetic Algorithm) 参数寻优的python实现
本博文首先简单介绍遗传算法(GA)的基础知识,然后以为非线性SVM为例给出遗传算法多参数寻优的python实现。
一、遗传算法简介
遗传算法介绍部分主要参考(资料【1】),介绍过程中为方便大家理解相关概念,会有部分本人的理解,如有错误,请在评论区指正。
1、遗传算法由来
本人之前的两篇博文(资料【2,3】)分别介绍了如何用交叉验证方法进行单一参数和多参数寻优,交叉验证其实只是计算模型的评价指标的方法,参数的搜索过程其实是根据提前给出的取值范围进行穷举,如果参数取值范围给定的不合理,最优化参数就无从谈起。有没有一种方法,参数的取值范围可以随机设定,并在优化过程中不断变化,直到找到最优参数?
遗传算法是模拟达尔文生物进化论的自然选择和遗传学机理的生物进化过程的计算模型,是一种通过模拟自然进化过程搜索最优解的方法。
2、遗传算法名词概念
染色体(个体):在参数寻优问题中一个染色体代表一个参数,染色体的十进制数值,用于获取待寻优参数实际输入到模型中的值。
基因:每一个染色体都对应多个基因,基因是二进制数,基因的取值长度是染色体的长度。遗传算法的交叉、变异操作都是对染色体的基因进行的。
种群:初始化的染色体取值范围。
适应度函数:表征每一组染色体(每一组参数)对应的模型评价指标(本博文的python实例中,采用3-flod 交叉检验的平均值作为适应度函数值)。
假设对于两个参数的寻优,我们设定染色体长度为20,种群数为200,就是说初始化两个染色体,每个染色体初始化200个可能的值,每一个染色体的十进制数值用20位二进制数表示。
3、遗传算法中对染色体的操作
随机产生的初始化染色体取值范围中,不一定包含最优的参数取值,为了找到最优的染色体,需要不断对适应度函数值表现优秀的染色体进行操作,直到找到最优的染色体。具体操作如下:
3.1、选择
染色体选择是为了把适应度函数值表现好的染色体选出来,保持种群数量不变,就是将适应度函数值表现好的染色体按照更高的概率保存到下一代种群中(下一代种群中更多的染色体是上一代适应度函数值表现好的染色体),具体方法可参考【资料1】
3.2、交叉
交叉操作是为了产生新的染色体,将不同染色体的部分基因进行交换,具体方法有单点交叉和多点交叉。
3.3、变异
变异操作同样是为了产生新的染色体,通过改变部分染色体的部分基因。
选择、交叉、变异这三种操作,既保证了适应度函数值表现好的染色体尽可能的保留到下一代种群,又能保证下一代种群的变化,可以产生新的染色体。
二、遗传算法多参数寻优思路
多参数寻优和单一参数寻优的基本思路是相同的,不过在种群初始化时,每次需要初始化两个染色体,并且在选择、交叉、变异操作时,两个染色体是分别进行的。
步骤一:种群、染色体、基因初始化。
步骤二:计算种群中每一组染色体对应的适应度函数值。
步骤三:对种群中的染色体进行选择、交叉、变异操作,产生新的种群。
步骤四:判断是否满足终止条件?如果不满足,跳转到步骤二;如果满足,输出最优参数。
步骤五:结束。
三、遗传算法多参数寻优的python实现
完整的python代码及数据样本地址:https://github.com/shiluqiang/GA_python-for-multi-parameters-optimization
## 2. GA优化算法
class GA(object):
###2.1 初始化
def __init__(self,population_size,chromosome_num,chromosome_length,max_value,iter_num,pc,pm):
'''初始化参数
input:population_size(int):种群数
chromosome_num(int):染色体数,对应需要寻优的参数个数
chromosome_length(int):染色体的基因长度
max_value(float):作用于二进制基因转化为染色体十进制数值
iter_num(int):迭代次数
pc(float):交叉概率阈值(0<pc<1)
pm(float):变异概率阈值(0<pm<1)
'''
self.population_size = population_size
self.choromosome_length = chromosome_length
self.chromosome_num = chromosome_num
self.iter_num = iter_num
self.max_value = max_value
self.pc = pc ##一般取值0.4~0.99
self.pm = pm ##一般取值0.0001~0.1
def species_origin(self):
'''初始化种群、染色体、基因
input:self(object):定义的类参数
output:population(list):种群
'''
population = []
## 分别初始化两个染色体
for i in range(self.chromosome_num):
tmp1 = [] ##暂存器1,用于暂存一个染色体的全部可能二进制基因取值
for j in range(self.population_size):
tmp2 = [] ##暂存器2,用于暂存一个染色体的基因的每一位二进制取值
for l in range(self.choromosome_length):
tmp2.append(random.randint(0,1))
tmp1.append(tmp2)
population.append(tmp1)
return population
###2.2 计算适应度函数值
def translation(self,population):
'''将染色体的二进制基因转换为十进制取值
input:self(object):定义的类参数
population(list):种群
output:population_decimalism(list):种群每个染色体取值的十进制数
'''
population_decimalism = []
for i in range(len(population)):
tmp = [] ##暂存器,用于暂存一个染色体的全部可能十进制取值
for j in range(len(population[0])):
total = 0.0
for l in range(len(population[0][0])):
total += population[i][j][l] * (math.pow(2,l))
tmp.append(total)
population_decimalism.append(tmp)
return population_decimalism
def fitness(self,population):
'''计算每一组染色体对应的适应度函数值
input:self(object):定义的类参数
population(list):种群
output:fitness_value(list):每一组染色体对应的适应度函数值
'''
fitness = []
population_decimalism = self.translation(population)
for i in range(len(population[0])):
tmp = [] ##暂存器,用于暂存每组染色体十进制数值
for j in range(len(population)):
value = population_decimalism[j][i] * self.max_value / (math.pow(2,self.choromosome_length) - 10)
tmp.append(value)
## rbf_SVM 的3-flod交叉验证平均值为适应度函数值
## 防止参数值为0
if tmp[0] == 0.0:
tmp[0] = 0.5
if tmp[1] == 0.0:
tmp[1] = 0.5
rbf_svm = svm.SVC(kernel = 'rbf', C = abs(tmp[0]), gamma = abs(tmp[1]))
cv_scores = cross_validation.cross_val_score(rbf_svm,trainX,trainY,cv =3,scoring = 'accuracy')
fitness.append(cv_scores.mean())
##将适应度函数值中为负数的数值排除
fitness_value = []
num = len(fitness)
for l in range(num):
if (fitness[l] > 0):
tmp1 = fitness[l]
else:
tmp1 = 0.0
fitness_value.append(tmp1)
return fitness_value
###2.3 选择操作
def sum_value(self,fitness_value):
'''适应度求和
input:self(object):定义的类参数
fitness_value(list):每组染色体对应的适应度函数值
output:total(float):适应度函数值之和
'''
total = 0.0
for i in range(len(fitness_value)):
total += fitness_value[i]
return total
def cumsum(self,fitness1):
'''计算适应度函数值累加列表
input:self(object):定义的类参数
fitness1(list):适应度函数值列表
output:适应度函数值累加列表
'''
##计算适应度函数值累加列表
for i in range(len(fitness1)-1,-1,-1): # range(start,stop,[step]) # 倒计数
total = 0.0
j=0
while(j<=i):
total += fitness1[j]
j += 1
fitness1[i] = total
def selection(self,population,fitness_value):
'''选择操作
input:self(object):定义的类参数
population(list):当前种群
fitness_value(list):每一组染色体对应的适应度函数值
'''
new_fitness = [] ## 用于存储适应度函归一化数值
total_fitness = self.sum_value(fitness_value) ## 适应度函数值之和
for i in range(len(fitness_value)):
new_fitness.append(fitness_value[i] / total_fitness)
self.cumsum(new_fitness)
ms = [] ##用于存档随机数
pop_len=len(population[0]) ##种群数
for i in range(pop_len):
ms.append(random.randint(0,1))
ms.sort() ## 随机数从小到大排列
##存储每个染色体的取值指针
fitin = 0
newin = 0
new_population = population
## 轮盘赌方式选择染色体
while newin < pop_len & fitin < pop_len:
if(ms[newin] < new_fitness[fitin]):
for j in range(len(population)):
new_population[j][newin]=population[j][fitin]
newin += 1
else:
fitin += 1
population = new_population
### 2.4 交叉操作
def crossover(self,population):
'''交叉操作
input:self(object):定义的类参数
population(list):当前种群
'''
pop_len = len(population[0])
for i in range(len(population)):
for j in range(pop_len - 1):
if (random.random() < self.pc):
cpoint = random.randint(0,len(population[i][j])) ## 随机选择基因中的交叉点
###实现相邻的染色体基因取值的交叉
tmp1 = []
tmp2 = []
#将tmp1作为暂存器,暂时存放第i个染色体第j个取值中的前0到cpoint个基因,
#然后再把第i个染色体第j+1个取值中的后面的基因,补充到tem1后面
tmp1.extend(population[i][j][0:cpoint])
tmp1.extend(population[i][j+1][cpoint:len(population[i][j])])
#将tmp2作为暂存器,暂时存放第i个染色体第j+1个取值中的前0到cpoint个基因,
#然后再把第i个染色体第j个取值中的后面的基因,补充到tem2后面
tmp2.extend(population[i][j+1][0:cpoint])
tmp2.extend(population[i][j][cpoint:len(population[i][j])])
#将交叉后的染色体取值放入新的种群中
population[i][j] = tmp1
population[i][j+1] = tmp2
### 2.5 变异操作
def mutation(self,population):
'''变异操作
input:self(object):定义的类参数
population(list):当前种群
'''
pop_len = len(population[0]) #种群数
Gene_len = len(population[0][0]) #基因长度
for i in range(len(population)):
for j in range(pop_len):
if (random.random() < self.pm):
mpoint = random.randint(0,Gene_len - 1) ##基因变异位点
##将第mpoint个基因点随机变异,变为0或者1
if (population[i][j][mpoint] == 1):
population[i][j][mpoint] = 0
else:
population[i][j][mpoint] = 1
### 2.6 找出当前种群中最好的适应度和对应的参数值
def best(self,population_decimalism,fitness_value):
'''找出最好的适应度和对应的参数值
input:self(object):定义的类参数
population(list):当前种群
fitness_value:当前适应度函数值列表
output:[bestparameters,bestfitness]:最优参数和最优适应度函数值
'''
pop_len = len(population_decimalism[0])
bestparameters = [] ##用于存储当前种群最优适应度函数值对应的参数
bestfitness = 0.0 ##用于存储当前种群最优适应度函数值
for i in range(0,pop_len):
tmp = []
if (fitness_value[i] > bestfitness):
bestfitness = fitness_value[i]
for j in range(len(population_decimalism)):
tmp.append(abs(population_decimalism[j][i] * self.max_value / (math.pow(2,self.choromosome_length) - 10)))
bestparameters = tmp
return bestparameters,bestfitness
### 2.7 画出适应度函数值变化图
def plot(self,results):
'''画图
'''
X = []
Y = []
for i in range(self.iter_num):
X.append(i + 1)
Y.append(results[i])
plt.plot(X,Y)
plt.xlabel('Number of iteration',size = 15)
plt.ylabel('Value of CV',size = 15)
plt.title('GA_RBF_SVM parameter optimization')
plt.show()
### 2.8 主函数
def main(self):
results = []
parameters = []
best_fitness = 0.0
best_parameters = []
## 初始化种群
population = self.species_origin()
## 迭代参数寻优
for i in range(self.iter_num):
##计算适应函数数值列表
fitness_value = self.fitness(population)
## 计算当前种群每个染色体的10进制取值
population_decimalism = self.translation(population)
## 寻找当前种群最好的参数值和最优适应度函数值
current_parameters, current_fitness = self.best(population_decimalism,fitness_value)
## 与之前的最优适应度函数值比较,如果更优秀则替换最优适应度函数值和对应的参数
if current_fitness > best_fitness:
best_fitness = current_fitness
best_parameters = current_parameters
print('iteration is :',i,';Best parameters:',best_parameters,';Best fitness',best_fitness)
results.append(best_fitness)
parameters.append(best_parameters)
## 种群更新
## 选择
self.selection(population,fitness_value)
## 交叉
self.crossover(population)
## 变异
self.mutation(population)
results.sort()
self.plot(results)
print('Final parameters are :',parameters[-1])
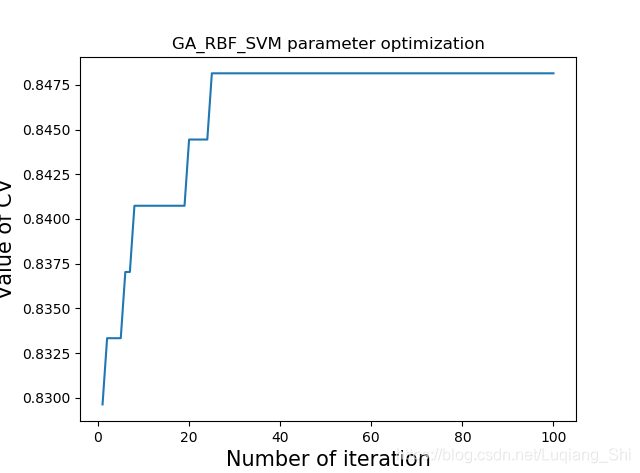
最优的适应度函数值随迭代次数的变化如下图所示。由图可知,在第24次迭代时,参数达到最优,最优参数为[7.79372018547235, 0.010538201696412052]。
参考资料
1、https://blog.csdn.net/quinn1994/article/details/80501542
2、https://blog.csdn.net/Luqiang_Shi/article/details/84570133
3、https://blog.csdn.net/Luqiang_Shi/article/details/84579052