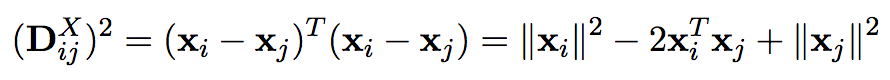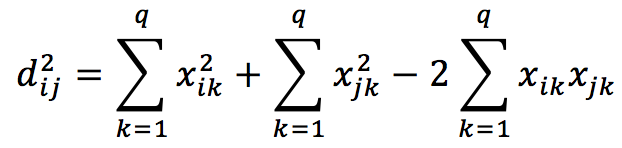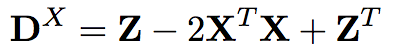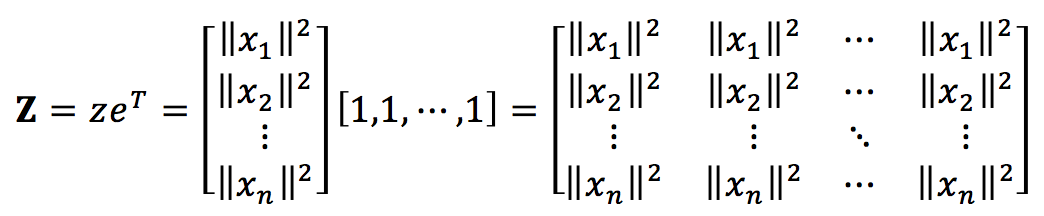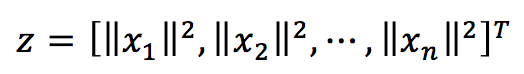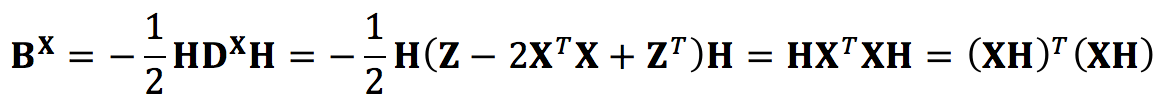流形学习(Manifold Learning)是机器学习中一大类算法的统称,而MDS就是其中非常经典的一种方法。多维标度法(Multidimensional Scaling)是一种在低维空间展示“距离”数据结构的多元数据分析技术,简称MDS。
多维标度法解决的问题是:当n个对象(object)中各对对象之间的相似性(或距离)给定时,确定这些对象在低维空间中的表示,并使其尽可能与原先的相似性(或距离)“大体匹配”,使得由降维所引起的任何变形达到最小。多维空间中排列的每一个点代表一个对象,因此点间的距离与对象间的相似性高度相关。也就是说,两个相似的对象由多维空间中两个距离相近的点表示,而两个不相似的对象则由多维空间两个距离较远的点表示。多维空间通常为二维或三维的欧氏空间,但也可以是非欧氏三维以上空间。
多维标度法内容丰富、方法较多。按相似性(距离)数据测量尺度的不同MDS可分为:度量MDS和非度量MDS。当利用原始相似性(距离)的实际数值为间隔尺度和比率尺度时称为度量MDS(metric MDS),本文将以最常用的Classic MDS为例来演示MDS的技术与应用。
首先我们在R中把csv格式存储的数据文件读入,如下所示:
> data.csv = read.csv("/Users/fzuo/Desktop/data.csv", header = T, row.names = 1)
> data.csv
ATL ORD DEN HOU LAX MIA JFK SFO SEA IAD
ATL 0 587 1212 701 1936 604 748 2139 2182 543
ORD 587 0 920 940 1745 1188 713 1858 1737 597
DEN 1212 920 0 879 831 1726 1631 949 1021 1494
HOU 701 940 879 0 1374 968 1420 1645 1891 1220
LAX 1936 1745 831 1374 0 2339 2451 347 959 2300
MIA 604 1188 1726 968 2339 0 1092 2594 2734 923
JFK 748 713 1631 1420 2451 1092 0 2571 2408 205
SFO 2139 1858 949 1645 347 2594 2571 0 678 2442
SEA 2182 1737 1021 1891 959 2734 2408 678 0 2329
IAD 543 597 1494 1220 2300 923 205 2442 2329 0在解释具体原理之前,我们先来调用R中的内置函数来实现上述数据的MDS,并展示一下效果,此处需要用到的函数是cmdscale()。
> citys<-cmdscale(data.csv, k=2)> cities.names = rownames(data.csv)
> plot(citys[,1],citys[,2],type='n')
> text(citys[,1],citys[,2],cities.names,cex=.7)与实际的地图对照,东西方向反了,应该是左东右西,所以可以把上面的绘图代码稍加修改,则有
> plot(-citys[,1],citys[,2],type='n', ylim=c(-600,600))
> text(-citys[,1],citys[,2],cities.names,cex=.7)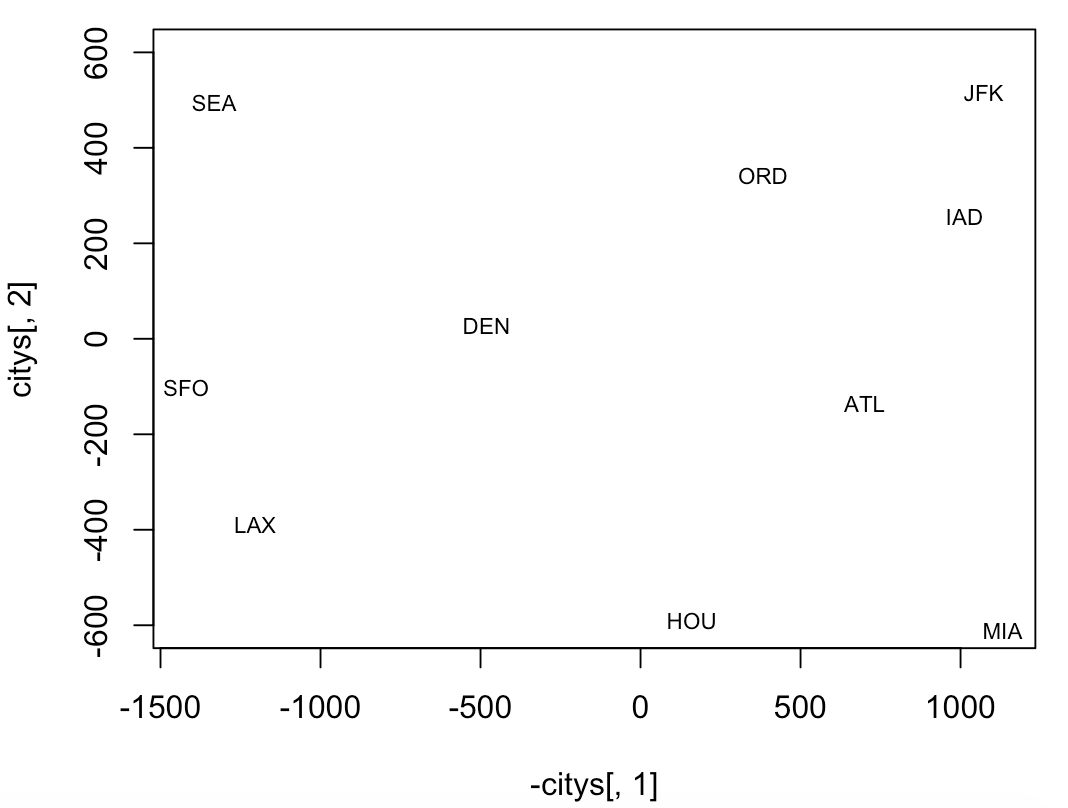
还可以把上图同实际的美国地图做个对照,易见各个城市在图中的位置与实际情况匹配得相当好。
如此神奇的MDS,它背后的原理到底是什么呢,或者它到底是如何实现的呢?下面我们就来抽丝剥茧。
假设X={x1, x2, ..., xn}是一个n×q的矩阵,n为样本数,q是原始的维度,其中每个xi是矩阵X的一列,xi∈Rq。我们并不知道xi在空间中的具体位置,也就是说对于每个xi,其坐标(xi1, xi2, ... , xiq) 都是未知的。我们所知道的仅仅是the pair-wise Euclidean distances for X,我们用一个矩阵DX来表示。因此,对于DX中的每一个元素,可以写成
或者可以写成
对于矩阵DX,则有
其中,
这里的z为
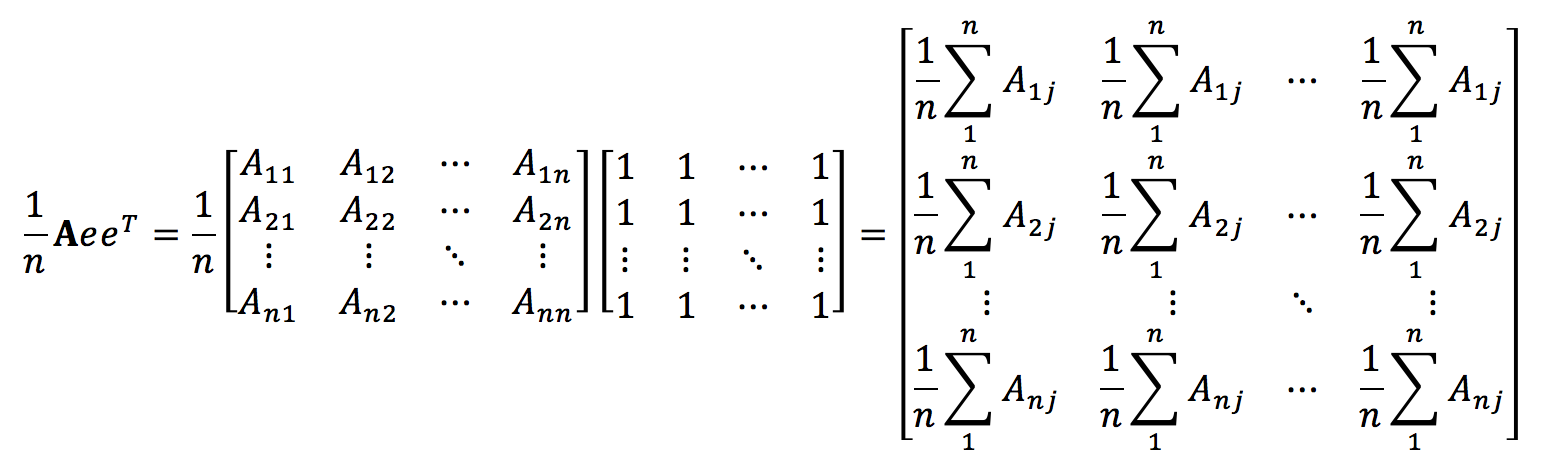
现在让我们来做平移,从而使得矩阵DX中的点具有zero mean,注意平移操作并不会改变X中各个点的相对关系。为了便于理解,我们先来考察一下AeeT/n和eeTA/n的意义,其中A是一个n×n的方阵。
不难发现AeeT/n中第i行的每个元素都是A中第i行的均值,类似的,我们还可以知道,eeTA/n中第i列的每个元素都是A中第i列的均值。因此,我可以定义centering matrix H如下
所以DXH的作用就是从DX中的每个元素里减去列均值,HDXH的作用就是在此基础上再从DX每个元素里又减去了行均值,因此centering matrix的作用就是把元素分布的中心平移到坐标原点,从而实现zero mean的效果。更重要的是,Let D be a distance matrix, one can transform it to an inner product matrix (Kernel Matrix) by K=-HDH/2, 即
上一步之所以成立,因为
因为BX是一个内积矩阵(Gram Matrix/Kernel Matrix),所以它是对称的,这样一来,它就可以被对角化,即BX=U∑UT。
而我们的最终目的是find a concrete set of n points (Y) in k dimensions so that the pairwise Euclidean distances between all the pairs in the concrete set Y is a close approximation to the pair-wise distances given to us in the matrix DX i.e. we want to find DY such that
Note that after applying the ”double centering” operation to both X and Y,上式服从
最终这个问题的解就是Y=U∑1/2。
下面我们首先来演示在MATLAB中计算上述MDS问题的过程,为了展示其原理,下面的代码并不会直接使用MATLAB中内置的用于求解MDS的现成函数。
>> D = [[0,587,1212,701,1936,604,748,2139,2182,543],
[587,0,920,940,1745,1188,713,1858,1737,597],
[1212,920,0,879,831,1726,1631,949,1021,1494],
[701,940,879,0,1374,968,1420,1645,1891,1220],
[1936,1745,831,1374,0,2339,2451,347,959,2300],
[604,1188,1726,968,2339,0,1092,2594,2734,923],
[748,713,1631,1420,2451,1092,0,2571,2408,205],
[2139,1858,949,1645,347,2594,2571,0,678,2442],
[2182,1737,1021,1891,959,2734,2408,678,0,2329],
[543,597,1494,1220,2300,923,205,2442,2329,0]];
>> DSquare = D.^2;
>> H = eye(10)-ones(10)/10;
>> K = -0.5*H*DSquare*H;
>> [eigVec, eigVal] = eig(K);
>> Y = eigVec(:,1:2)*sqrt(eigVal(1:2,1:2))
Y =
1.0e+03 *
0.7188 -0.1430
0.3821 0.3408
-0.4816 0.0253
0.1615 -0.5728
-1.2037 -0.3901
1.1335 -0.5819
1.0722 0.5190
-1.4206 -0.1126
-1.3417 0.5797
0.9796 0.3355最后,我们在给出R中实现的示例代码,同样这里我们不会调用R中内置的用于求解MDS的现成函数。
> data.csv = read.csv("/Users/fzuo/Desktop/data.csv", header = T, row.names = 1)
> D <- as.matrix(data.csv)
> DSqure = D^2
> H = diag(10) - matrix(rep(1,100), nrow = 10)/10
> K = -0.5*H%*%DSqure%*%H
> result = eigen(K, symmetric = FALSE)
> vals = c(result$values[1],result$values[2])
> result$vectors[,1:2] %*% diag(sqrt(vals))
[,1] [,2]
[1,] 718.7594 -142.99427
[2,] 382.0558 340.83962
[3,] -481.6023 25.28504
[4,] 161.4663 -572.76991
[5,] -1203.7380 -390.10029
[6,] 1133.5271 -581.90731
[7,] 1072.2357 519.02423
[8,] -1420.6033 -112.58920
[9,] -1341.7225 579.73928
[10,] 979.6220 335.47281参考文献
【1】http://www.cs.umd.edu/~djacobs/CMSC828/MDSexplain.pdf