这是我自己写的一篇有关遗传算法的文章。
摘 要 很多计算机科学算法往往都受自然界和生物的启发,遗传算法就是受到了生物的自然选择过程而产生的一个启发式算法,启发式算法会尝试通过一些假设来更快地解决问题,因此启发式算法并不是最优的。即便如此,在很多领域都有遗传算法的应用。因此,本文就使用Go语言来实现简单的遗传算法。
关键词 遗传算法 Go语言
Abstract Selection by biological process and produce a Heuristic Algorithm, Heuristic Algorithm will try to through some assumptions to faster to solve the problem, so the Heuristic Algorithm is not optimal. Even so, the application of Genetic Algorithm had been appeared in many fields. Therefore, this article will use the Golang to implement a simple Genetic Algorithm.
Keywords: Genetic Algorithm Golang
1 遗传算法简述
遗传算法(GA)是由美国Michigan大学的Holland教授于1975年首先提出来的。它源于达尔文的进化论、孟德尔的群体遗传学说和威茨曼的物种选择学说;其基本思想是模拟自然界遗传机制和生物进化论而形成的一种过程搜索最优解的算法。
近年来,遗传算法已被成功地应用于工业、经济管理、交通运输、工业设计等不同领域,解决了许多实际问题。例如,可靠性优化、作业车间调度、机器调度、流水车间调度、设备布局设计、图像处理以及数据挖掘等。本文将用一个小程序来实现遗传算法,并对遗传算法做一些必要的分析。
2 遗传算法的基本原理
自然选择作为进化的关键机制。这是一个自然地过程,会导致群体随着时间的推移适应环境。这些群体的形状有所不同,具有更适合的个体生物在环境中会有更高的存货机会。从这些幸存的生物中交叉产生的下一代将继承它们的性状,因此,拥有这些性状的群体会越来越多,而没有这些性状的群体就会越来越少,这就是自然选择。
遗传算法与自然选择有什么关系呢?下面就列举遗传算法中用到的成员和方法。
DNA:定义一个具有DNA的生物个体
种群:一定数量的拥有不同的DNA的生物群体
适应度:每个个体对环境的适应度
选择:选择出环境适应能力强的群体,由此产生下一代群体
繁殖(交叉):生物个体会继承自其父代中的部分DNA
突变:对于每一个生物体都有可能突变,但是概率较小
3 遗传算法简单实现想法的来由
有一个理论叫无限猴子理论,大概是说无限多的猴子坐在打字机打字,如果时间足够长,将写成一部莎士比亚全集。这个理论就是我简单实现遗传算法的一个模板,因为,遗传算法是启发式的,通过自然选择,我们可以在一堆数据中选出最接近莎士比亚全集的文字,所以遗传算法应用与此可是再合适不过了。
4 代码实现
4.1 生物个体的表现形式
我们可以用一个包含有DNA和适应度的结构体来表现一个生物个体:
type Organism struct {
DNA []byte
Fitness Float64
}
4.2 产生一个生物个体
要产生一个群体,首先要从产生一个生物个体开始:
// 生成一个个体
func createOrganism(Target []byte) (organism Organism) {
ba := make([]byte, len(Target))
for i := 0; i < len(Target); i++ {
// 生成一个随机字符
ba[i] = byte(rand.Intn(95) + 32)
}
organism = Organism{
DNA: ba,
Fitness: 0,
}
// 计算适应度
organism.calcFitness(Target)
return
}
在这里Target就是我们想要的最终目标,一句莎士比亚的名言:No matter how long the night, the day will come.首先随机产生一个与目标语句一样长度的随机字符串,也就是生物个体的DNA,calcFitness是一个计算该生物个体的适应度的函数。
4.3 产生一个群体
既然已经可以产生一个生物个体了,那么产生一个群体就不在话下了:
// 初始化一个族群
func createPopulation(Target []byte) (population []Organism) {
population = make([]Organism, PopSize)
for i := 0; i < PopSize; i++ {
population[i] = createOrganism(Target)
}
return
}
根据参数PopSize的大小产生这么多个的生物个体群体,这个参数是影响整个算法的一个重要参数。
4.4 计算生物个体的适应度
在产生一个生物体的函数中我们用到了这个函数,在其他地方也会被调用:
// 计算适应度
func (d *Organism) calcFitness(Target []byte) {
score := 0
for i := 0; i < len(d.DNA); i++ {
if d.DNA[i] == Target[i] {
score++
}
}
d.Fitness = float64(score) / float64(len(d.DNA))
return
}
原理很简单,就是用该生物个体的DNA和目标的DNA进行比较,相同的越多适应度就越高,这是一个0到1的数。
4.5 选择适应能力强的生物个体
这个函数将产生新一代的群体,但数目远大于原本的群体,它根据各个生物个体的适应度来复制产生多个该生物个体的克隆,这样间接来实现在接下来的繁殖中适应度高的个体被选中的概率就大
// 根据前一代适应度的生物来克隆多个生物个体
func createPool(population []Organism, Target []byte, maxFitness float64) (pool []Organism) {
pool = make([]Organism, 0)
// 产生一个族群池,为以后模拟自然选择的基础
for i := 0; i < len(population); i++ {
population[i].calcFitness(Target)
num := int((population[i].Fitness / maxFitness) * 100)
for n := 0; n < num; n++ {
pool = append(pool, population[i])
}
}
return
}
4.6 自然选择
这个函数是整个遗传算法的核心,正是因为这个函数体现了自然选择
// 产生下一代族群
func naturalSelection(pool []Organism, population []Organism, Target []byte) []Organism {
next := make([]Organism, len(population))
for i := 0; i < len(population); i++ {
// 在前面的基础上中随机找两个个体,这样适应度高的生物个体被选中的概率就大
r1, r2 := rand.Intn(len(pool)), rand.Intn(len(pool))
a := pool[r1]
b := pool[r2]
child := crossover(a, b)
child.mutate()
child.calcFitness(Target)
next[i] = child
}
return next
}
函数中写的crossover(交叉),mutate(变异),将在下面给出,next就是下一代群体。
4.7 交叉的实现
func crossover(d1 Organism, d2 Organism) Organism {
child := Organism{
DNA: make([]byte, len(d1.DNA)),
Fitness: 0,
}
// 子代得到父母的DNA各一部分
mid := rand.Intn(len(d1.DNA))
for i := 0; i < len(d1.DNA); i++ {
if i > mid {
child.DNA[i] = d1.DNA[i]
} else {
child.DNA[i] = d2.DNA[i]
}
}
return child
}
交叉的实现可以千变万化,我这里采用的方法是子代得到父母DNA的各一部分DNA,这样产生的子代也将大概率的比其父母适应能力更强。
4.8 变异的实现
func (d *Organism) mutate() {
for i := 0; i < len(d.DNA); i++ {
// 随机改变DNA中的某一个
if rand.Float64() < MutationRate {
d.DNA[i] = byte(rand.Intn(95) + 32)
}
}
}
根据MutationRate(变异的概率),这也是一个很重要的参数,对结果有很大的影响,在后面的结果中就能看到。
4.9 找出群体中最优的生物个体
func getBest(population []Organism) Organism {
best := 0.0
index := 0
for i := 0; i < len(population); i++ {
if population[i].Fitness > best {
index = i
best = population[i].Fitness
}
}
return population[index]
}
最优即该生物个体的适应度在整个群体中是最大的,这个函数将在主函数的循环中调用。
4.10 主函数
func main() {
fmt.Printf("MutationRate = %f\n", MutationRate);
fmt.Printf("PopSize = %d\n", PopSize);
fmt.Printf("Target = %s\n", Target);
rand.Seed(time.Now().UTC().UnixNano())
// 根据目标字符串生成一个族群
population := createPopulation(Target)
found := false
generation := 0
// 计时
start := time.Now()
// 结束条件:一直迭代直到找到完全匹配的字符串
for !found {
generation++
bestOrganism := getBest(population)
fmt.Printf("\rgeneration: %d | %s | fitness: %2f", generation, string(bestOrganism.DNA), bestOrganism.Fitness)
if bytes.Compare(bestOrganism.DNA, Target) == 0 {
found = true
} else {
// 没完全匹配就一直自然选择
maxFitness := bestOrganism.Fitness
pool := createPool(population, Target, maxFitness)
population = naturalSelection(pool, population, Target)
}
}
elapsed := time.Since(start)
fmt.Printf("\nTime taken: %s\n", elapsed)
}
不同的遗传算法的结束条件有所不同,我采用的是找到完全匹配的字符串即结束算法,也可以用迭代次数来作为结束条件。我们根据运行的时间来判断算法的好坏。
4.11 可调的、重要的参数
// 变异的概率
var MutationRate = 0.005
// 种群数量
var PopSize = 500
// 目标字符串
var Target = []byte("No matter how long the night, the day will come")
这三个参数都是可以随机设定的,不同的值可能会对结果有很大的影响。
5 运行结果
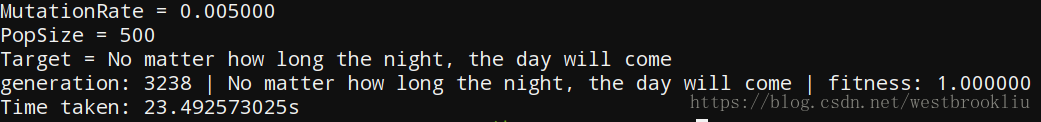
先展示上面源代码参数的运行结果:

图一 运行结果一
图二 运行结果二
上面的两个运行结果从参数都一样,大概花了二十多、三十秒就迭代出了目标字符串,以这些参数运行多次,时间在三十秒上下波动,这看似体现了遗传算法较为稳定的特性。
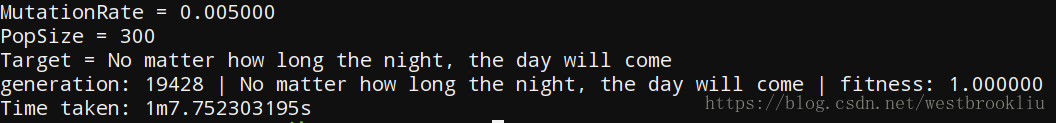
接下来修改PopSize这个参数,看看会有什么影响:
图三 运行结果三
PopSize改为300,看起来还能接受,但如果改为200或100这个程序将可能陷入局部最优,也就是一直迭代下去,时间可能是无限长,这也是遗传算法的一个弊病,所以有些遗传算法的实现的结束条件是迭代次数,防止程序无限期的运行下去。
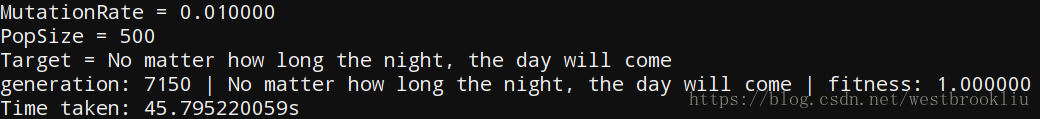
接着来修改MutationRate这个参数,看看会有什么影响:
图四 运行结果四
不难看出变异的概率虽然更大了但是运行时间却更长了,这是由于变异的概率反而增大了遗传算法的随机性,这个变异的方向可能是好的方向也可能是不好的方向,如果变异概率更大的话会更早陷入局部最优:
图五 运行结果五
可以发现MutationRate为0.05,由于程序的fitness一直在0.4上下波动,因此程序陷入了局部最优。
6 结论
遗传算法在这种类似问题的求解与应用中展现了其独特的魅力,但也暴露了它的局限性和缺陷,很容易陷入局部最优是其比较严重的问题,其次它的随机性也是一个不容忽视的问题。因此要利用好遗传算法,参数的选择和结束条件的设置极为重要,这就要根据题目的不同设置不同的方案。所以关于遗传算法的研究重点在一下几个方面:
1) 算法的改进:针对不同领域的问题来对遗传算法进行改进与完善。
2) 算法的数学理论基础:包括算法的收敛性、收敛速度估计、早熟机理的探索与预防,交叉算子的几何意义与统计解释。