目录
从一篇文章入手(基于多模态深度学习的泛癌症整合组织-基因组分析)
Local explanations. V.s. Global Explanations
用于千兆像素整张幻灯片图像生存预测的多模态共同注意转换器 (MCAT)
HFBSurv:用于癌症生存预测的分解双线性模型的分层多模式融合
Hierarchical Graph Pathomic Network for Progression Free Survival Prediction
GPDBN:整合基因组数据和病理图像的深度双线性网络用于乳腺癌预后预测
从一篇文章入手(基于多模态深度学习的泛癌症整合组织-基因组分析)
- 局部可解释:注意力Heatmap+ROI(ROI Heatmap) ;分子解释:高-低特征相关
- 全局可解释:三种模型生存曲线下面积+Integrate gradient attribution
- 评价模型预测能力的一致性指数(c-Index)
1.工具:
Python画attention map与原图叠加的热力图 - 知乎
2.数据信息:来自TCGA数据库的14种肿瘤类型,包含5720个病人,6592张十亿像素的全片扫描图像,同时还有匹配的分子学数据,包括拷贝数变异(CNV)、基因突变信息(MUT)和RNA-seq数据。
模型:

- (1)AMIL模型(仅使用组织学数据)为HE 全片扫描图像(WSI)提供服务,AI记录学习分析每个病人的切片信息, 然后反馈病人病理切片的信息,并以一个高分辨率关注度的热图呈现出来,不同区域的图像热度代表不同的预后风险。
- (2)SNN算法(仅使用组织学数据)为每个病人的分子信息做服务,所含内容包括CNV, mutation,RNA-seq,他们使用SHAP形式的归因图来可视化不同分子特征的比重和方向。
- (3)将两种算法进行融合,得到多模态融合模型MMF(multimodel fusion)。MMF通过整合WSI信息和分子谱数据来对病人的风险等级进行划分(high risk or low risk)并进行生存分析
3.信息贡献率:
紧接着作者对全切片图像(WSIs)为癌症预后分析所贡献的比重进行了定量,发现在14种肿瘤类型中,WSIs信息贡献平均占比16.8%,这表明在MMF风险预测时,组学数据依然是主要依据。但是也有部分癌症较高程度依赖WSIs信息,如在UCEC(子宫内膜癌)中WSIs占比高达55.1%,在HNSC(头颈癌),STAD(胃腺癌)和LIHC(肝癌)等其他癌种中也观察到较高的贡献比例。说明当我们想用一个统一的模型预测所有的癌症类型时,模型的反应会有很大不同。同时因为MMF综合了两个维度的数据,所以相比于AMIL能反馈更多的基因组学数据,而相比于SNN又能反馈更多的组织学数据,所以是一个更优的深度学习模型。
评价:肿瘤浸润的淋巴细胞(TIL)可以作为癌症中可靠的预后指标,说明这个模型的关注度聚焦在高免疫区域,它也证明了免疫反应可以作为许多癌症中可靠的预后指标,可为对高低风险患者分级提供依据。
Post hoc Explainability
Local explanations. V.s. Global Explanations
- Feature importance/ Rule-based/ Prototypes/ Counterfactual
- LIME: Learn sample liner model on weighted samples; liner model explain
- SHAP: marginal contribution of each feature towards the prediction
- Anchor: perturb a given instance to generate a local neighborhood(maximum coverage of local neighborhood)
- Saliency Maps:
1/N *input Gradient (x+
),

- Influence Function and Activation Maximization: cook's distance; (UpWeighted) ERM solution -> 缺点:hessian 矩阵+ non-convexity
- 最小化counterfactual距离(cost)+基于counterfactual预测与y' label的距离
2. Collection/ Representation Based/ Distillation
- SP-LIME/ Ancor k explanations
- Global explanation
Evaluation
faithfulness stability fairness
- 删减重要特征;只用重要特征
- input 扰动最小化结果变化
- majority v.s. minority


LIME SHAP unstable inconsistency-> distribution instead importances
用于千兆像素整张幻灯片图像生存预测的多模态共同注意转换器 (MCAT)
1.目的
多模式共同注意力机制的transformer(MCAT)框架,该框架在WSIs和基因特征组成的嵌入空间学习有解释性的,密集的共同注意映射。
受到视觉问答(Visual question answering VQA)的启发:该方法可以确定,在回答问题时,单词嵌入是如何关注图像中的突出对象;当预测病人的生存情况时,MCAT学习病理图像是如何与基因相关的。除了可视化的多模式交互,1) 作者的共同注意力transformer减少了 WSI bags的空间复杂度,2)这也使得transformer 层在多实例学习中能够作为通用的编码器主干。
2.挑战
- 定位在占总图像很小比例的小像素区域中。(一个大海捞针的问题,比如区分正常组织和微转移瘤)
- 贯穿于整个WSI的组成部分,并且依赖于组成部分的交互(这个切片级的标签,可能单看某一部分无法判断其标签,要整体去评价,充分利用他们的上下文信息。)一个精细的视觉识别问题,例如涉及了解基质、肿瘤聚集、免疫细胞和其他视觉概念的复杂环境的问题。
解决:
- 由于WSI具有巨大的上千兆分辨率,许多方法采用两阶段的多实例学习来解决WSI的表示学习
- 随机在WSI中采用图片并提取他们的实例级别的特征表示
- 然后在实例级别组成的包中运用全局聚合策略去获得WSI级别的表示来进行后续的监督任务。虽然在实例级之间不能去建立这种复杂的相互反应,但是,多实例学习(MIL)可以解决病理学中许多大海捞针的问题(needle-in-a-haystack problems),例如正常组织和微转移的分类,它仅依赖于区分二元实例级的视觉感念(也就是不需要上下文信息)
- 生存分析是一个有挑战的有序回归任务,它旨在去预测癌症死亡的相对风险,它适用于后续的精细度视觉识别问题。传统的MIL方法无法区分这些概念,例如对肿瘤细胞和淋巴细胞的共同定位可以得到一个良好的预后结果,而这需要对WSI中实例之间的中长期相互作用进行建模。
- 病理学家对组织学和基因组学的手动评估是患者分类、风险评估和分层到治疗组的金标准(人的标准还是金标准)
- 而基因组特征通常是一些1*1的表格属性。因此需要多模态交互信息
- 虽然可以很自然的将WSI转化为patch的序列(sequence)或是包(bag),但是相比于单词嵌入来说,单词嵌入最大长度最多512dim,而对于一个20倍放大下的wsi,他的包的尺寸包含大约15000个256*256的image patches,最大的序列长度有200,000个patch,由WSI巨大的空间复杂性,使用Transformer 和一些其他的堆叠的自注意力的网络框架,在相应的MIL任务中,是一个计算行不通的。
贡献:
文中使用一个跨模态的注意力机制(cross-modality attention, co-attention),也被称为基因指导的跨模态的注意力机制,以此来作为早期的融合策略,(Mobadersany使用特征拼接来对病理学特征和基因特征进行聚合,然后做生存预测;Chen等使用kronecker product 来对image,graph,和基于基因的特征进行聚合,基于后期融合的,特征只融合到了倒数第二层网络,并且提供了有限的多模态交互解释能力。)他使用基因组特征作为查询从大型置换不变集中识别信息实例,这个生存分析带来了两个好处:1.基因指导的跨模态的注意力机制;2.展示了GCA层是如何将WSI包的有效”序列长度“从M个实例级特征减少到N个基因引导的视觉概念。N是基因嵌入的有效序列长度集合(M>>N),这也使得我们能够使用self-attention 和Transformers进行监督训练开发更复杂的功能聚合策略,作者将基因指导的视觉概念可视化为热图,以此来分析WSI和基因之间特征的相互作用。并评估形态特征如何影响每个基因的模态。
作者也证明了这个GCA层是如何可以减少WSI bags的空间复杂度,这种基于集合的数据结构也称作包,每一个 包是一个无序的实例集合(具有置换不变性,permutation-invariant),这些包的大小可能不同,实例级别的标签不完整。对于单标签分类,给定一个包X = {x 1 ,…,x M },目标是学习一个置换不变函数来预测这个包的标签,而不需要一些详细的实例知识
方法:
用的wsi中的patch(一个病人可能有多个wsi),而不是在ROI中去采样patch,对于patch切割的方式,采用不重叠的切割(大小为256*256),然后用一个Resnet-50+ fc(在ImageNet上预训练)的模型进行特征提取。在跨多个WSI利用整个组织微观环境的过程中,训练和推理期间,平均包的大小包括115231个实例,而最大的包包含230,000个实例。从这一步开始,传统方法运用加法等进行全局聚合形成最后的特征输出,然后后面跟一个拼接或是双线性池化和基因进行融合
在嵌入空间表述基因(formulating genes in an embedding space),基因组特征(如基因突变状态、拷贝数变化和大量RNA序列丰度)通常量化为1×1测量值或属性,仅此一项不包含任何语义信息来描述基因在生物系统中的功能影响。
Instance-Level 256 x 256 Patch Embedings (H_bag before GCA):
torch.Size([15231, 1, 256])
Genomic Embeddings (G_bag before GCA):
torch.Size([6, 1, 256])
Genomic-Guided WSI-Level Embeddings (H_bag after GCA becomes H_coattn):
torch.Size([6, 1, 256])
Genomic Embeddings (G_bag after GCA stays same):
torch.Size([6, 1, 256])
Co-Attention Matrix:
torch.Size([6, 15231])
H_coattn after Transformers:
torch.Size([6, 1, 256])
G_bag after Transformers:
torch.Size([6, 1, 256])
Final WSI-Level Representation (h^L):
torch.Size([256])
Final Genomic Representation (g^L):
torch.Size([256])
Final shared representation (h_final):
torch.Size([256])
基因特征包括六类:1) Tumor Supression(肿瘤抑制)2)瘤形成 3)蛋白激酶类 4)细胞分化 5)转录 6)细胞因子与生长
a.基因指导的跨模态注意力层:
对于单个基因嵌入,GCA层去计算该基因嵌入和所有的patch特征的相似度,这样的相似度分数被用来去计算一个新的patch嵌入。如果Gin是表达潜在的生物通路的基因嵌入(这个通路负责肿瘤形成),那个这个相似度分数将会聚焦定位在含有肿瘤区域的图像上,并高度关注这个图像,然后聚合这些特征作为WSI级别的表示
b.限制:该基因集合具有潜在的重叠功能影响(用人话说就是这些基因集合中可能有的基因表达的功能是重叠的,多个基因可能表达同一个功能),所以未来的工作将侧重于研究WSI与更细粒度、更独特的生物基因集的早期融合,进一步量化表型-基因型的对应关系。
Pathomic Fusion: An Integrated Framework for Fusing Histopathology and Genomic Features for Diagnosis and Prognosis
使用注意力门控和张量融合来整合组织学图像和组学数据的简单且可扩展的方法。可以使用CNN或GCN处理组织病理学图像以提高参数效率或两者的组合。该设置适用于将多组学模式与组织病理学相结合,并可用于改进诊断、预后和治疗反应的确定。
1.background & motivation
2.研究现状(用于肿瘤学多模式数据集成的人工智能)
HFBSurv:用于癌症生存预测的分解双线性模型的分层多模式融合
癌症生存预测可以极大地帮助临床医生规划患者治疗并改善他们的生活质量。最近的证据表明,多模态数据(例如基因组数据和病理图像)的融合对于理解癌症异质性和增强生存预测至关重要。作为一种强大的多模态融合技术,Kronecker 乘积在预测生存方面显示了其优越性。然而,该技术引入了大量参数,可能导致高计算成本和过拟合风险,从而限制了其适用性和性能提升。为了解决上述局限性,我们提出了一种名为 HFBSurv 的新型分层多模态融合方法,该方法采用因式分解双线性模型逐步融合基因组和图像特征。具体来说,HFBSurv 采用多重融合策略将融合问题分解为不同层次,每个层次从低层次到高层逐步集成和传递信息,从而导致更专业的融合过程和表现力多模态表示。在这个分层框架中,模态特定和跨模态注意因子分解双线性模块的设计不仅可以捕获和量化多模态数据中的复杂关系,还可以显着降低计算复杂性。

MLA-GNN “Multi-Level Attention Graph Neural Network Based on Co-expression Gene Modules for Disease Diagnosis and Prognosis”
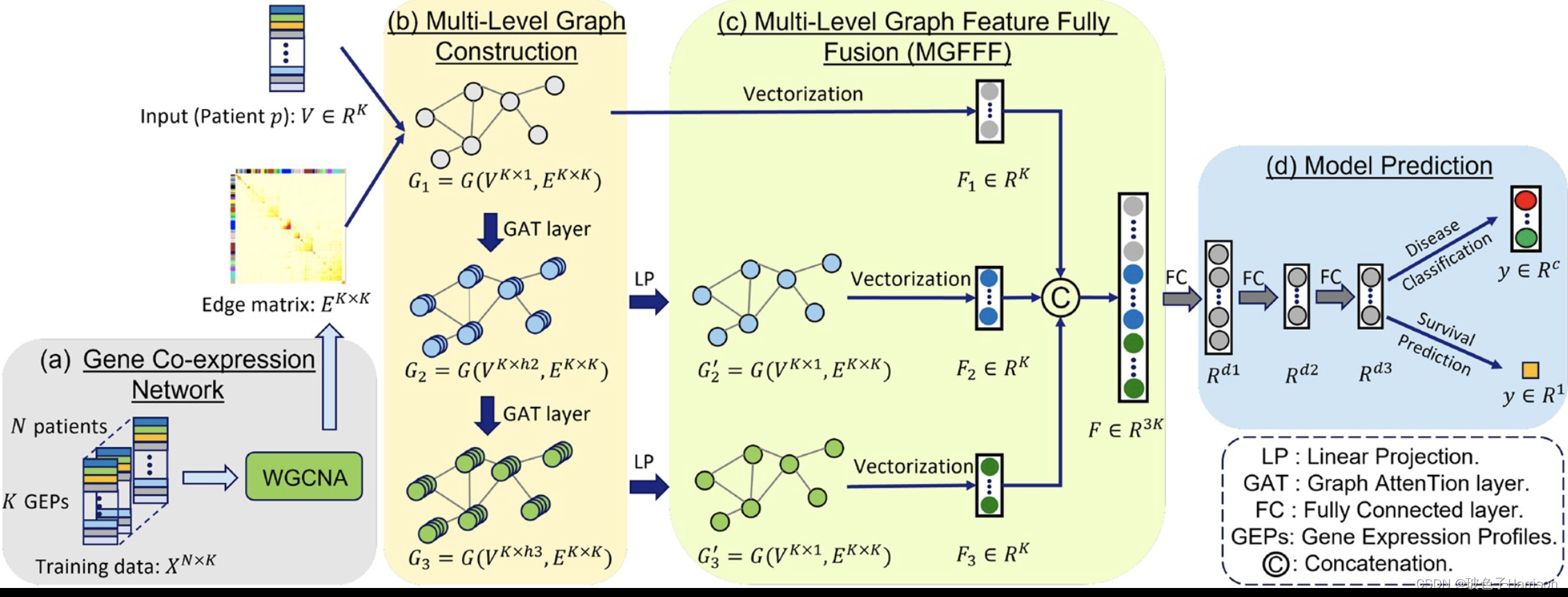
Hierarchical Graph Pathomic Network for Progression Free Survival Prediction

- Clinical Features. We utilized clinical variables including age, PSA value, Gleason grade group (GG), and pathologic T stage to fit a baseline Cox model.
- Linear Cox Model.
- Attention MIL-Based Aggregation.
- Texture-Based Features.
- Self-supervised CNN Features.
- Handcrafted Cell Graph Features.
GPDBN:整合基因组数据和病理图像的深度双线性网络用于乳腺癌预后预测
提出了一种模态间双线性特征编码模块来模拟复杂的模态间关系,以充分利用跨不同模态的特征的内在关系。同时,也有利于预后预测的模态内关系由两个模态内双线性特征编码模块捕获。此外,为了利用模态间和模态内关系之间的互补信息,GPDBN 通过使用多层深度神经网络进一步结合模态间和模态内双线性特征进行最终预后预测。
:批判文章有什么不足 自己有什么想法
@inproceedings{chen2021multimodal,
title={Multimodal Co-Attention Transformer for Survival Prediction in Gigapixel Whole Slide Images},
author={Chen, Richard J and Lu, Ming Y and Weng, Wei-Hung and Chen, Tiffany Y and Williamson, Drew FK and Manz, Trevor and Shady, Maha and Mahmood, Faisal},
booktitle={Proceedings of the IEEE/CVF International Conference on Computer Vision},
pages={4015--4025},
year={2021}
}
@article{chen2021pan,
title={Pan-cancer integrative histology-genomic analysis via multimodal deep learning},
author={Chen, Richard J and Lu, Ming Y and Williamson, Drew FK and Chen, Tiffany Y and Lipkova, Jana and Shaban, Muhammad and Shady, Maha and Williams, Mane and Joo, Bumjin and Noor, Zahra and others},
journal={Cancer Cell},
year={2022}
}


