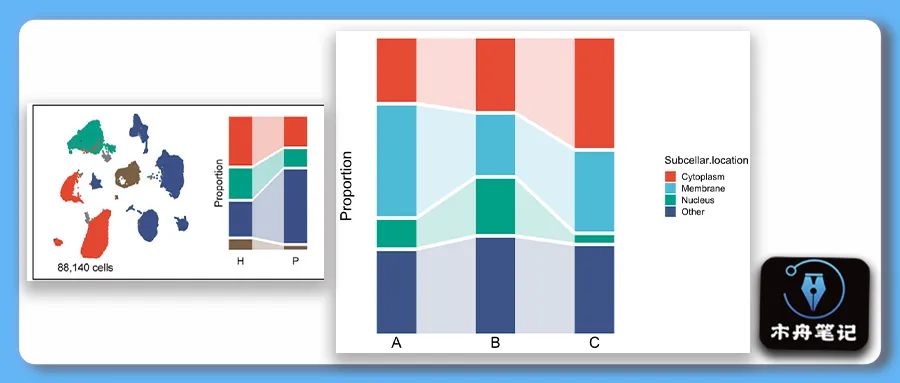
今天我们复现一幅2021年Cell上Graphical abstract的图。
Title:Human oral mucosa cell atlas reveals a stromal-neutrophil axis regulating tissue immunity
DOI:10.1016/j.cell.2020.07.009
读图
复现结果
示例数据和代码领取
木舟笔记2022年度VIP企划
开始绘制
饼图
堆叠条形图
原图复现
往期内容
读图
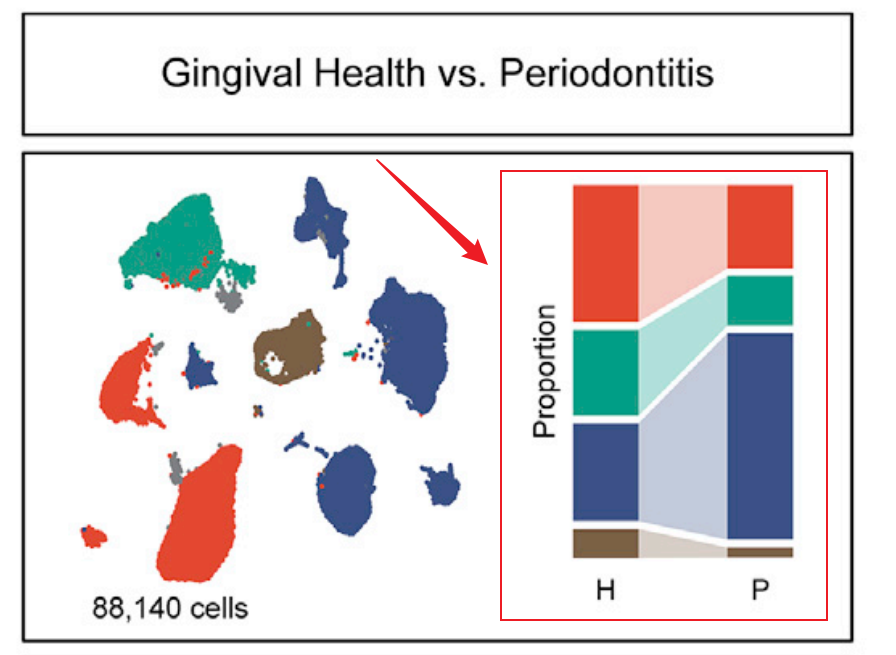
来看一下这个摘要图(红色箭头),作者用堆叠条形图来展示两组样本间比例的变化,中间又增加了连线让相同组分之间对对比更加的直观。一般这种数据就是以饼图或是堆叠条形图来展现,今天我们就用饼图和条形图两种形式都来复现一下。
复现结果
示例数据和代码领取
点赞、在看 本文,分享至朋友圈集赞20个并保留60分钟,截图发至微信mzbj0002领取。
木舟笔记2022年度VIP可免费领取。
木舟笔记2022年度VIP企划
权益:
2022年度木舟笔记所有推文示例数据及代码(在VIP群里实时更新)。

木舟笔记科研交流群。
半价购买
跟着Cell学作图系列合集(免费教程+代码领取)|跟着Cell学作图系列合集。
收费:
99¥/人。可添加微信:mzbj0002 转账,或直接在文末打赏。

开始绘制
饼图
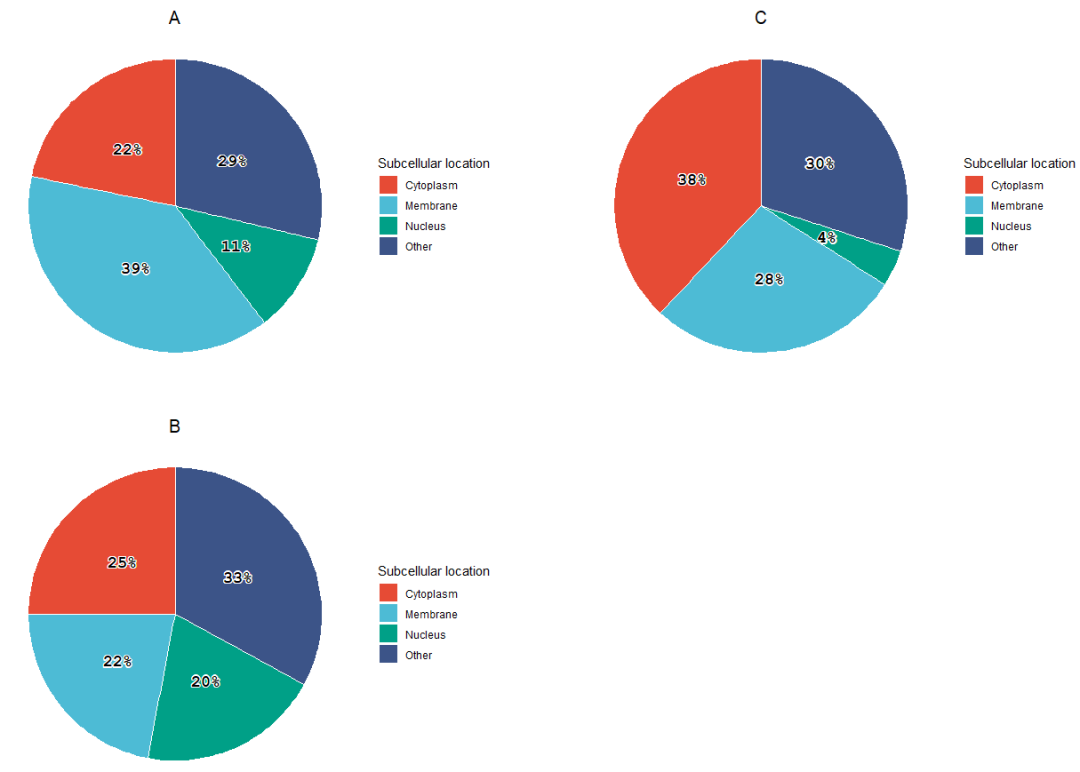
数据是三组样本蛋白质的亚细胞定位数据,一般的可视化方法就会用饼图来展示,饼图十分常见而且制作特别简单,大部分情况用excel或origin等软件都可以快速制作,所以在R中的相关函数很少。这里我们用到了tastypie包,其中的pie_bake函数生成一个ggplot对象可以非常方便的调整你的饼图。
# 加载包及导入数据
library(ggplot2)
library(ggthemes)
library(tidyverse)
library(ggalluvial)
library(ggsci)
library(cowplot)
library(tastypie)
rm(list = ls())
df=read.csv('Sublocation.csv',header = T)
head(df)p1=pie_bake(data = data.frame(subloc=df$Subcellar.location,ratio=df$A),
template = "blue1",#调色盘是必要参数,不影响手动添加颜色
perc = TRUE,#显示百分比
group_name = "a",
title ='A')+
theme_classic()+
scale_fill_npg()+#ggsci中的调色盘
theme(panel.background = element_blank(),
axis.line.x = element_blank(),
axis.line.y = element_blank(),
axis.title.y = element_blank(),
axis.title.x = element_blank(),
axis.text.x = element_blank(),
axis.text.y = element_blank(),
axis.ticks.x = element_blank(),
axis.ticks.y = element_blank(),
legend.title = element_text(),
plot.title = element_text(hjust = 0.5,vjust=-2,size = 15))+labs(fill = 'Subcellular location')
p2=pie_bake(data = data.frame(subloc=df$Subcellar.location,ratio=df$C),
template = "blue1",
perc = TRUE,
group_name = "a",
title ='C')+
theme_classic()+scale_fill_npg()+
theme(panel.background = element_blank(),
axis.line.x = element_blank(),
axis.line.y = element_blank(),
axis.title.y = element_blank(),
axis.title.x = element_blank(),
axis.text.x = element_blank(),
axis.text.y = element_blank(),
axis.ticks.x = element_blank(),
axis.ticks.y = element_blank(),
legend.title = element_text(),
plot.title = element_text(hjust = 0.5,vjust=-2,size = 15))+labs(fill = 'Subcellular location')
p3=pie_bake(data = data.frame(subloc=df$Subcellar.location,ratio=df$B),
template = "blue1",
perc = TRUE,
group_name = "a",
title ='B')+
theme_classic()+scale_fill_npg()+
theme(panel.background = element_blank(),
axis.line.x = element_blank(),
axis.line.y = element_blank(),
axis.title.y = element_blank(),
axis.title.x = element_blank(),
axis.text.x = element_blank(),
axis.text.y = element_blank(),
axis.ticks.x = element_blank(),
axis.ticks.y = element_blank(),
legend.title = element_text(),
plot.title = element_text(hjust = 0.5,vjust=-2,size = 15))+
labs(fill = 'Subcellular location')
plot_grid(p1,p2,p3)在R中制作饼图也是十分的简单,但当多组进行比较的时候多个饼图的对比就没那么直观了,这时候我们就需要做堆叠条形图。
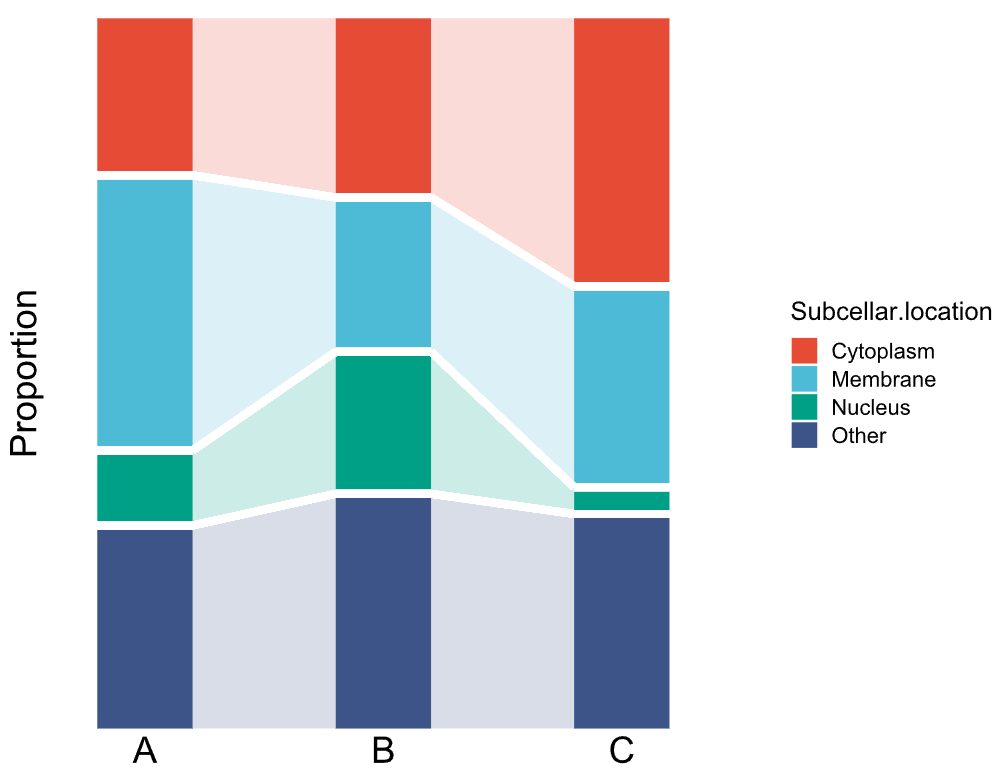
堆叠条形图
首先对数据进行前处理做成适合ggplot作图的数据格式,把宽表转换为长表,创建一列组成比例用于后面分割图形。
mdf=df%>%
pivot_longer(-Subcellar.location,names_to = 'Sample',values_to = 'Value')%>%
group_by(Sample)%>%
mutate(Proportion=Value/sum(Value))geom_bar或是geom_col可以很轻松的做出堆叠条形图。
ggplot(mdf, aes(x = Sample,y=Proportion,fill = Subcellar.location)) +
geom_col(width = 0.4)+
theme_base()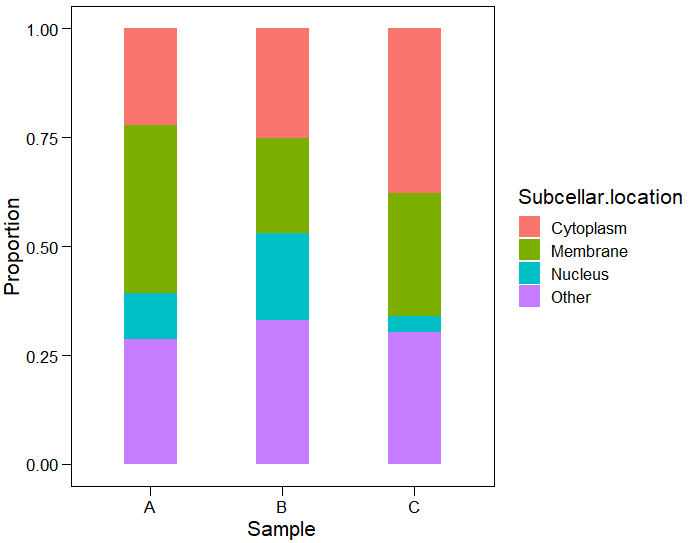
但为了复现文章中原图的格式,我们调用ggalluvial包中的geom_alluvium或是geom_flow函数来制作桑葚图(冲击图),后面我们会专门对桑葚图(冲击图)进行讲解,这里作为实战应用简单讲解。
g=ggplot(mdf, aes(x = Sample,y=Proportion,fill = Subcellar.location,
stratum = Subcellar.location, alluvium = Subcellar.location)) +
geom_col(width = 0.4,color=NA)+
geom_flow(width = 0.4,alpha = 0.2,knot.pos = 0) +#knot.pos可以使连线更直
#geom_alluvium( width = 0.4,alpha = 0.2,knot.pos = 0)+ 与geom_flow效果相似
scale_fill_manual(values = pal_npg()(4))+
theme_map()+
theme(axis.text.x=element_text(size=20,vjust = 5),
legend.position = 'none')
g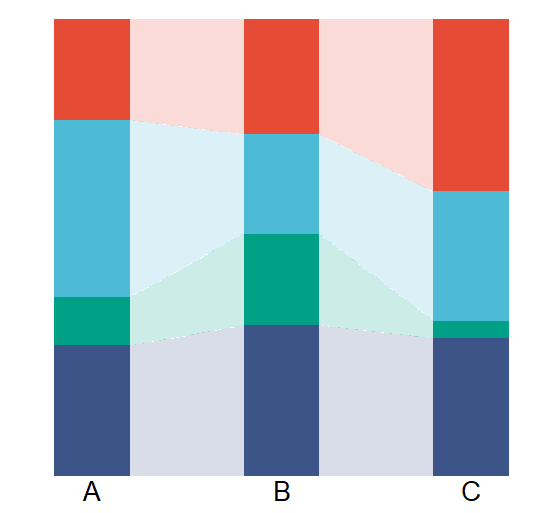
原图复现
大体结构已经做出来了,后面就是添加一些连线,强烈建议在AI中修改,只需要几分钟就可以修改好,在R中有几种实现的方法,但总的来说都比较麻烦这里介绍一种,有朋友有好的实现思路也可以在后台留言。一种实现方法就是计算出各节点的坐标然后用geom_segment添加白色线段。