# ITS
ATCATTA| → → →
← ← ← GACCTCA|AATCA
# LSU
TAACAAGG|TCCCC → → →
← ← ← GAAGAAGCC|笨办法,仅供参考,后续会有更新!!
-
ITS
ITS5(正向)ITS4(反向)序列较短,一般可不用进行序列反向互补和序列比对,若出现套峰则要进行序列反向互补和序列比对。
用snapgene打开ab1文件,用记事本打开seq文件。
如下峰图,其碱基序列可直接使用
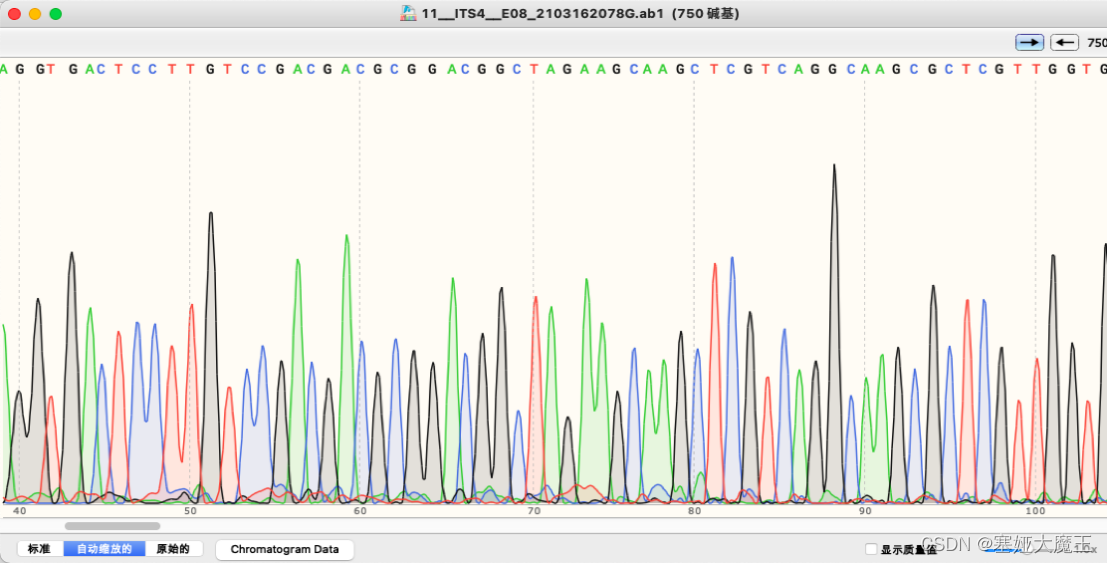
如下峰图(双峰叠加),需人工读峰图

如下峰图,其碱基序列直接舍弃

在看峰图的同时与如下序列进行比对,修改。
#在测序质量较好的情况下可直接用ITS5进行序列修改和blast。若要更为严谨,可使用ITS4和ITS5两者结合的方式进行比对(如下文中的LSU比对教程)。

编辑测序所得原始碱基序列
# ITS
ATCATTA| → → →
← ← ← GACCTCA|AATCA-
LSU
LR0R(正向)LR7(反向)序列较长,需要进行序列反向互补和序列比对。
用snapgene打开ab1文件,用记事本打开seq文件。
如下峰图,其碱基序列可直接使用

如下峰图(双峰叠加),需人工读峰图
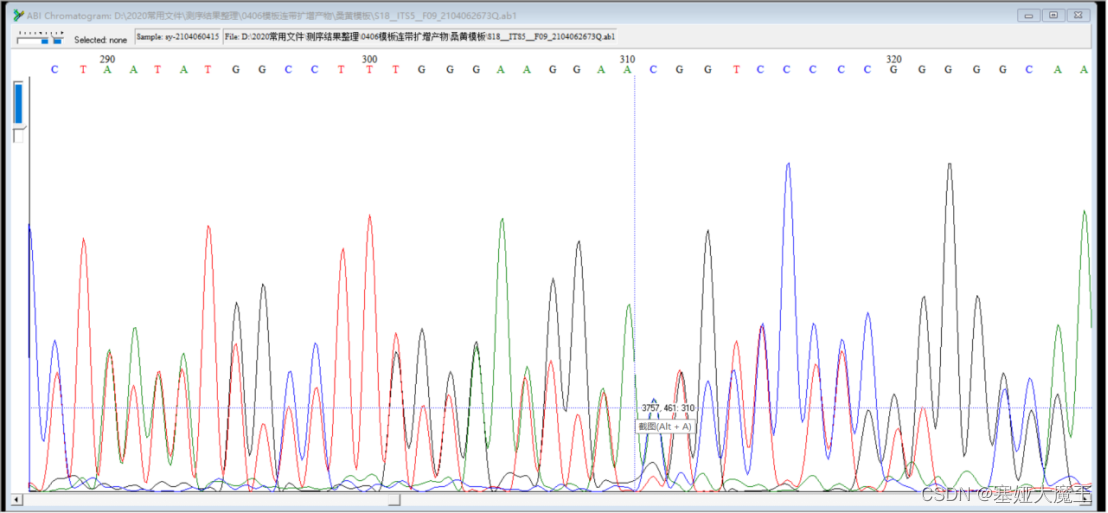
如下峰图,其碱基序列直接舍弃

在看峰图的同时与如下序列进行比对,修改。

LR7(反向)的ab1文件拖入chromas软件
点击edit中的reverse+complement

序列反向互补如下

复制上图(鼠标放在图中,ctrl+C),粘贴(ctrl+V)到LR0R所在的seq文件,入下图

删去反向互补序列间的空格,如下图
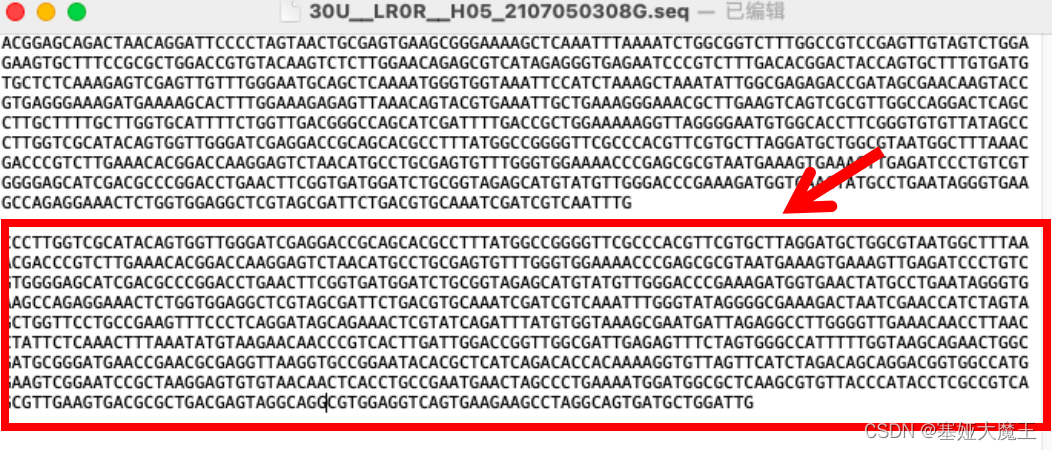
如下图,添加“>”,保存。
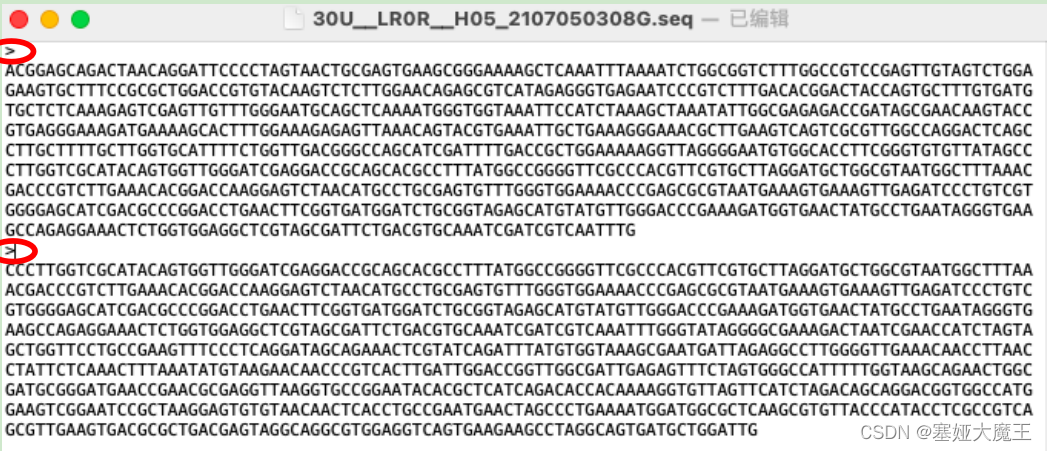
把上述文件拖入到bioedit,如下图

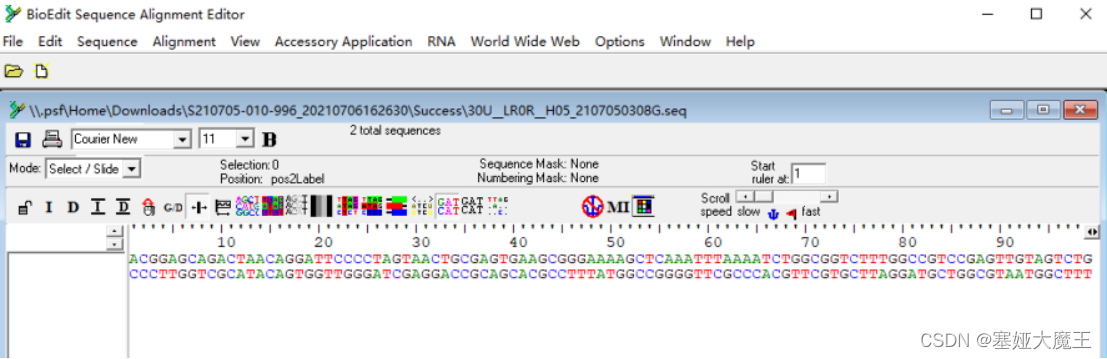
出现下图界面
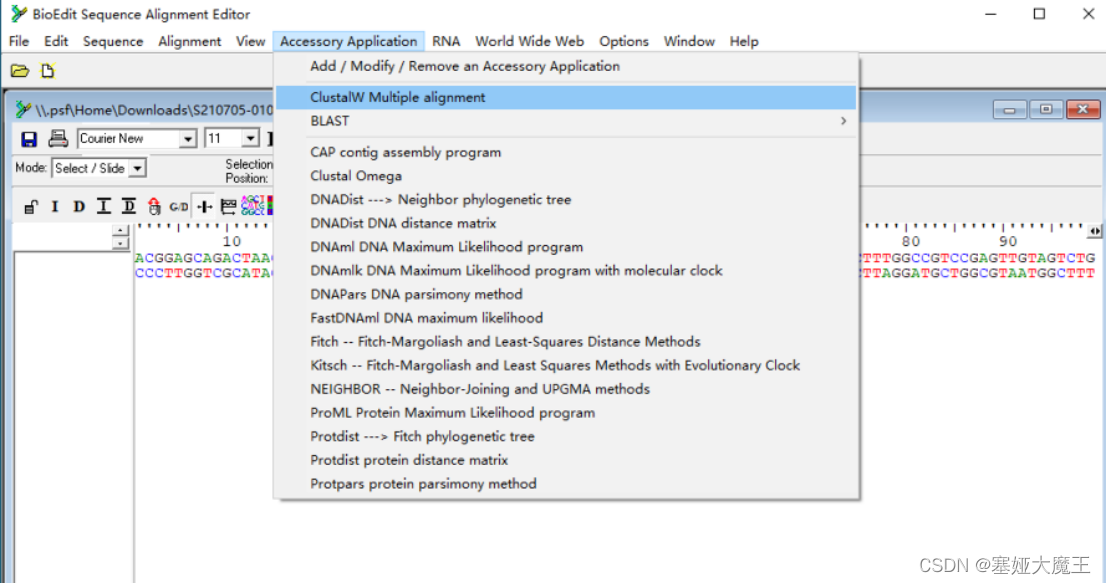
选择accessory application中的clustalw multiple alignment选项
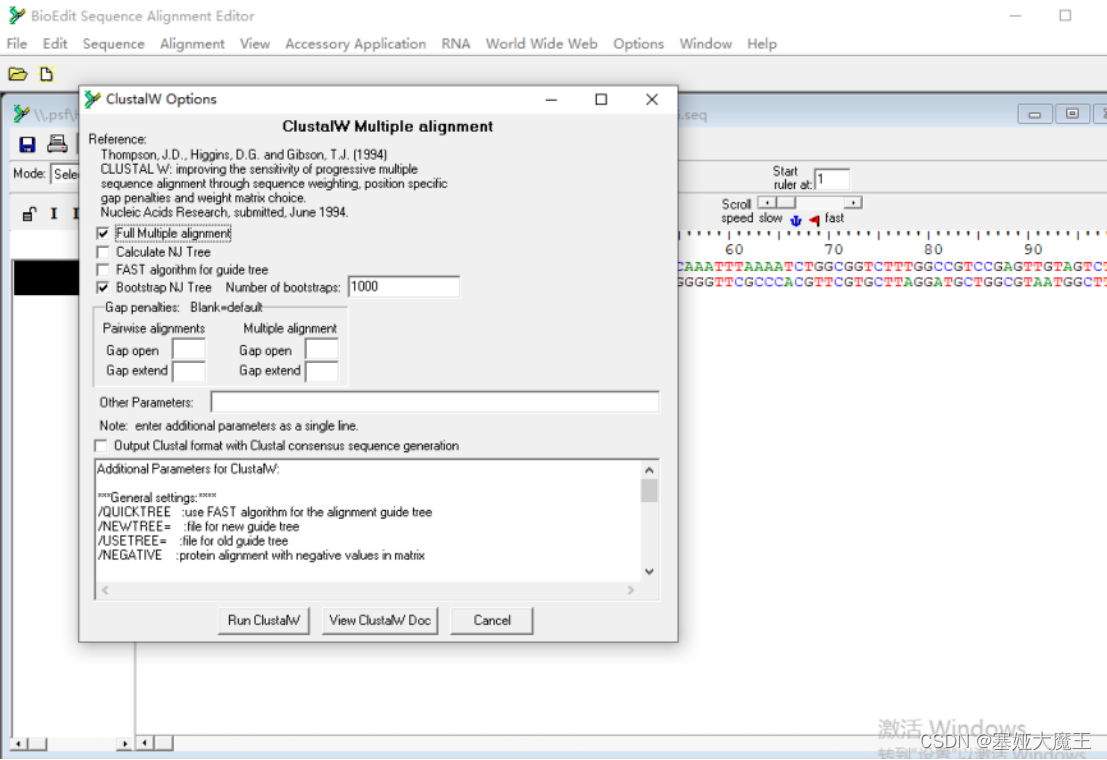
点击run clustalw,再点击ok。
出现如图界面,如图点击。

根据上图用ctrl+F查找,将序列修剪拼接,如下图

编辑测序所得原始碱基序列
# LSU
TAACAAGG|TCCCC → → →
← ← ← GAAGAAGCC|