1. 打开GDC数据库:
- 登陆TCGA数据库GDC界面:https://portal.gdc.cancer.gov/
TCGA GDC界面 -
首先确保Cart中没有之前的文件记录,如果有其他文件(即文件数不为0),清空Cart。
核对Cart已清空 -
如果Cart文件数不为0,则点击进入Cart界面进行清空。
-

清空Cart
2. 选择样本类型及性质:
- 点击Repository进入数据仓库,随后点击Cases样本类型及性质的选择:

点击Cases -
首先确定样本部位,以前列腺癌样本举例:
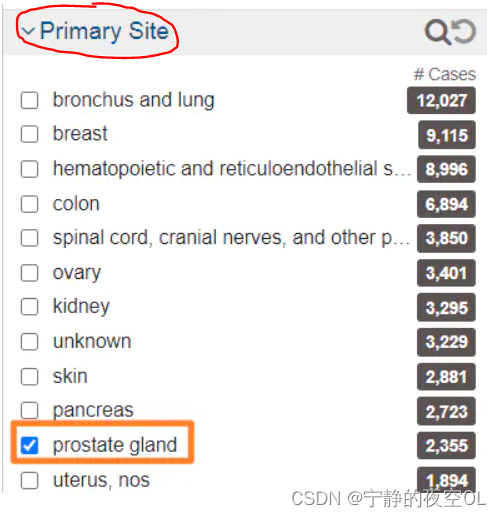
选择样本部位 -
选择样本来源项目,如果只分析TCGA的样本,则只选择TCGA:
扫描二维码关注公众号,回复: 15410287 查看本文章

选择项目来源 -
我们之前的一些选择,会不断缩小样本范围,所以我们发现Project选项下只有一个TCGA-PRAD,我们可不用点击,不选择表示该选项下的内容都要。
Disease Type这里根据分析需要进行选择,这里我为了统一病理类型,进行了选择。
Gender无特殊需要可不进行选择。
Vital Status一般我们需要进行生存分析的话,就选择alive和dead的患者,not reported的患者表示生存资料不全,可以进行剔除。
Age at Diagnosis以及Days to Death根据自己课题需要进行设定,一般情况下默认不设定筛选条件。 -
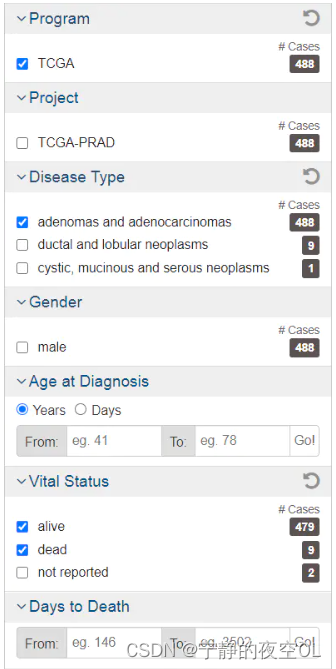
更加精细的筛选 -
Race和Ethnicity一般情况下不设定筛选条件,并且这里的nor reported的样本过于多,我们不进行筛选了,以免丢失过多样本数。
-
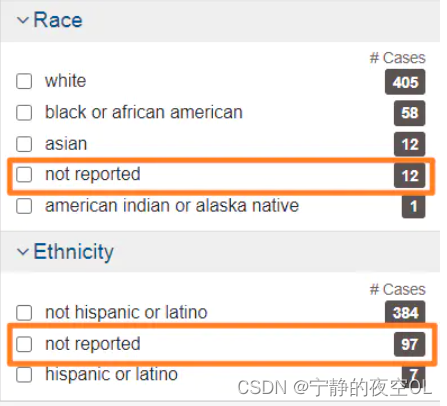
人种和族裔选择
3. 选择组学数据类型及格式:
- 点击Files选择数据类型及格式。
- Data Category这里用最常见的转录组数据举例,选择transcriptome profiling.
- Data Type选择Gene Expression Quantification,代表蛋白编码基因和长链非编码基因的测序数据。miRNA基因的测序数据不包含在其中,需要选择miRNA Expression Quantification而不是Gene Expression Quantification。
- Experimental Strategy只有一个选择,默认不选,Workflow Type根据自己需求,一般常用的是Counts数据或FPKM数据。
*一般选到这里就不再点击其他筛选条件了,而且一般其他选项也只剩一个选项了。 -
Access表明数据权限,我们普通用户只能使用open的数据,如果出现了非开放的数据,记得这里只点击open。

选择数据类型及格式
4. 下载选择好的数据:
-
将选择好的数据加入购物车,随后点击Cart进入购物车界面。

将选择好的数据加入购物车 - 在Cart界面分别点击Metadata(下载注释文件)以及Download(下载数据)。Download选项提供两种数据下载途径:Manifest表示下载Manifest文件后使用gdc-client软件下载数据(gdc-client下载数据方法),这种方法适合下载大文件;Cart表示通过浏览器直接下载,该方法更方便,但是不适合下载很大的文件。
-

数据的两种下载方式 -
至此TCGA数据下载已完成。
5、TCGA文件的命名规则
TCGA:Project名, 所有TCGA样本名均以这个开头。
02: issue source site,组织来源编码。更多标注:https://gdc.cancer.gov/resources-tcga-users/tcga-code-tables/tissue-source-site-codes
0001: Participant, 参与者编号。一个患者可能会对应多个样本,如TCGA-A6-6650可以得到3个样本数据:TCGA-A6-6650-01A-11R-1774-07,TCGA-A6-6650-01A-11R-A278-07,TCGA-A6-6650-01B-02R-A277-07
01: Sample,关键数字,其中编号01~09表示肿瘤,10~19表示正常对照
A: Vial, 在一系列患者组织中的顺序,绝大多数样本该位置编码都是A; 很少数的是B
01: Portion, 同属于一个患者组织的不同部分的顺序编号
D: Analyte, 分析的分子类型
0182: Plate, 在一系列96孔板中的顺序,值大表示制板越晚
07: Center, 测序或鉴定中心编码

6、通过 GDC Data Transfer Tool读取数据
①原始方法:
- 将下载下来的压缩包进行解压缩,得到gdc-client.exe。将MANIFEST.txt文件和gdc-client.exe放在一个文件夹下。

-
在该文件目录下打开cmd命令窗口。
-
输入gdc-client download -m MANIFEST.txt (注:-m 后加的是下载好的manifest文件,需要改成自己的文件名。还可以在后方加--latest,表示最新文件数据,下载临床数据的时候比较方便),按Enter键,开始下载。
-
gdc-client download -m MANIFEST.txt #or gdc-client download -m MANIFEST.txt --latest
下载页面
②下载数据+预处理数据:
Downloading TCGA cohorts + WSI pre-processing
- Download the GDC Data Transfer Tool executable (not included here for license issues)
- Constitute any cohort on the TCGA GDC Data Portal, then download the associated manifest file, and place it in a
source_folder - Launch the download and pre-processing pipeline with
python -m code.data_processing.main --gdc gdc_executable_path source_folderThis script first downloads all files in the manifest file, then tiles WSI, extracts tiles of a given magnification, removes background tiles, and finally seeks to extract per-slide binary labels from their name.