近日,腾讯 AI Lab 三项研究分别入选国际顶级学术期刊 Nature Methods 和 Nature Communications,再次展示了在生命科学前沿领域上国际领先的技术实力。
这三项研究成果都属于生物细胞研究中的空间组学技术,对于推动精准医疗、细胞图谱绘制、人类生命健康等领域的研究具有重要意义。
细胞是生命的基本单位,但当前人类对细胞的认知有限。空间组学技术的出现和发展,让我们可以获取细胞在时间和空间维度上的基因表达模式以及细胞之间交互的情况,从高精度的分子层面去理解器官和组织的功能,这对于理解细胞生物学,发育生物学,神经生物学,肿瘤生物学等的关键信息至关重要,可以填补组织和器官水平上位置与功能关系研究的空白。
空间组学技术是高通量转录组测序、单细胞测序等技术的升级,它通过在细胞分析中,增加「空间」这一重要的信息维度,使得研究员可以从更全局、立体、精细地认识生物系统的运行规律。
近年来,通过深度学习等人工智能技术的引入,空间组学技术领域不断迎来新的突破,本次腾讯 AI lab 的三项成果,分别从细胞类型注释、微环境建模以及数据库三个方面重点突破,并在准确率、数据规模以及方法创新性上均展示了超出业界标准的水平,推动国际学术社区和相关研究的发展。
它们分别是:
-
一种基于迁移学习和空间嵌入的空间转录组细胞类型注释方法 Spatial-ID
-
一种利用空间组学数据进行微环境建模的通用方法 SOTIP
-
一个目前业界最大规模(超过5000万个细胞)、最多种类(26种)的空间组学数据库
在空间组学相关技术上,腾讯 AI Lab 的核心攻坚方向和优势在于 AI 算法研究,长期以来,腾讯 AI Lab 与业内知名研究所和医院广泛合作,实现在生命科学的研究和临床场景中落地,具体应用包括建立细胞图谱,尤其是灵长类大脑图谱,促进脑科学的研究。
临床方面,腾讯 AI Lab 将通过空间组学研究肿瘤的微环境和发展轨迹,助力推进有针对性的精准医疗。
腾讯在医疗及生命科学领域已有多项探索。2022年, 腾讯 AI Lab 联合北京协和医院,共同发布了便携式智能化手术导航系统,临床初步应用取得成功。此外,实验室创新性地提出关于单细胞注释的 scBERT 算法,成果发表于国际顶级学术期刊《Nature Machine Intelligence》杂志。
Nature Methods 是 Nature(《自然》)系列期刊中的关注前沿科学研究的期刊,每年评选生命科学领域的年度方法,2022 年期刊影响因子 47.99,在生物研究方法领域排名第一。Nature Communications 是 Nature(《自然》)系列期刊中的综合类子刊,专门收录来自于自然科学各个领域的高质量研究成果,2022 年期刊影响因子 17.69,在综合类期刊中位列第三。
三项研究详解
研究1:一种基于迁移学习和空间嵌入的空间转录组细胞类型注释方法 Spatial-ID
英文标题:Spatial-ID: a cell typing method for spatially resolved transcriptomics via transfer learning and spatial embedding.
论文链接:https://www.nature.com/articles/s41467-022-35288-0)
该研究利用了单细胞转录组中细胞类型的细胞表达谱信息作为参考知识,并采用图神经网络的算法描绘空间转录组中细胞的空间位置关系,提供了一种快速的空间转录组细胞类型注释方法。
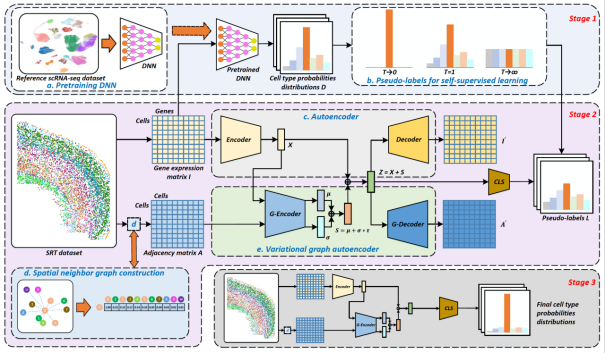
图1:Spatial-ID 算法流程
迁移学习可从已有的单细胞转录组数据集迁移单细胞表达谱知识。空间信息嵌入则利用细胞在空间背景下与相邻细胞之间的可能存在的交互关系或共表达模式,提升细胞类型识别的准确性。
从结果上看,在四个不同的空间转录组公开数据集(两个鼠脑数据集,一个鼠脑生殖细胞数据集,一个人类非小细胞肿瘤数据集)上开展基准测试,与现有 8 种 SOTA 方法(Seurat, SingleR, Scmap, Cell-ID, ScNym, SciBet, Tangram, Cell2location)进行性能对比。
Spatial-ID 在 4 个数据集上分别取得了 92.75%,87.74%,60.45%和 69.76% 的准确性,均显著地优于 SOTA 方法;尤其是在小鼠下丘脑视前区的三维空间转录组数据集上,与最佳的 SOTA 方法对比,Spatial-ID 的细胞类型注释平均准确性提升了约 6.5%。
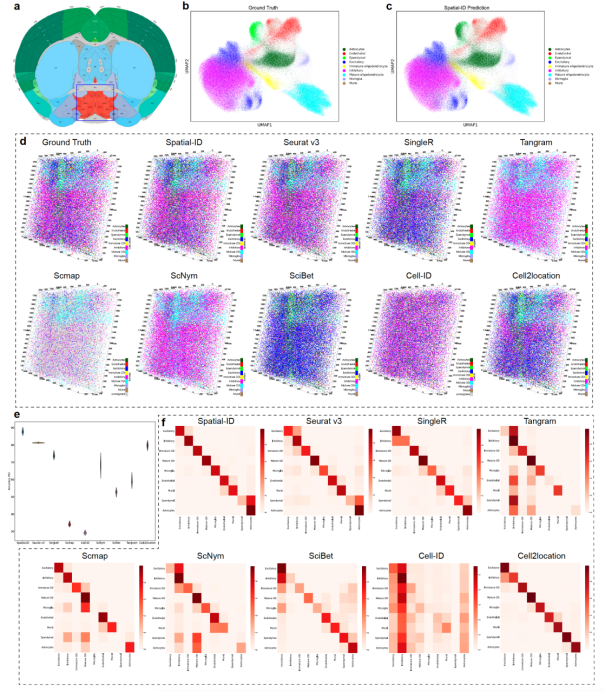
图2:小鼠下丘脑视前区的三维空间转录组数据集基准测试结果
另外,Spatial-ID 还提供了新细胞类别发现的流程,发现参考数据集中没有的细胞类型。
据论文作者介绍,基于空间转录组的细胞类型注释对单个细胞中基因的特异性表达和细胞所处空间微环境展开研究,系统性地分类组织中的细胞类型,并统一描绘每一种细胞类型的分子特征以及它们在组织内的位置,这将改变我们对生物学和疾病的理解,并可能带来疾病诊断和治疗方式的重大突破。
未来,Spatial-ID 空间转录组细胞注释算法,可以为大规模空间转录组测序数据进行细胞类型注释,致力于构建大规模的组织细胞图谱,例如全鼠脑/猴脑的空间转录组细胞图谱。
研究2:一种利用空间组学数据进行微环境建模的通用方法 SOTIP
英文标题:SOTIP is a versatile method for microenvironment modeling with spatial omics data
论文链接:https://www.nature.com/articles/s41467-022-34867-5
该研究利用人工智能领域的最优传输理论,同时结合细胞在物理空间和状态空间的连续性,对微环境进行建模。
其亮点在于,通过构建微环境之间的相互关系网络,将细胞分子表达谱的低维流形与空间局部拓扑特征联系起来,达到多个重要计算任务的同时分析,包括微环境异质性定量、空间域识别及差异微环境分析。
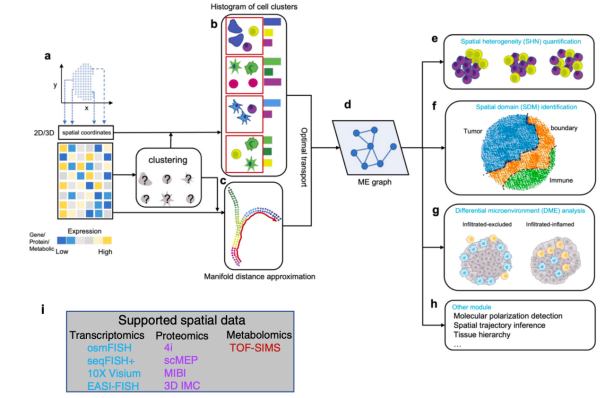
SOTIP 在多种空间转录组、蛋白组和代谢组数据的测试中展现出较好的准确性、稳定性和鲁棒性。
在空间异质性定量方面,SOTIP 在亚细胞层面上精准勾画了肿瘤细胞核膜和内质网膜的轮廓 (AUC=0.85);在组织层面上识别了肿瘤和正常肌肉组织的边界(Spearman系数=0.847)。
在空间域识别方面,SOTIP 在多种空间蛋白组和转录组数据中呈现出较高的准确性,精确识别不同脑区及肿瘤结构,在人类脑区识别中达到了 0.58 的 ARI,效果优于 BayesSpace、SpaGCN、STAGATE 等经典算法,并能适用于三维空间数据。
在差异微环境分析方面,SOTIP 利用识别出的微环境发现了三阴性乳腺癌的两种亚型,在 34 个病人队列中有着显著不同的预后(显著性p value=9.2*10^-6)。
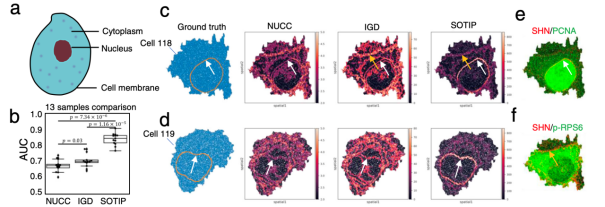
SOTIP 的两大应用场景是脑科学研究和肿瘤。
脑科学研究中的一大挑战是研究不同神经细胞类型之间、脑功能区之间、及神经细胞与功能区之间的相互作用关系。SOTIP 可以无需任何人工干预地、准确识别大脑中不同功能性空间域,为大规模脑图谱的构建建立基础。
肿瘤最重要的细胞类型是免疫细胞和肿瘤细胞,它们之间的空间临近关系和相互作用构筑了复杂的肿瘤微环境。在临床诊断方面,许多疾病的分型无法通过免疫细胞和肿瘤细胞的各亚类的数量比例完成,SOTIP 前景是可以从微环境层面而不单单是细胞层面,识别不同肿瘤亚类的标志性微环境,从而对病人进行针对性治疗。
研究3:目前业界最大规模(超过5000万个细胞)、最多种类(26种)的空间组学数据库
英文标题:SODB facilitates comprehensive exploration of spatial omics data
论文链接:https://www.nature.com/articles/s41592-023-01773-7
对于生物学家来说,新的生物学和病理学发现理应用不同的技术和分子组学来验证,减少科学发现中的假阳性。将各种空间组学数据集中在一起并进行逻辑管理,使研究员可以根据需要快速搜索、定位和获取多模态数据,从而充分利用已发布的数据,避免一些不必要的生物实验。
从生物信息学家的角度来看,SODB 可以支持许多计算方法的 benchmark数据需求,让方法开发人员只需专注于计算模型本身。
该数据库提供了目前业界最大规模(超过 5000 万个细胞)、最多种类(26 种空间组学技术)的空间组学数据,所有数据均由标准流程处理为 Anndata 形式(空间组学业界标准格式)。并提供了多种数据分析和新型可视化模块,快速地对整个组织进行可视化并识别组织区域。

此外,还提供了配套 Python 工具包 pysodb,仅用一行代码可以方便的读取数据,时间效率比传统方式提升 160 倍(以 Slide-seq 数据为例,传统方法读取需要 19.04 分钟,pysodb 仅需 7.16 秒)。
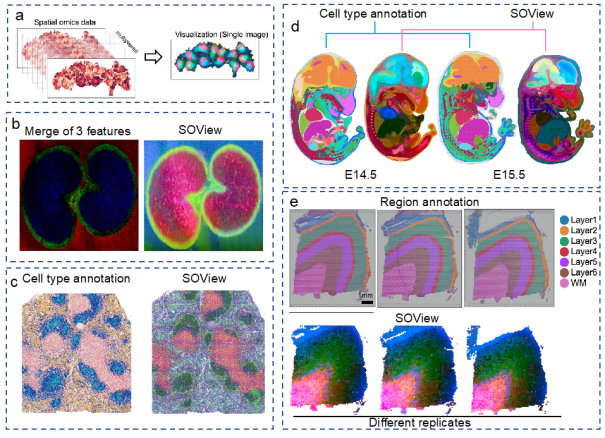
目前,该数据库的数据开放给所有人:
数据集:https://gene.ai.tencent.com/SpatialOmics/
Python包:https://github.com/TencentAILabHealthcare/pysodb
参考链接:
1. Shen, R., Liu, L., Wu, Z. et al. Spatial-ID: a cell typing method for spatially resolved transcriptomics via transfer learning and spatial embedding. Nat Commun 13, 7640 (2022). https://doi.org/10.1038/s41467-022-35288-0
2. Yuan, Z., Li, Y., Shi, M. et al. SOTIP is a versatile method for microenvironment modeling with spatial omics data. Nat Commun 13, 7330 (2022). https://doi.org/10.1038/s41467-022-34867-5
3. Yuan, Z., Pan, W., Zhao, X., et al. SODB facilitates comprehensive exploration of spatial omics data. Nat Methods (2023). https://doi.org/10.1038/s41592-023-01773-7