如果用一句话概括遗传算法: 在程序里生宝宝, 杀死不乖的宝宝, 让乖宝宝继续生宝宝.
步骤:
找一个好的fitness方程
所有的遗传算法 (Genetic Algorithm), 后面都简称 GA, 我们都需要一个评估好坏的方程, 这个方程通常被称为 fitness. 在今天的问题中, 我们找到下面这个曲线当中的最高点. 那么这个 fitness 方程就很好定, 越高的点, fitness 越高.
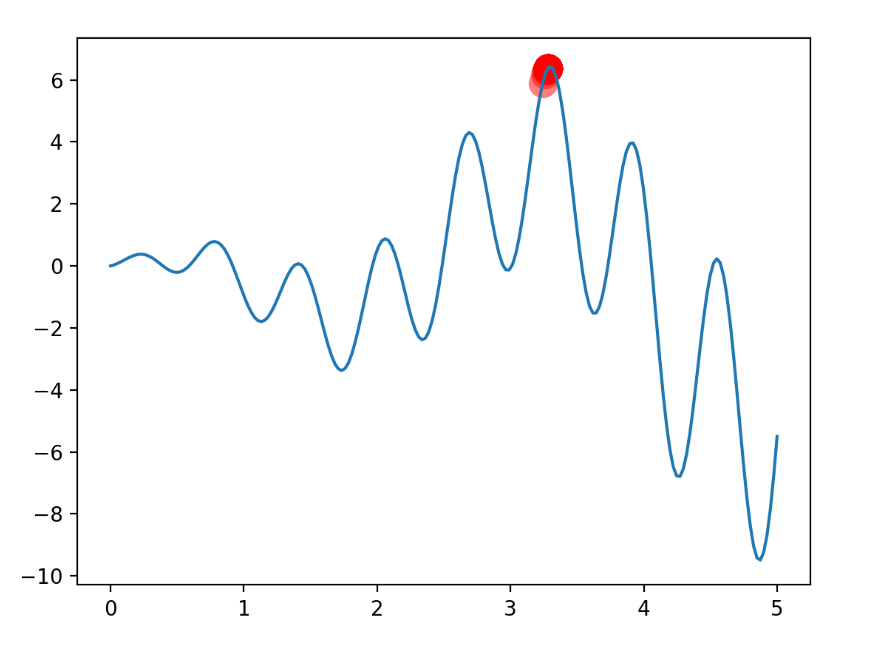
如果这个曲线上任一点的 y 值是 pred 的话, 我们的 fitness 就是下面这样:
def get_fitness(pred):
return predDNA 编码
在 GA 中有基因, 为了方便, 我们直接就称为 DNA 吧. GA 中第二重要的就是这 DNA 了, 如何编码和解码 DNA, 就是你使用 GA 首先要想到的问题. 传统的 GA 中, DNA 我们能用一串二进制来表示, 比如:
DNA1 = [1, 1, 0, 1, 0, 0, 1]
DNA2 = [1, 0, 1, 1, 0, 1, 1]但是长成这样的 DNA 并不好使用. 如果要将它解码, 我们可以将二进制转换成十进制, 比如二进制的 11 就是十进制的 3.
但是有时候我们会需要精确到小数, 其实也很简单, 只要再将十进制的数浓缩一下就好. 比如我有 1111 这么长的 DNA, 我们产生的十进制数范围是 [0, 15], 而我需要的范围是 [-1, 1], 我们就将 [0, 15] 缩放到 [-1, 1] 这个范围就好.
def translateDNA(pop):
return pop.dot(2 ** np.arange(DNA_SIZE)[::-1]) / float(2**DNA_SIZE-1) * X_BOUND[1]
注意, 这里的 pop 是一个储存二进制 DNA 的矩阵, 他的 shape 是这样 (popsize, DNAsize).
选择
适者生存的 select() 很简单, 我们只要按照适应程度 fitness 来选 pop 中的 parent 就好. fitness 越大, 越有可能被选到.
def select(pop, fitness):
idx = np.random.choice(np.arange(POP_SIZE), size=POP_SIZE, replace=True,
p=fitness/fitness.sum()) # p 就是选它的比例
return pop[idx]
交叉
接下来进行交叉配对. 方式很简单. 比如这两个 DNA, Y 的点我们取 DNA1 中的元素, N 的点取 DNA2 中的. 生成的 DNA3 就有来自父母的基因了.

这里从 pop_copy 中随便选一个当另一个父辈 和 parent 进行随机的 crossover
变异
mutation 将某些 DNA 中的 0 变成 1, 1 变成 0.
def mutate(child):
for point in range(DNA_SIZE):
if np.random.rand() < MUTATION_RATE:
child[point] = 1 if child[point] == 0 else 0
return child整体流程:

代码:
"""
Visualize Genetic Algorithm to find a maximum point in a function.
Visit my tutorial website for more: https://mofanpy.com/tutorials/
"""
import numpy as np
import matplotlib.pyplot as plt
import matplotlib
matplotlib.use('TkAgg')
DNA_SIZE = 10 # DNA length
POP_SIZE = 100 # population size
CROSS_RATE = 0.8 # mating probability (DNA crossover)
MUTATION_RATE = 0.003 # mutation probability
N_GENERATIONS = 200
X_BOUND = [0, 5] # x upper and lower bounds
# 得到当前DNA对应的函数F值(适应度),可能为负数
def F(x):
return np.sin(10*x)*x + np.cos(2*x)*x
# 将上一步F得到的适应度进一步处理,全部变为正数,最小值为0.001
def get_fitness(pred):
return pred + 1e-3 - np.min(pred)
# 将二进制的DNA转换为十进制,并将其归一化到(0,5)的范围内【0 1 0 0 1 0 0 1 0 1】--> 0.4512
def translateDNA(pop):
return pop.dot(2 ** np.arange(DNA_SIZE)[::-1]) / float(2**DNA_SIZE-1) * X_BOUND[1]
# nature selection wrt pop's fitness
def select(pop, fitness):
idx = np.random.choice(np.arange(POP_SIZE), size=POP_SIZE, replace=True, p=fitness/fitness.sum())
return pop[idx]
# 交叉:随机选择一个点,将其后面的值替换为另一个个体的值
def crossover(parent, pop):
if np.random.rand() < CROSS_RATE:
i_ = np.random.randint(0, POP_SIZE, size=1) # select another individual from pop
cross_points = np.random.randint(0, 2, size=DNA_SIZE).astype(np.bool) # choose crossover points
parent[cross_points] = pop[i_, cross_points] # mating and produce one child
return parent
# 变异:随机选择一个点,将其值取反
def mutate(child):
for point in range(DNA_SIZE):
if np.random.rand() < MUTATION_RATE:
child[point] = 1 if child[point] == 0 else 0
return child
# 初始化人群的DNA,每一行代表一个人的DNA:(100,10)【0 1 0 0 1 0 0 1 0 1】
pop = np.random.randint(2, size=(POP_SIZE, DNA_SIZE))
plt.ion()
x = np.linspace(*X_BOUND, 200)
plt.plot(x, F(x))
for _ in range(N_GENERATIONS):
# 1、计算当前DNA的适应度:计算当前DNA对应的函数F值,得到可能为负数的适应度
F_values = F(translateDNA(pop))
if 'sca' in globals():
sca.remove()
sca = plt.scatter(translateDNA(pop), F_values, s=200, lw=0, c='red', alpha=0.5); plt.pause(0.05)
# 将可能为负数的适应度进一步处理,全部变为正数,最小值为0.001
fitness = get_fitness(F_values)
print("Most fitted DNA: ", pop[np.argmax(fitness), :])
# 2、选择父母:在当前人群pop中,根据适应度fitness的大小,选择POP_SIZE个个体,作为下一代的父母
pop = select(pop, fitness)
pop_copy = pop.copy()
# 3、父母进行交叉得到孩子:这里父母是完全相同的,遍历每一个父亲的值,让它与随机一个目前的后半段交叉
for parent in pop:
child = crossover(parent, pop_copy)
# 4、孩子还有可能自己发生变异
child = mutate(child)
# 5、将当前的孩子替换掉父母
parent[:] = child
plt.ioff(); plt.show()