Efficient evolution of human antibodies from general protein language models
哈佛大学化学与化学生物学系和圣路易斯华盛顿大学的研究人员共同完成的一篇论文,发表在Nature Biotechnology上。
抗体是一种大分子,属于免疫球蛋白家族,它们的分子量通常在150 kDa以上,具有高度特异性的结合能力,可以与抗原结合并进行免疫反应。
这篇文章介绍了一种使用通用蛋白质语言模型来加速人类抗体进化的方法。研究人员训练了一种基于大规模蛋白质序列数据的语言模型,该模型可以预测蛋白质序列的下一个氨基酸。然后,他们使用这个模型来预测在人类抗体的可变区域中进行的突变,并针对这些突变进行计算机模拟,以确定哪些突变可能会增加抗体的亲和力。最终,他们使用这种方法成功地改进了三种抗体的亲和力。这种方法可以提高人类抗体的快速进化,并有望在药物开发中发挥重要作用。
近年来,抗体的制备技术在医学领域的应用越来越广泛。然而,高亲和力的抗体的制备仍然是一项具有挑战性的任务。为了提高抗体的亲和力,科学家们通常会利用定向进化技术,但这种方法需要大量的实验操作和时间。本项研究不依赖于特定的抗体序列或结构信息,而是利用通用的蛋白质语言模型,根据自然界的进化规律,筛选出符合要求的氨基酸替换,从而提高抗体的亲和力。这种方法为开发更有效的抗体提供了新思路。下面我们将对这项研究进行详细介绍。
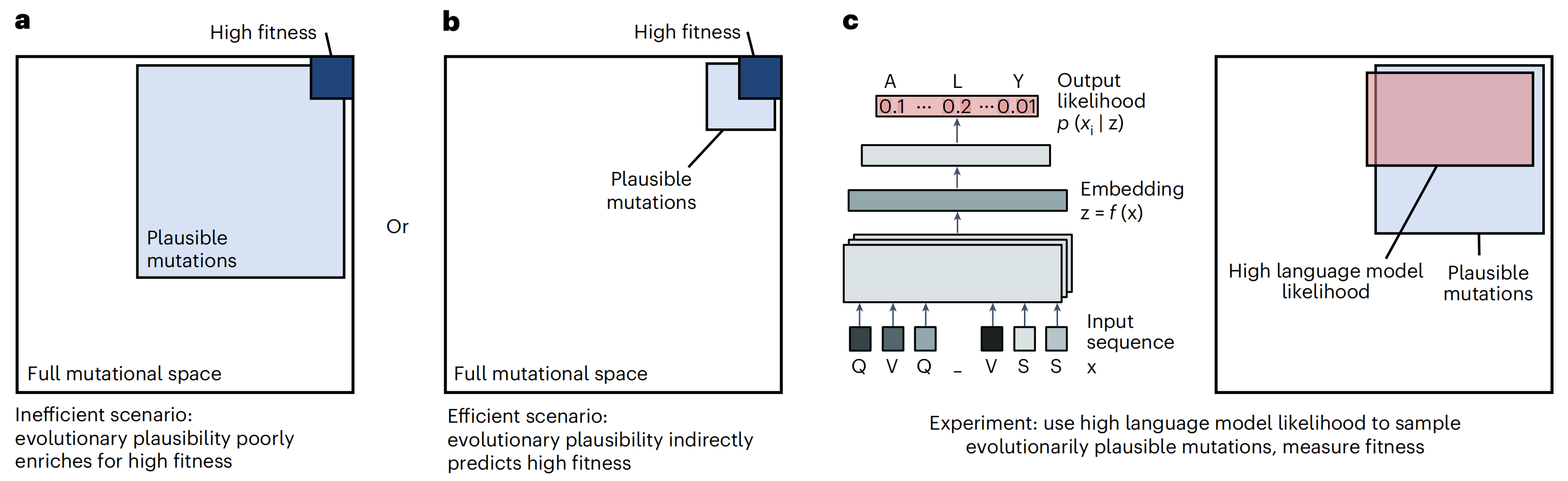
Fig. 1 | Guiding evolution with protein language models. a,b, Two possible models for relating the space of mutations with high evolutionary plausibility (for example, mutations seen in antibodies) to the space with high fitness under specific selection pressures (for example, mutations that result in high binding affinity to a specific antigen). Both models assume that mutations with high fitness make up a rare subset of the full mutational space and that, in general, high-fitness mutations are also evolutionarily plausible. Under the first model (a), mutations with high fitness are rare within the subset of mutations that are evolutionarily plausible. Under the second model (b), when restricted to the regime of plausible mutations, improvements to fitness become much more common. c, Protein language models, trained on millions of natural protein sequences learn amino acid patterns that are likely to be seen in nature. We hypothesized that most mutations with high language model likelihood would also be evolutionarily plausible. Assuming that this is true, and if the second model (b) better describes nature, then a language model with no information about specific selection pressures can still efficiently guide evolution.
通过广义语言模型获得氨基酸替代方案的方法
该研究分别使用多个蛋白质语言模型预测目标抗体中可能进行的突变,并使用多个模型一致推荐的预测方案得到符合要求的氨基酸替换策略。
我们将一个给定的原始序列x作为输入

,其中, 是一组氨基酸序列,N代表该序列的长度。另外我们还需要一组经过预训练的masked language models用以产生条件似然概率。为了指导基于特定语言模型的进化,我们首先计算语言模型可能性高于野生类型的替换集,其公式可表示如下:

其中 表示其中一个语言模型,表示野生型抗体的一个氨基酸残基,并且.为了进一步过滤仅具有最高可能性的替换方案,我们选择多个模型一致推荐的预测方案作为最终的替换方案,其中,对于新的氨基酸,我们计算

然后,我们在多个语言模型中获得比野生型更高可能性的替换集,该替换集可用公式表示如下:

其中是截止值,其控制要测量的对应变体的数量。
在这篇文章中使用了六个大规模的masked language models,即来自 https://github.com/facebookresearch/esm 的ESM-1b模型和五个模型,它们组合在一起形成ESM-1v20。ESM-1b是在UniRef50的2018-03版本上进行训练的,该版本包含约2700万个序列,而ESM-1v中的五个模型则分别在UniRef90的2020-03版本上进行训练,该版本包含约9800万个序列。
通过使用语言模型得到符合要求的氨基酸替换后,再从这些方案中筛选出可能会增加抗体亲和力的替换方案。然后,他们使用计算机模拟来评估这些替换方案的效果,并选择最优的替换进行实验验证。
实验结果
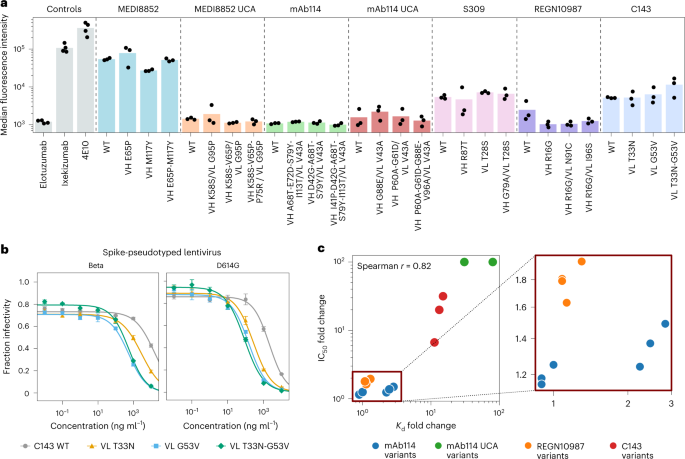
作者通过在语言模型的指导下进行抗体的模拟进化来验证文章中的假设,以亲和力成熟的七种抗体作为本次实验的对象,这七种抗体分别是MEDI8852,MEDI8852 UCA,mAb114,mAb114 UCA,S309,REGN10987,C143。作者使用ESM-1b语言模型和ESM-1v五种语言模型的集合(总共六种语言模型)进行了进化。作者使用这些语言模型来计算对重链(VH)或轻链(VL)的抗体可变区的所有单个氨基酸替换的可能性,并选择其中具有更高进化可能性的替换。这些替换在六个语言模型的一致结果下高于野生型。在第一轮进化中,通过含有一个单个氨基酸替换的变异体的生物膜干涉技术(BLI)来测量抗体与抗原相互作用强度。在第二轮中,测量了经过替换组合后的变体,并根据第一轮的结果选择原抗体或经过氨基酸替代的变异体。作者对所有七种抗体进行了这两轮进化,每种抗体在第一轮中测量了8-14个变体,在第二轮中每种抗体测量了1-11个变体,实验结果如下图a所示。另外,下图b说明了从野生型到每种抗体的最高亲和力变体的进化轨迹。经过这两轮的进化之后得到的最高亲和力的变体与原抗体的对比如下图c所示。

另外,作者还对进化后抗体的附加特征进行了详细的分析,作者发现,在推荐的31种语言模型中,有21种的Fabs,测试的亲和力增强变体比野生型具有更高的熔化温度(Tm),并且所有变体都保持热稳定性(Tm > 70 °C)。当S309进化为具有更高的亲和力时,最佳的Tm为72.8 °C,相比之下野生型为72.5°C,而sotrovimab中引入VH N55Q取代策略将Tm降低至69.6°C,如上图b所示。mAb114、mAb114-UCA、REGN10987和C143的进化变体也保留或改善了Tm;当进化出mAb114 UCA时,作者观察到的最高变化是从74.5 °C增加至82.5°C。然而,热稳定性的改善并不能完全解释亲和力成熟结果,因为作者观察到MEDI8852及其UCA的亲和力成熟变体的Tm有所降低,尽管这些Fab仍然是热稳定性的。
此外,作者还测试了亲和力成熟设计的多种可能的特异性结合,因为在治疗环境中结合非预期的靶标可能会导致不良的副作用。对于七种抗体中的每一种,作者使用评估与可溶性膜蛋白非特异性结合的多特异性测定法测试了野生型和三种亲和力成熟的变体。作者观察到所有七种抗体的任何变体的多特异性没有实质性变化,并且所有测试的抗体的多特性值都在治疗可行范围内,如下图a所示。
另外,本文还研究了亲和力成熟变体是否具有更好的病毒中和活性。作者使用灭话病毒中和试验(方法)测试了四种抗体的亲和力增强变体,在所有情况下,变体的半最大抑制浓度值(IC50)有了显著的改善,包括针对埃博拉假病毒的最佳mAb114变体也发生了1.5倍的提升;针对严重急性呼吸系统综合征冠状病毒2β灭活病毒的最佳REGN10987变体发生了两倍提升;针对贝塔灭活病毒的最佳C143变体提高了32倍,如下图b所示。此外,与野生型相比,mAb114 UCA的亲和力成熟变体在>100倍的低浓度下表现出可检测的中和作用。一般来说,结合亲和力的变化与中和作用的变化很好地相关,如下图c所示。
结论
本研究通过基于通用蛋白质语言模型的方法实现了人类抗体的高效进化,成功地提高了抗体的亲和力和特异性。研究结果表明,通用蛋白质语言模型可以作为一种有效的工具来指导抗体亲和力成熟的研究,有望在生物医药领域发挥重要作用。