1. ANOVA模型拟合
1.1 aov()函数
语法:aov(formula, data = dataframe)
R表达式中的特殊符号:

R语言中常见研究设计的表达式:

1.2 表达式中各项的顺序
表达式中各项的顺序在两种情况下会造成影响,例如,y ~ AB与y ~ BA的结果不同:
- 因子有多个,且非平衡设计 (即每组样本数不同)
- 存在协变量 (每个样本之间会有差别,而引起这些差别的变量叫做协变量)
R语言默认为类型I(序贯型)
又如:y~A+B+A:B:
- A对y的影响
- 控制A时,B对y的影响
- 控制A和B的主效应时,A与B的交互效应
各因子的顺序对结果的影响:

样本大小越不平均,效应项顺序对结果的影响越大。所以,越基础性的因子越应该放在表达式前面,即协变量应该放在主变量之前,接下来是双因素的交互项,再接下来是三因素的交互项。
对于主效应,越基础的变量越应该放在表达式的前面,比如,性别应该放在处理方式的前面。
car包中的Anova()函数提供使用类型2和类型3方法,aov()函数使用类型1方法。
2. 单因素方差分析(One-Way ANOVA)
单因子方差分析,比较分类因子定义的两个或者多个组别中因变量均值。以multcomp包中的cholesterol数据集为例(取自Westfall、Tobia、Rom、Hochberg,1999),50个患者均接受降低胆固醇药物治疗(trt)五种疗法中的一种疗法。其中三种治疗条件使用药物相同,分别是20mg一天一次(1time)、10mg一天两次(2times)和5mg一天四天(4times)。剩下的两种方式(drugD和drugE)代表候选药物。哪种药物疗法降低胆固醇(响应变量)最多?
install.packages('multcomp')
library(multcomp)
attach(cholesterol) #打开连接:直接使用其中的变量名而不需要使用符号
colnames(cholesterol)
table(trt)
## 计算各组平均值mean
## aggregate()函数是用于对数据进行分组统计的函数:
# aggregate()函数是用于对数据进行分组统计的函数。
# response是一个向量,trt是一个因子变量,表示不同的处理组。
# aggregate(response, by=list(trt), FUN=mean)的作用是计算response向量中每个处理组的平均值
aggregate(response, by=list(trt), FUN=mean)
response
## 计算各组标准差sd
aggregate(response, by=list(trt), FUN=sd)
## 检验组间差异
fit <- aov(response ~ trt)
summary(fit)
#自由度 平方和 均方和 F值 P值
结果:
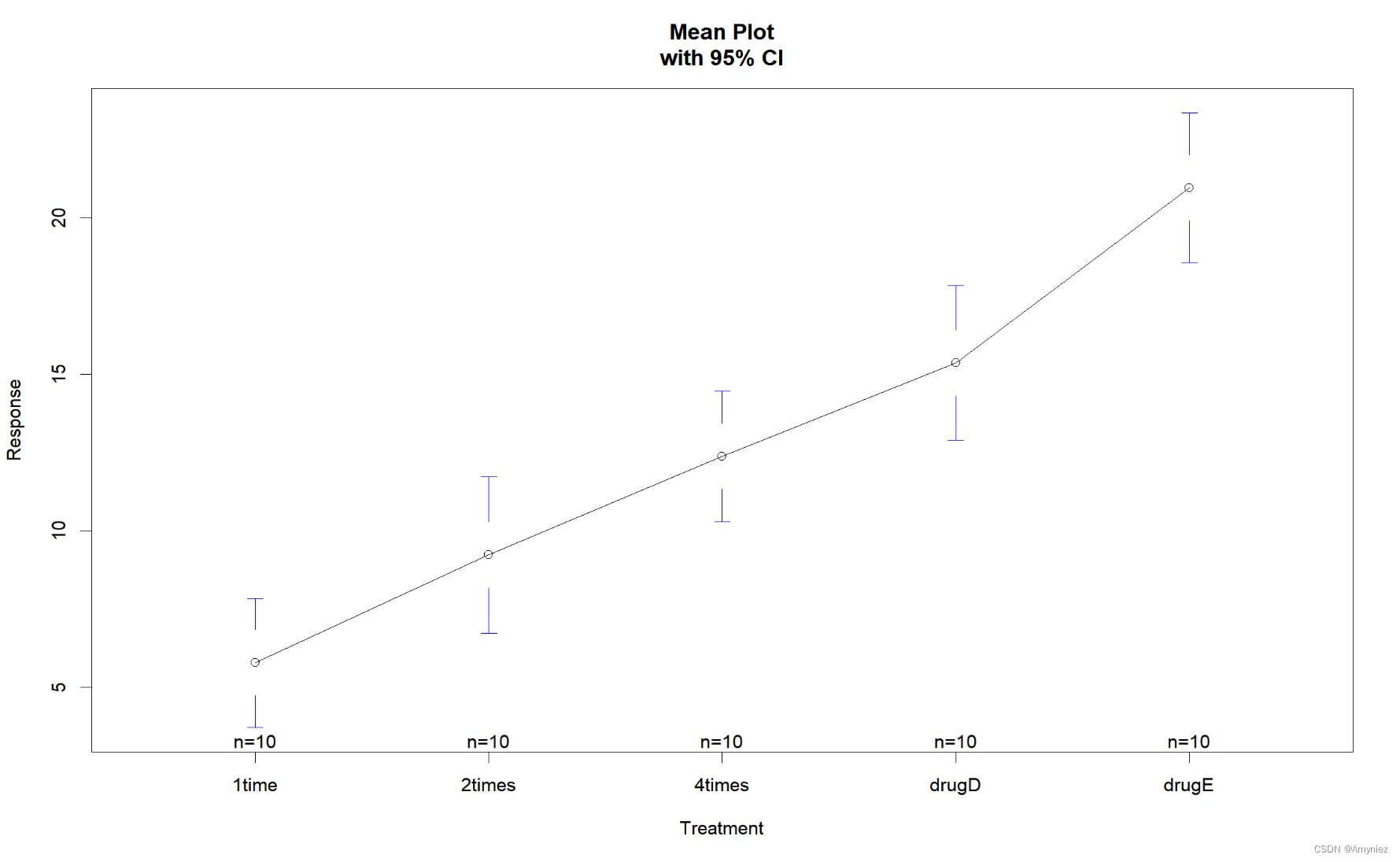
> aggregate(response, by=list(trt), FUN=mean)
Group.1 x
1 1time 5.78197
2 2times 9.22497
3 4times 12.37478
4 drugD 15.36117
5 drugE 20.94752
> aggregate(response, by=list(trt), FUN=sd)
Group.1 x
1 1time 2.878113
2 2times 3.483054
3 4times 2.923119
4 drugD 3.454636
5 drugE 3.345003
> summary(fit)
Df Sum Sq Mean Sq F value Pr(>F)
trt 4 1351.4 337.8 32.43 9.82e-13 ***
Residuals 45 468.8 10.4
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
ANOVA对治疗方式(trt)的F检验非常显著(p<0.0001),说明五种疗法的效果不同。
2.1 绘制各组均值及其置信区间的图形
## 绘制各组均值及其置信区间的图形
library(gplots)
plotmeans(response ~ trt, xlab="Treatment", ylab="Response",
main="Mean Plot\nwith 95% CI")
detach(cholesterol) # 关闭连接
自由度: 考虑样本量为n的样本(n个数),如果我们知道样本均值,那么我们只需要知道前n-1个数,就可以得知全部信息,因为最后一个数可以借助均值计算得出。所以,在给定样本均值(约束条件)的情况下,我们相当于在原本n个数的基础上消减了一个自由度,样本提供的信息只有n-1个独立的部分。
2.2 多重比较-TukeyHSD
虽然ANOVA对各疗法的F检验表明五种药物疗法效果不同,但是并没有告诉你哪种疗法与其他疗法不同。多重比较可以解决这个问题,其中TukeyHSD()函数提供了对各组均值差异的成对检验。
TukeyHSD(fit)
结果:
Tukey multiple comparisons of means
95% family-wise confidence level
Fit: aov(formula = response ~ trt)
$trt
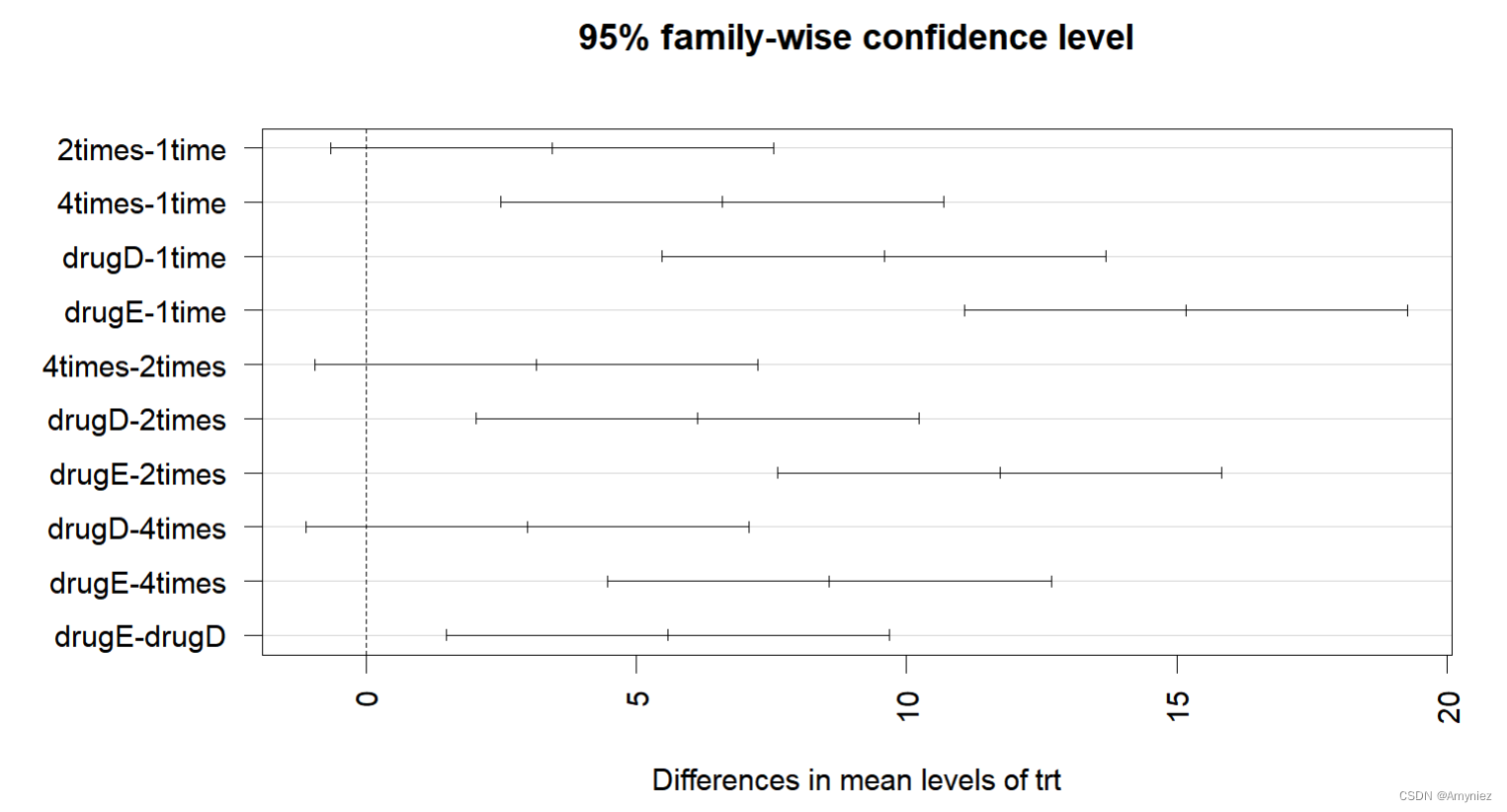
diff lwr upr p adj
2times-1time 3.44300 -0.6582817 7.544282 0.1380949
4times-1time 6.59281 2.4915283 10.694092 0.0003542
drugD-1time 9.57920 5.4779183 13.680482 0.0000003
drugE-1time 15.16555 11.0642683 19.266832 0.0000000
4times-2times 3.14981 -0.9514717 7.251092 0.2050382
drugD-2times 6.13620 2.0349183 10.237482 0.0009611
drugE-2times 11.72255 7.6212683 15.823832 0.0000000
drugD-4times 2.98639 -1.1148917 7.087672 0.2512446
drugE-4times 8.57274 4.4714583 12.674022 0.0000037
drugE-drugD 5.58635 1.4850683 9.687632 0.0030633
可以看到,1time和2times的均值差异不显著(p=0.138),而1time和4times间的差异非常显著(p<0.001)。
2.2.1 成对比较图
第一个par语句用来旋转轴标签,第二个用来增大左边界的面积,可使标签摆放更美观。图形中置信区间包含0的疗法说明差异不显著(p>0.5)
在R语言中,par(las=2)是用来设置绘图区域的标签方向的。其中,las是一个整数,用于指定标签的方向。las=0表示标签平行于坐标轴,las=1表示标签垂直于坐标轴,las=2表示标签垂直于坐标轴并且文字是从右向左书写的。
par(las=2)
par(mar=c(5,8,4,2))
plot(TukeyHSD(fit))
结果展示:
2.2.2 多重均值比较-TukeyHSD(更易理解)
multcomp包中的glht()函数提供了多重均值比较更为全面的方法,既适用于线性模型),也适用于广义线性模型。下面的代码重现了Tukey HSD检验,并用一个不同的图形对结果进行展示。
library(multcomp)
par(las=0)
par(mar=c(5,4,6,2))
tuk <- glht(fit, linfct=mcp(trt="Tukey"))
plot(cld(tuk, level=.05),col="lightgrey") #cld()函数中的level选项设置了使用的显著水平0.05
结果展示:
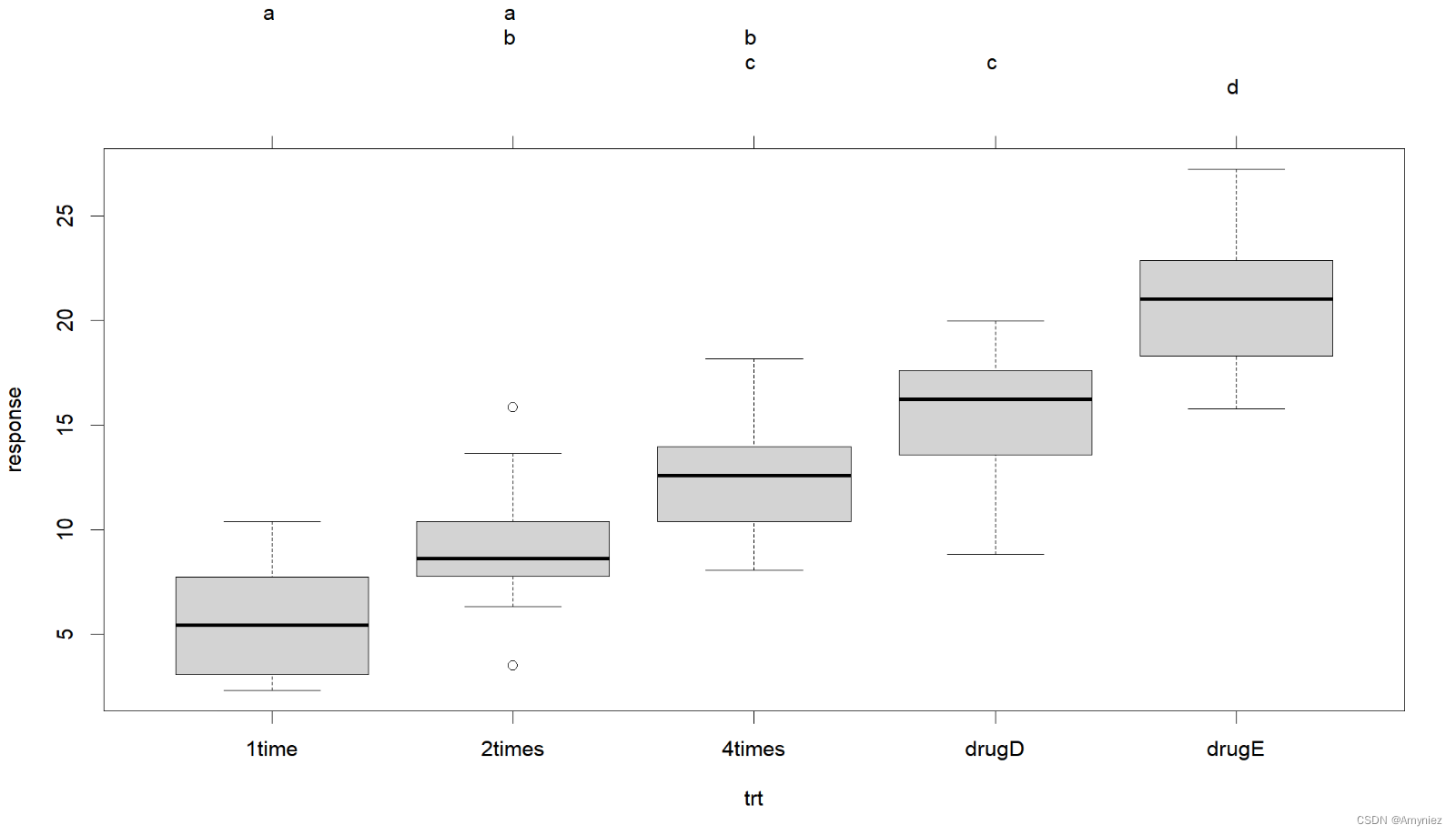
图像解释:
有相同字母的组(用箱线图表示)说明均值差异不显著。可以看到,1time和2times差异不显著(有相同的字母a),2times和4times差异也不显著(有相同的字母b),而1time和4times差异显著(它们没有共同的字母)。个人认为,该图比2.2.1中的成对比较图更易理解。
2.3 评估检验的假设条件
2.3.1 正态性检验
我们对于结果的可靠性取决于做统计检验时数据满足假设条件的程度。单因素方差分析中,我们假设因变量服从正态分布,各组方差相等。可以使用Q-Q图来检验正态性假设:
library(car)
# qqPlot()要求用lm()拟合
qqPlot(lm(response ~ trt, data=cholesterol),
simulate=TRUE, main="Q-Q Plot", labels=FALSE)
Q-Q图:
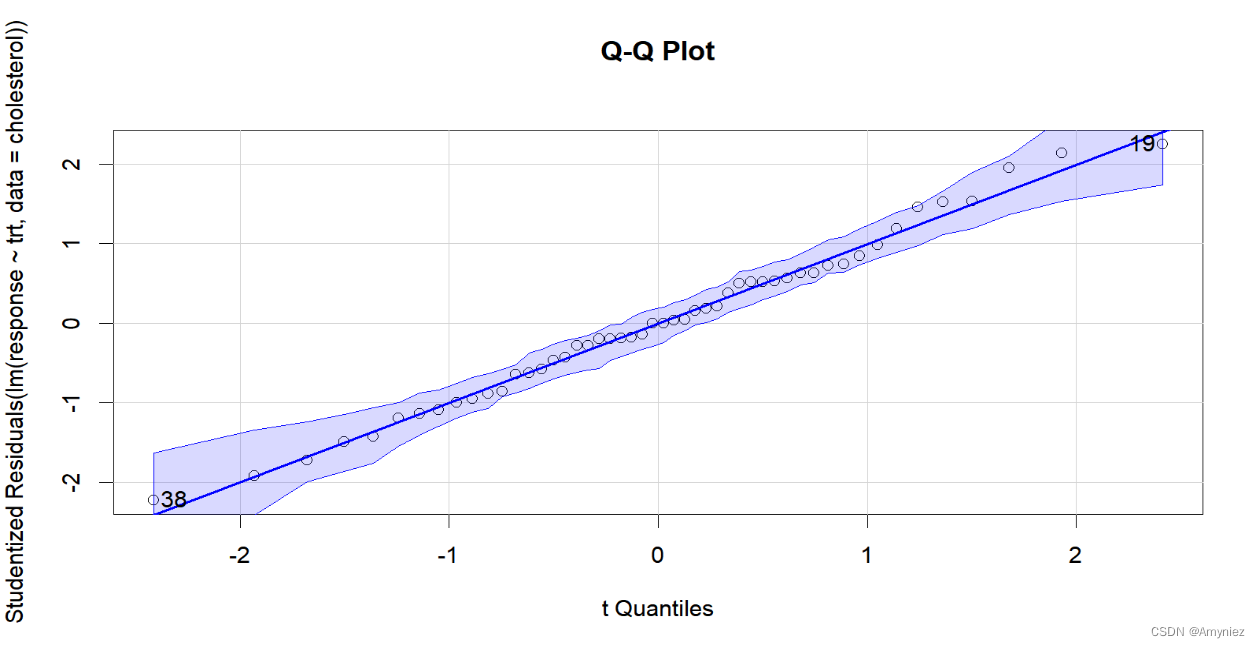
数据落在95%的置信区间范围内,说明满足正态性假设。
2.3.2 方差齐性检验
library(car)
qqPlot(lm(response ~ trt, data=cholesterol),
simulate=TRUE, main="Q-Q Plot", labels=FALSE)
结果展示:
Bartlett test of homogeneity of variances
data: response by trt
Bartlett's K-squared = 0.57975, df = 4, p-value = 0.9653
还有其他检验如Fligner-Killeen检验(fligner.test()函数)和Brown-Forsythe检验(HH包中的hov()函数),它们获得的结果与Bartlett检验相同。
2.3.3 离群点检验
离群点 :预测效果不佳的点,具有加大残差。
根据最大的残差值的显著性来判断是否存在离群点:
- 若不显著 Bonferonni P>0.05 ,表明没有离群点;
- 若显著 Bonferonni P<0.05 ,表明该最大残差值点为离群点,需要删去,然后对删除该点后的拟合模型再次进行离群点的检验。
方差齐性分析对离群点非常敏感。可利用car包中的outlierTest()函数来检测离群点:
library(car)
outlierTest(fit)
结果展示:
No Studentized residuals with Bonferroni p < 0.05
Largest |rstudent|:
rstudent unadjusted p-value Bonferroni p
19 2.251149 0.029422 NA
结果分析:
这个结果是关于特定回归分析的统计学测试的输出内容。对于第19个数据点,该测试计算出了学生化残差值为2.251,对应的未经校正的p值为0.029422。然而,经过Bonferroni校正后的p值小于0.05的学生化残差值没有被发现,因此输出显示了“无Bonferroni p < 0.05的学生化残差”。由此可以得出结论,此数据点的学生化残差值并不显著,即不具有统计学上的显著性 (不具有统计学上的显著性意味着,结果不足以证明假设成立。在这种情况下,我们不能拒绝原假设。例子中,没有学生化残差的Bonferroni p值小于0.05,这意味着没有离群点)。
从输出结果来看,并没有证据说明胆固醇数据中含有离群点(当p>1时将产生NA)。因此根据Q-Q图、Bartlett检验和离群点检验,该数据似乎可以用ANOVA模型拟合得很好。这些方法反过来增强了我们对于所得结果的可靠性。
3. 单因素协方差分析
单因素协方差分析(ANCOVA)扩展了单因素方差分析(ANOVA),包含一个或多个定量的协变量。下面的例子来自于multcomp包中的litter数据集。怀孕小鼠被分为四个小组,每个小组接受不同剂量(0、5、50或500)的药物处理。产下幼崽的体重均值为因变量,怀孕时间为协变量。
#单因素协方差分析
data(litter, package="multcomp")
attach(litter)
table(dose)
aggregate(weight, by=list(dose), FUN=mean)
fit <- aov(weight ~ gesttime + dose) # 注意aov()使用的是类型1
summary(fit)
结果展示:
> table(dose)
dose
0 5 50 500
20 19 18 17
> aggregate(weight, by=list(dose), FUN=mean)
Group.1 x
1 0 32.30850
2 5 29.30842
3 50 29.86611
4 500 29.64647
> summary(fit)
Df Sum Sq Mean Sq F value Pr(>F)
gesttime 1 134.3 134.30 8.049 0.00597 **
dose 3 137.1 45.71 2.739 0.04988 *
Residuals 69 1151.3 16.69
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
结果分析:
利用table()函数,可以看到每种剂量下所产的幼崽数并不相同:0剂量时(未用药)产崽20个,500剂量时产崽17个。再用aggregate()函数获得各组均值,可以发现未用药组幼崽体重均值最高(32.3)。ANCOVA的F检验表明:
(a) 怀孕时间与幼崽出生体重相关;
(b) 控制怀孕时间,药物剂量与出生体重相关。
控制怀孕时间,确实发现每种药物剂量下幼崽出生体重均值不同。
3.1 去除协变量的影响
由于使用了协变量,你可能想要获取调整的组均值,即去除协变量效应后的组均值。可使用 effects包中的effects()函数 来计算调整的均值:
library(effects)
effect("dose", fit)
结果:
dose effect
dose
0 5 50 500
32.35367 28.87672 30.56614 29.33460
调整的均值与aggregate()函数得出的未调整的均值类似,但并非所有的情况都是如此。 总之,effects包为复杂的研究设计提供了强大的计算调整均值的方法,并能将结果可视化。
3.2 用户定义对照的多重比较(User-defined Contrasts)
剂量的F检验虽然表明了不同的处理方式幼崽的体重均值不同,但无法告知我们哪种处理方式与其他方式不同。同样,我们使用multcomp包来对所有均值进行成对比较。另外,multcomp包还可以用来检验用户自定义的均值假设。假定你对未用药条件与其他三种用药条件影响是否不同感兴趣,可使用以下代码实现:
library(multcomp)
contrast <- rbind("no drug vs. drug" = c(3, -1, -1, -1)) #rbind表示“row bind”,即“按行绑定”,其中每个对象应该具有相同的列数和相同的类型
summary(glht(fit, linfct=mcp(dose=contrast)))
rbind(“no drug vs. drug” = c(3, -1, -1, -1))中的c(3, -1, -1, -1)是一个长度为4的数值向量,其中第一个数值(即3)表示“未注射药物的比重”,后面三个数值(即-1,-1,-1)表示“三种剂量的比重”。这个向量用于定义将三个剂量合并为一个组并将其与未注射药物组进行比较的比较向量。
结果:
Simultaneous Tests for General Linear Hypotheses
Multiple Comparisons of Means: User-defined Contrasts
Fit: aov(formula = weight ~ gesttime + dose)
Linear Hypotheses:
Estimate Std. Error t value Pr(>|t|)
no drug vs. drug == 0 8.284 3.209 2.581 0.012
no drug vs. drug == 0 *
---
Signif. codes:
0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
(Adjusted p values reported -- single-step method)
对照c(3, -1, -1, -1)设定第一组和其他三组的均值进行比较。假设检验的t统计量(2.581)在p<0.05水平下显著,因此,可以得出未用药组比其他用药条件下的出生体重高的结论。
3.3 评估检验的假设条件
ANCOVA与ANOVA相同,都需要正态性和同方差性假设,另外ANCOVA还假定回归斜率相同。本例中,假定四个处理组通过怀孕时间来预测出生体重的回归斜率都相同。ANCOVA模型包含怀孕时间×剂量的交互项时,可对回归斜率的同质性进行检验。交互效应若显著,则意味着时间和幼崽出生体重间的关系依赖于药物剂量的水平。
library(multcomp)
fit2 <- aov(weight ~ gesttime*dose, data=litter)
summary(fit2)
结果:
Df Sum Sq Mean Sq F value Pr(>F)
gesttime 1 134.3 134.30 8.289 0.00537 **
dose 3 137.1 45.71 2.821 0.04556 *
gesttime:dose 3 81.9 27.29 1.684 0.17889
Residuals 66 1069.4 16.20
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
可以看到交互效应不显著,支持了斜率相等的假设。若假设不成立,可以尝试变换协变量或因变量,或使用能对每个斜率独立解释的模型,或使用不需要假设回归斜率同质性的非参数ANCOVA方法。sm包中sm.ancova()函数为后者提供了一个例子。
总之,如果交互效应不显著,就说明自变量对因变量的影响是线性的,可以使用统一的回归方程来描述它们之间的关系。
3.4 结果可视化
HH包中的ancova()函数可以绘制因变量、协变量和因子之间的关系图:
library(HH)
ancova(weight ~ gesttime + dose, data=litter)
结果:
Analysis of Variance Table
Response: weight
Df Sum Sq Mean Sq F value Pr(>F)
gesttime 1 134.30 134.304 8.0493 0.005971 **
dose 3 137.12 45.708 2.7394 0.049883 *
Residuals 69 1151.27 16.685
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
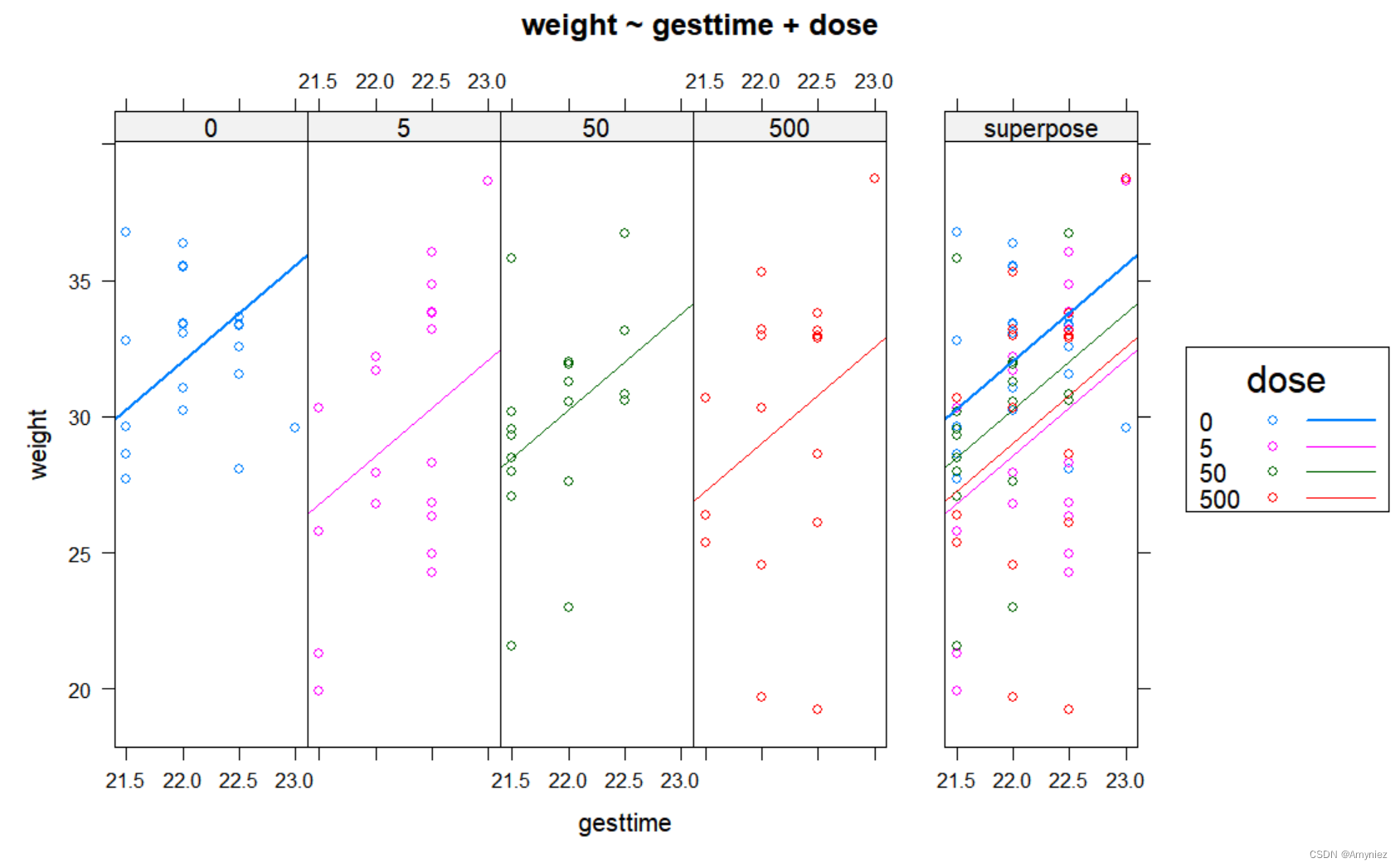
从图中可以看到,用怀孕时间来预测出生体重的回归线相互平行,只是截距项不同。随着怀孕时间增加,幼崽出生体重也会增加。另外,还可以看到0剂量组截距项最大,5剂量组截距项最小。由于你上面的设置,直线会保持平行,若使用ancova(weight ~ gesttime*dose),生成的图形将允许斜率和截距项依据组别而发生变化,这对可视化那些违背回归斜率同质性的实例非常有用。
4. 双因素方差分析(ANOVA)
在双因素方差分析中,受试者被分配到两因子的交叉类别组中。 以基础安装中的ToothGrowth数据集为例,随机分配60只豚鼠,分别采用两种喂食方法(橙汁或维生素C),各喂食方法中抗坏血酸含量有三种水平(0.5mg、1mg或2mg),每种处理方式组合都被分配10只豚鼠。牙齿长度为因变量。
#双因素方差分析
attach(ToothGrowth)
table(supp, dose)
head(ToothGrowth)
aggregate(len, by=list(supp, dose), FUN=mean)
aggregate(len, by=list(supp, dose), FUN=sd)
dose <- factor(dose)
fit <- aov(len ~ supp*dose) # 双因素影响
summary(fit)
detach(ToothGrowth)
结果:
> head(ToothGrowth)
len supp dose
1 4.2 VC 0.5
2 11.5 VC 0.5
3 7.3 VC 0.5
4 5.8 VC 0.5
5 6.4 VC 0.5
6 10.0 VC 0.5
> aggregate(len, by=list(supp, dose), FUN=mean)
Group.1 Group.2 x
1 OJ 0.5 13.23
2 VC 0.5 7.98
3 OJ 1.0 22.70
4 VC 1.0 16.77
5 OJ 2.0 26.06
6 VC 2.0 26.14
> aggregate(len, by=list(supp, dose), FUN=sd)
Group.1 Group.2 x
1 OJ 0.5 4.459709
2 VC 0.5 2.746634
3 OJ 1.0 3.910953
4 VC 1.0 2.515309
5 OJ 2.0 2.655058
6 VC 2.0 4.797731
> summary(fit)
Df Sum Sq Mean Sq F value Pr(>F)
supp 1 205.4 205.4 15.572 0.000231 ***
dose 2 2426.4 1213.2 92.000 < 2e-16 ***
supp:dose 2 108.3 54.2 4.107 0.021860 *
Residuals 54 712.1 13.2
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
结果分析:
table语句的预处理表明该设计是均衡设计(各设计单元中样本大小都相同),aggregate语句处理可获得各单元的均值和标准差。dose变量被转换为因子变量,这样aov()函数就会将它当做一个分组变量,而不是一个数值型协变量。 用summary()函数得到方差分析表,可以看到主效应(supp和dose)和交互效应都非常显著。
4.1 结果可视化
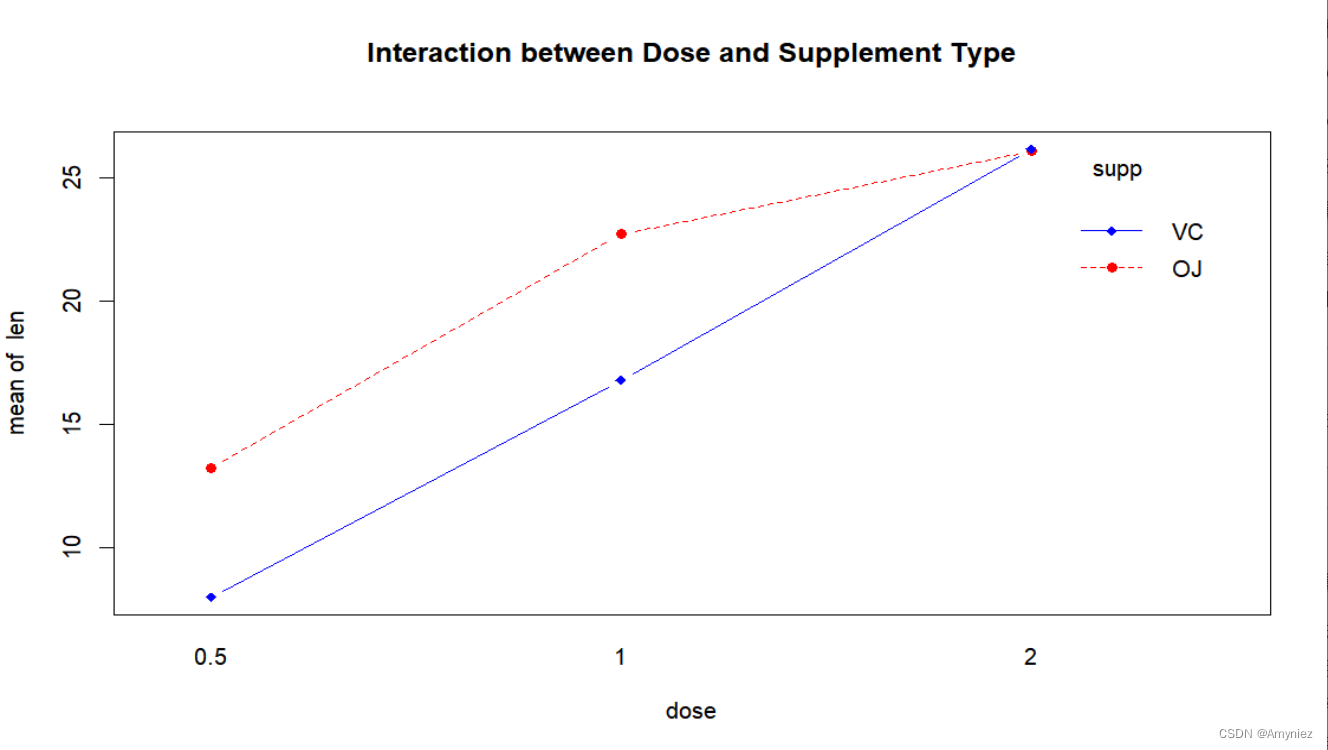
此处可用interaction.plot()函数来展示双因素方差分析的交互效应。
interaction.plot(dose, supp, len, type="b",
col=c("red","blue"), pch=c(16, 18),
main = "Interaction between Dose and Supplement Type")
图形展示了各种剂量喂食下豚鼠牙齿长度的均值:
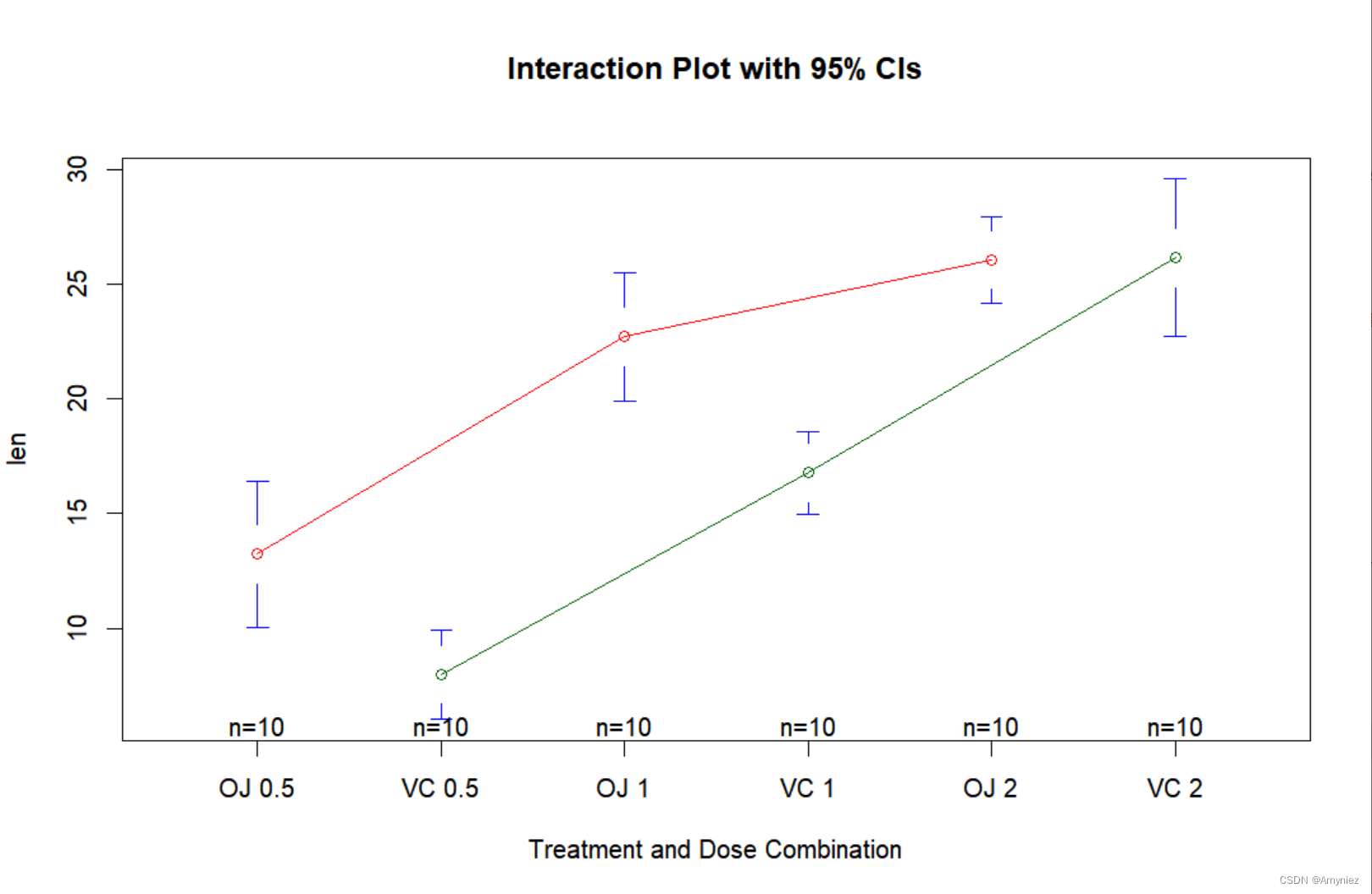
用gplots包中的plotmeans()函数来展示交互效应:
library(gplots)
plotmeans(len ~ interaction(supp, dose, sep=" "),
connect=list(c(1,3,5),c(2,4,6)),
col=c("red", "darkgreen"),
main = "Interaction Plot with 95% CIs",
xlab="Treatment and Dose Combination")
图形展示了均值、误差棒(95%的置信区间)和样本大小:
用HH包中的interaction2wt()函数来可视化结果,图形对任意顺序的因子设计的主效应和交互效应都会进行展示:
library(HH)
interaction2wt(len~supp*dose)
ToothGrowth数据集的主效应和交互效应:
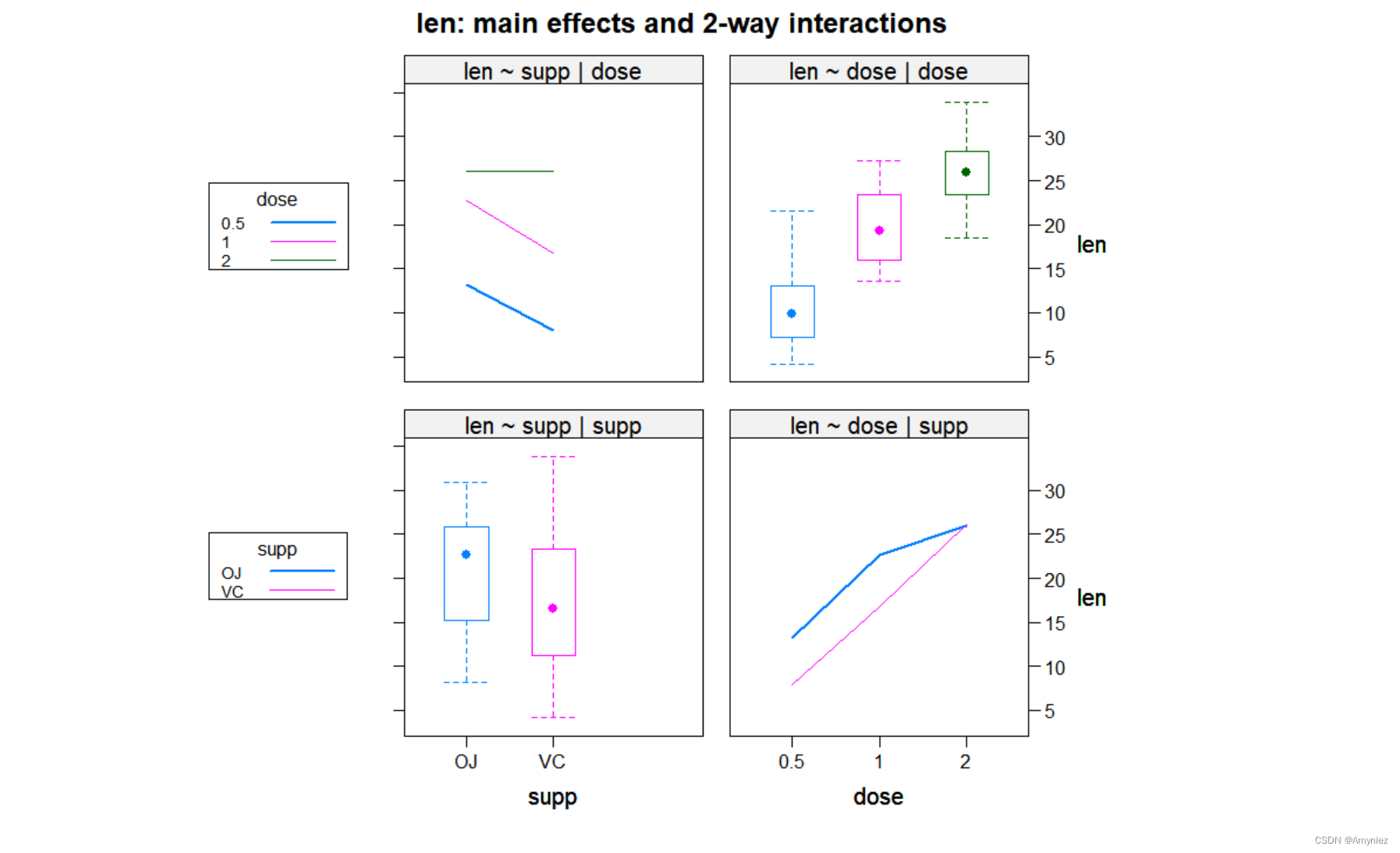
以上三幅图形都表明随着橙汁(QJ)和维生素C(VC)中的抗坏血酸剂量的增加,牙齿长度变长。对于0.5mg和1mg剂量,橙汁比维生素C更能促进牙齿生长;对于2mg剂量的抗坏血酸,两种喂食方法下牙齿长度增长相同。三种绘图方法中,我更推荐HH包中的interaction2wt()函数,因为它能展示任意复杂度设计(双因素方差分析、三因素方差分析等)的 主效应(箱线图) 和交互效应。
此处没有涵盖模型假设检验和均值比较的内容,因为它们只是之前方法的一个自然扩展而已。此外,该设计是均衡的,故而不用担心效应顺序的影响。