
导读
为了发挥清华大学多学科优势,搭建跨学科交叉融合平台,创新跨学科交叉培养模式,培养具有大数据思维和应用创新的“π”型人才,由清华大学研究生院、清华大学大数据研究中心及相关院系共同设计组织的“清华大学大数据能力提升项目”开始实施并深受校内师生的认可。项目通过整合建设课程模块,形成了大数据思维与技能、跨界学习、实操应用相结合的大数据课程体系和线上线下混合式教学模式,显著提升了学生大数据分析能力和创新应用能力。
回首2022年,清华大学大数据能力提升项目取得了丰硕的成果,同学们将课程中学到的数据思维和技能成功地应用在本专业的学习和科研中,在看到数据科学魅力的同时,也将自己打造成为了交叉复合型的创新型人才。下面让我们通过来自8个院系的10位同学代表一起领略他们的风采吧!
多基因疾病风险预测:利用遗传大数据辅助精准医疗
人类复杂疾病的全基因组关联研究(GWAS)已经发现了数以万计的相关遗传变异,提供了关于疾病机制的新见解,并揭示了广泛的多基因遗传结构。在GWAS发现的临床转化中,我们利用基因组信息,对人类复杂疾病进行预测,进而帮助更好的疾病预防和治疗,推进精准医疗的发展。目前我们已针对此研究问题发表三篇相关的SCI论文,并开发相应的计算软件。
由于遗传数据样本量和维数均非常庞大(数以百万),利用传统的统计方法不能很有效的结局,同时考虑到不同位点之间复杂的相关结构,需要利用高维数据分析的技术来进行建模。
我们首先利用经验贝叶斯统计方法,提出利用正态混合先验,基于单核苷酸多态性在全基因组关联分析中的效应的分布对先验参数进行估计,并最小化贝叶斯预测风险。与传统方法P+T相比,我们的方法将哮喘、乳腺癌、乳糜泻、克罗恩病、帕金森病和二型糖尿病六种人类复杂疾病的遗传准确性提升307.1%、42.8%、25.5%、3.1%、74.3%和49.6%,论文Leveraging effect size distributions to improve polygenic risk scores derived from summary statistics of genome-wide association studies发表在SCI期刊PLoS Computational Biology。
在此基础上,我们借助转录组信息进一步提升预测准确性,以基因表达作为中介,建立从基因型-基因表达-表型的预测关系,论文A novel transcriptional risk score for risk prediction of complex human diseases发表在SCI期刊Genetic Epidemiology。
同时,我们考虑到不同的贝叶斯先验信息对于不同的遗传结构的具有特定的优越性。例如,精神类疾病的遗传结构通常由多基因组成,适合连续型先验分布;而免疫类疾病的遗传结构更加稀疏,适合离散型先验分布。参考神经网络的想法构思,我们创新性地利用“神经先验”构建了一个统一的贝叶斯回归多基因风险评分框架,使其能够适应不同的遗传结构。这种构造将传统统计贝叶斯模型、神经网络、以及统计遗传学的思想结合在一起,通过统一的框架和高效的MCMC计算,大幅提升了复杂一并预测的准确性。“神经先验”涵盖了贝叶斯线性回归中常用的各种类型的稀疏和收缩先验,如连续和离散的spike-and-slab先验、拉普拉斯先验、柯西先验、马蹄形先验等。我们的方法在广泛的贝叶斯先验分布中进行搜索,无论是共轭还是非共轭,离散混合还是连续分级,重尾或是轻尾,均可以覆盖到(表1,图1)。
表1:三种神经先验构造


图1:用于构建多基因风险评分的先验分布形式
我们的模型构建精确到每条染色体,即允许不同染色体具有不同的遗传结构(图2)。我们还考虑到数据的隐私型等问题,对个体层级的数据进行处理,利用汇总统计量建模。这种灵活的算法可以根据不同疾病的遗传结构自动选取广泛的先验选择来提高复杂疾病的整体预测准确性,并且具有很高的计算效率。

图2:不同疾病在染色体层级上对先验的选择
为了评估NeuPred和其他基于汇总统计的PRS方法的性能,我们分析了7种WTCCC复杂疾病和8个具有独立测试数据集的大规模GWAS研究。我们比较了12种现有的预测方法,我们的方法在预测准确性上有一致的提升,在独立验证数据集中也表现稳健(图3)。同时,我们的方法不需要对参数进行调试,在外部数据集中具有良好的普适性,论文A data-adaptive Bayesian regression approach for polygenic risk prediction发表在SCI期刊Bioinformatics。
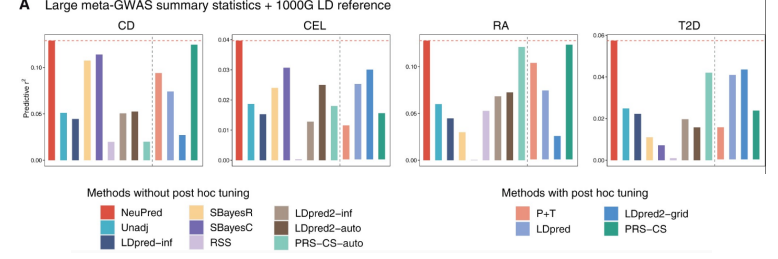
图3:NeuPred与其他常用方法的比较,取得了预测效果的显著提升
编辑:文婧
校对:龚力
