大家好,我是邓飞。
今天介绍一下曼哈顿图如何打印出SNP的名称,类似这样的:

1. 软件包 qqman 下载
在CRAN中下载:
install.packages("qqman")
2. 导出示例数据
# qqman
library(qqman)
data("gwasResults")
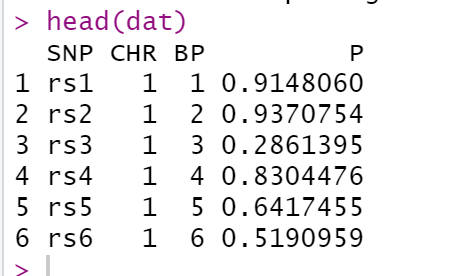
dat = gwasResults
head(dat)
3. 示例曼哈顿图
manhattan(dat)

4. 打印显著性的SNP名称
这里,参数:annotatePval,注意,这里的值,不是-log10转化的,而是原始的p值,比如,这里,我们想打印1e-8的snp名称,默认一个染色体只显示一个snp名称:
manhattan(dat,annotatePval = 1e-8)

如果我们想把所有的符合条件的snp名称都显示出来,增加参数:annotateTop = F
snp如果很多的话,就遮盖了。
manhattan(dat,annotatePval = 1e-7,annotateTop = F)

设置p值为1e-3,看一下效果:
manhattan(dat,annotatePval = 1e-3,annotateTop = F)

5. 指定特定的snp名称,高亮
比如我们选择每条染色体p值最小的snp,首先筛选,这里用tidyverse来处理:
library(tidyverse)
head(dat)
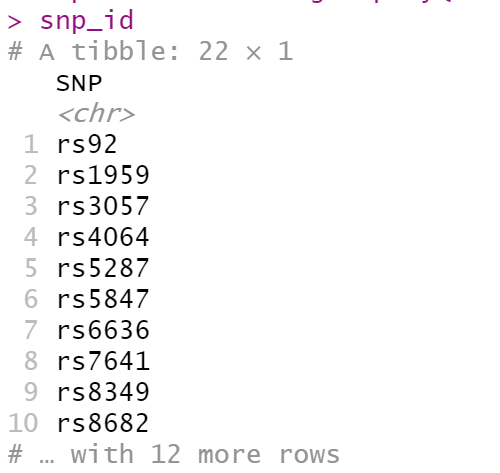
snp_id = dat %>% group_by(CHR) %>% slice_min(P) %>% ungroup %>% select(SNP)
snp_id
manhattan(dat,highlight = snp_id$SNP)
挑选的snp:
将挑选的snp高亮:
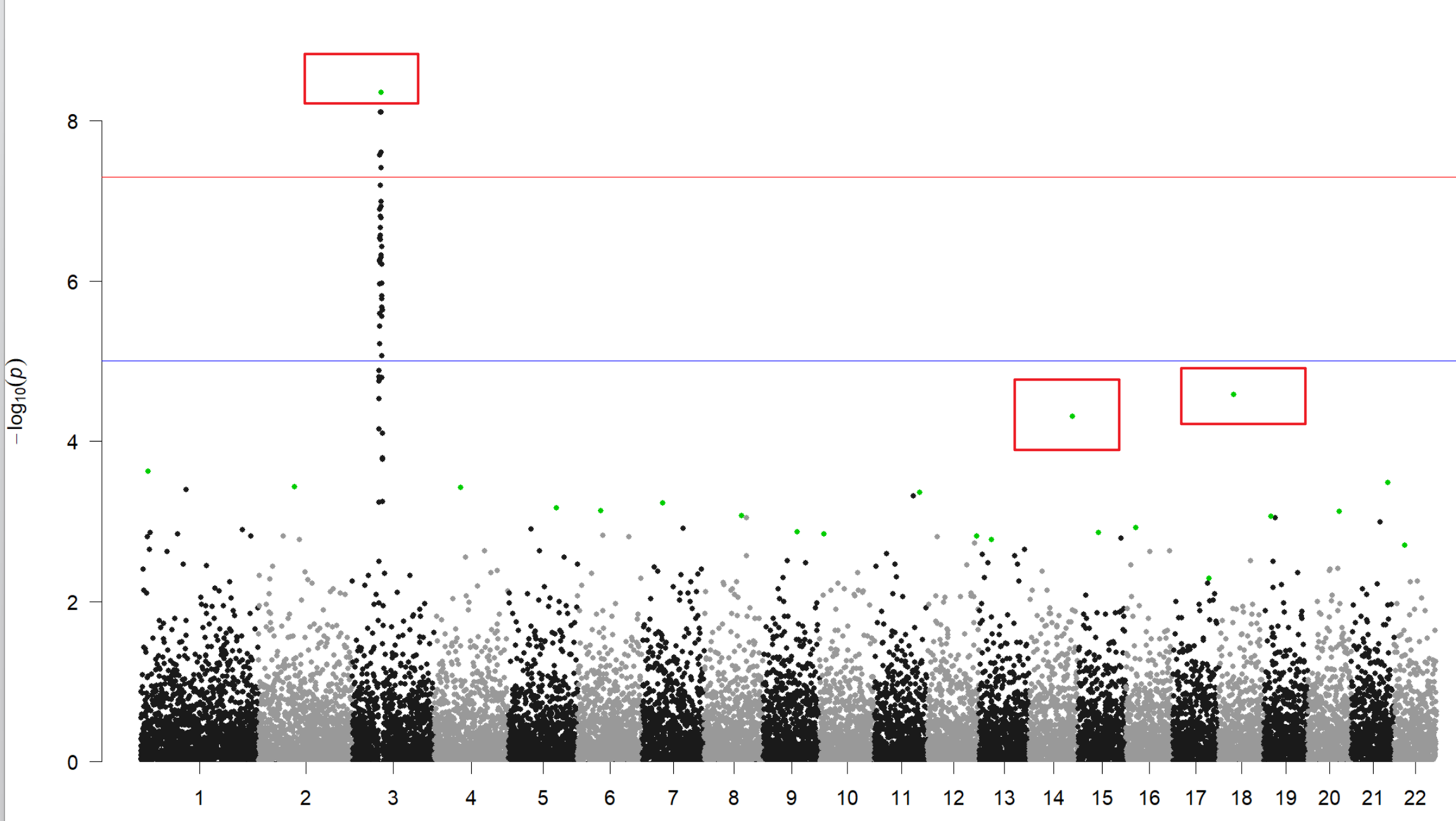
这就算搞定了。
其它需求,可以参考函数的说明文档。