1 功能
sc.pp.normalize_per_cell()和sc.pp.normalize_total()功能是一致的。在最新的Scanpy中sc.pp.normalize_total()替代了sc.pp.normalize_per_cell(),具体的情况见参考文献【2】。这个函数的功能就是对细胞的计数矩阵进行归一化操作。
2 例子
import numpy as np
import scanpy as sc
x = np.array([[1, 2, 3],
[2, 3, 4],
[1, 1, 3],
[0, 2, 10]])
adata = sc.AnnData(x)
adata.raw = adata.copy()
print(np.dot(np.diag(ff), x))
[[1.25 2.5 3.75 ]
[1.66666667 2.5 3.33333333]
[1.5 1.5 4.5 ]
[0. 1.25 6.25 ]]
sc.pp.normalize_per_cell(adata)
print(adata.X)
[[1.25 2.5 3.75 ]
[1.66666667 2.5 3.33333333]
[1.5 1.5 4.5 ]
[0. 1.25 6.25 ]]
sc.pp.normalize_total(adata)
print(adata.X)
[[1.25 2.5 3.75 ]
[1.66666667 2.5 3.33333333]
[1.5 1.5 4.5 ]
[0. 1.25 6.25 ]]
# 可以看到两个函数的输出是相同的
sc.pp.log1p(adata)
print(adata.X)
[[0.8109302 1.2527629 1.5581446 ]
[0.98082924 1.2527629 1.466337 ]
[0.91629076 0.91629076 1.704748 ]
[0. 0.8109302 1.9810015 ]]
# 最终的计算如下公式所示
接下来看具体的归一化是如何计算的
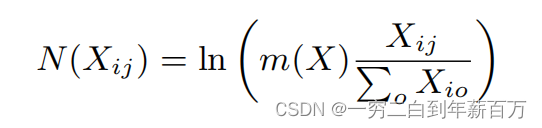
上述公式中 X X X表示计数矩阵, i j ij ij分别表示行列, m m m表示计数中值, O O O也表示列。下面介绍具体的Python实现代码:
# 每个细胞所有基因的计数值的和
np.sum(x, axis=1)
# 每个细胞的计数中值
np.median(np.sum(x, axis=1))
# 上面公式的计算部分
ff = np.median(np.sum(x, axis=1)) / np.sum(x, axis=1)
np.dot(np.diag(ff), x)
# 取log
sc.pp.log1p(adata)
3 参考文献
[1]sc.pp.normalize_per_cell和sc.pp.normalize_total()
[2]scanpy.pp.normalize_total