1,开局一张图
下面的Nature上发表的一张图:https://www.nature.com/news/2008/081105/pdf/456018a.pdf

消失的遗传力,就像悬疑片中消失的子弹一样,科学家就是侦探,通过各种工具(生物统计、生物信息、数量遗传、基因测序)来找到消失的遗传力到底去哪里了。
所以,科学家的形象是这样的:

如果我们找到了消失的遗传力,弄明白它消失的原因以及找到它的方法,报酬是大大滴:

2,丢失的遗传力
“遗传力丢失”( Missing heritability)是一个重要的数量遗传学问题,即通过遗传标记估计的遗传力以及通过全基因组关联分析(GWAS)发现的所有相关基因所贡献的遗传力总和均低于实际的遗传力。
举个栗子:
比如,如果身高的遗传力是0.8,但是GWAS分析中显著性的SNP只解释了45%的变异(遗传力为0.45),这样还是有0.35的遗传力丢失了,这就叫做丢失的遗传力。
3,消失遗传力的进一步剖分
参考:
Witte JS, Visscher PM, Wray NR. The contribution of genetic variants to disease depends on the ruler. Nat Rev Genet. 2014 Nov;15(11):765-76. doi: 10.1038/nrg3786. Epub 2014 Sep 16. PMID: 25223781; PMCID: PMC4412738.
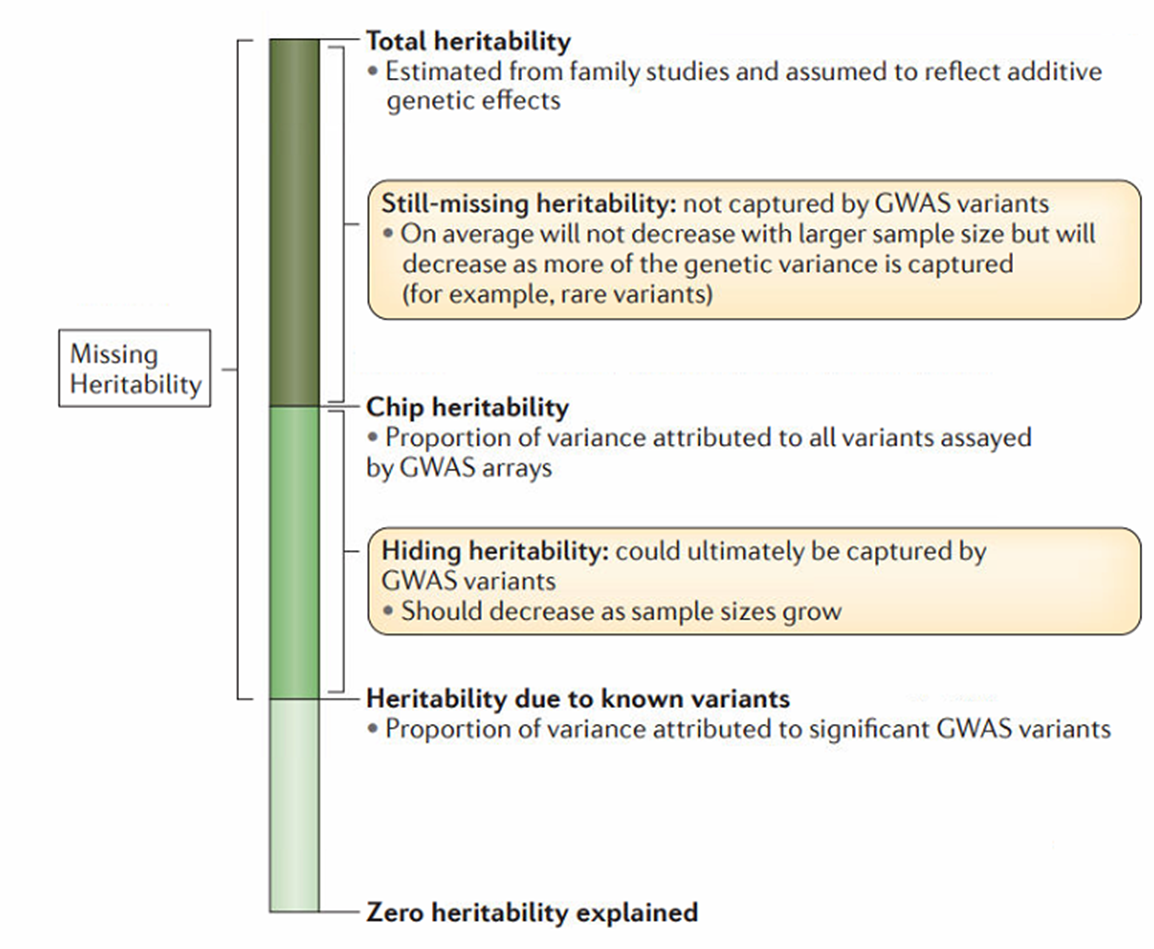
遗传力细分:
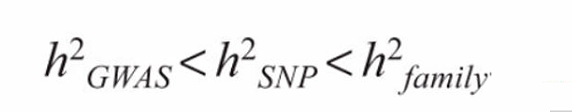
-
Total heritability,是实际的遗传力,主要是从家系群体(比如同卵上胞胎、全同胞)估计的遗传力,对应的是family遗传力
-
Chip heritability,是通过REML估计的遗传力,使用的是全部SNP数据,比如GCTA、asreml中的GBLUP,估计的遗传力都属于此,对应的是SNP遗传力
-
Heritability due to known variants,是GWAS分析中显著的SNP估计的遗传力,对应的是PVE(解释百分比),对应的是GWAS遗传力
一般我们认为消失的遗传力为:
h f a m i l y 2 − h G W A S 2 h^2_{family} - h^2_{GWAS} hfamily2−hGWAS2
但是它还可以进一步剖分为两部分:
-
第一部分:Still-missing heritability,是依旧消失的遗传力,它的计算公式是 h f a m i l y 2 − h S N P 2 h^2_{family} - h^2_{SNP} hfamily2−hSNP2,它的来源是GWAS无法捕捉到的变异,比如CNV,SV等,以通过增加变异来源(CNV,SV,PVA),稀有变异来解决。(泛基因组的应用区间)
-
第二部分:Hiding heritability,是隐藏的遗传力,它的计算公式是 h S N P 2 − h G W A S 2 h^2_{SNP} - h^2_{GWAS} hSNP2−hGWAS2,它主要是由于目前的统计方法导致一些效应小的位点没有检测出来(假阴性),可以通过样本量来降低这部分的值。
4, 如何利用消失的遗传力
育种中,遗传力的高低直接关系着如何应用以及选择的好坏。
比如遗传力0.9的百粒重,不用分子标记,不用系谱,不用GS,直接表型选择就行了,这才是真正的:
老子英雄儿好汉……
中等遗传力,就需要借助半同胞、全同胞,借助家系、系谱进行选择了
低遗传力,就需要借助MAS和GS进行选择了
而如果一个性状的遗传力,降低了消失的遗传力,是的可以操作的部分增加了,那么就会提升估计的准确性,加大了选择相应,增加了选择的效率。
因此,提升育种选择的效率,可以从两个方面:
第一个方面,降低“依旧消失的遗传力”,具体表现为寻找多种变异来源,比如泛基因组、图泛基因组找到更准更多的变异(SV,Indel),这种方法即可以用在MAS,也可以用在GS上。
第二个方面,降低“隐藏的遗传力”,具体表现就是增大样本量,GS中扩大参考群的数量,GWAS中增加样本的数量,增大SNP变异位点数等。
三体中一个观点是“锁死科技”,他认为科技的发展来源于对微观物质的掌握和控制,锁死人类的量子物理,就不能进行进一步的基因编辑、原子能利用、星际飞船、人脑计算机等领域。
同样的道理,对于遗传力,我们把消失的遗传力进一步剖分,找到对应的解决方案(比如增加变异来源,增大样本量),就有了方向,针对性的提升和解决。
这才是科学的解决问题的思路!