内容导览
1. 隐秘而强大的植物内生菌
2. 难以区分的植物内生菌
3. 更好的植物内生细菌测序方法
3.1 LNA-16S测序鉴定内生细菌原理
3.2 LNA-16S测序鉴定内生细菌占比高达99%
4. 植物内生菌研究思路和方法
4.1 代表性案例精解
4.2 研究思路和方法小结
1. 隐秘而强大的植物内生菌
在植物的生长历程中,会与许多不同种类的微生物邂逅相互作用,这或许就是植物能在陆地上定植的原因。植物叶绿体和线粒体利用核编码蛋白复制其DNA,与那些参与核基因组复制的蛋白质不同,这些蛋白质是专门为在细胞器环境中进行复制而构建的。这些细胞器定位的蛋白质起源于细菌和噬菌体基因,这支持了它们起源的内共生理论并建立为内共生体。因此,这一类几乎不为人知的微生物,默默在植物体内存在着发挥着自己独特的作用,它们被称为内生菌(Endophyte)。
内生菌被定义为在某一阶段或者全部阶段都生活在植物内部,又不形成明显侵染的微生物,包含内生细菌、内生真菌和内生放线菌。它们的名气正如它们的存在一样默默无闻,直到后来人们从植物内部分离出了它们,才知道原来植物体内还有微生物。几乎所有植物中都有内生菌的存在,许多植物离了内生菌甚至不能存活(图1)[1]。由于内生菌长期生活在植物体的特殊和环境里,并和宿主协同进化,在演变过程中二者形成了互利共生关系,一方面植物为内生菌提供光合产物和矿物质供内生菌生长需要,另一方面内生菌在宿主的生长发育和系统演化过程中起重要作用。
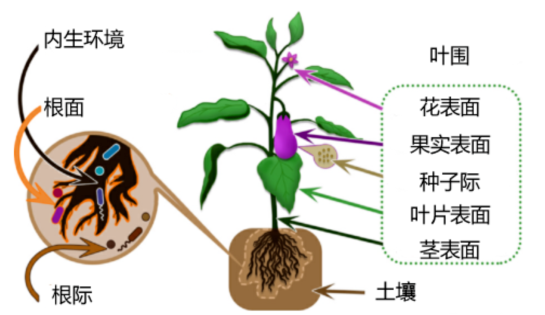
图1.微生物在植物的各个部位上定植和生长
内生菌产生的部分代谢物能刺激植物的生长发育,提高宿主植物对生物胁迫或非生物胁迫的抵抗能力。因为病原微生物和内生细菌在植物内占据相同的生态位,所以还可以接种内生细菌来控制植物体内的病原体。此外,内生真菌对植物生长还有着很多其它益处:固氮、溶磷、产生植物激素,改变根系形态、调节渗透、提高磷酸铁载体产量、增强溶解活性和调节气孔等作用(图2)[1]。
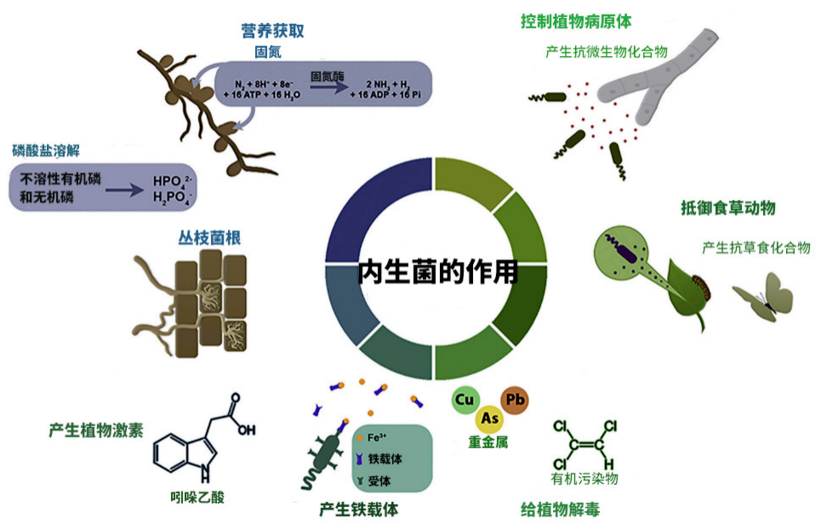
图2. 植物内生菌的作用
各种植物体中存在的内生菌的数量、种类与植物的种类、生存环境、生长阶段、营养供给及两者的基因型有密切关系。生长速度更快的植物要比生长速度慢的植物中内生菌数量多;生长时间长的植株比刚长出来的植株中内生菌数量多[2]。对于人类来说,内生菌还有很多未知的东西,比如内生菌的代谢物如何促进农作物产量和药用植物有效成分的增长机制,并且在生物肥料中使用内生菌有改善植物营养的作用,在多个领域都有着很大的研究前景。
2. 难以区分的植物内生菌
要检测植物内生菌特别内生细菌并不是一件容易的事,这是因为植物的线粒体16S rDNA (Mt16S)、叶绿体16S rDNA (Ct16S)与细菌的16S rDNA在全长上表现出70%以上的序列同源性,而保守区具有更高的序列同源性。这使得在用16S引物扩增植物内生细菌时,扩增子文库中会产生大量植物源DNA (Mt16S和Ct16S)污染(占比可高达99%以上),严重干扰后续的内生细菌数据分析。
为此,研究者采用了多种手段来尽可能降低植物源DNA的干扰,比如:
1)采用引物799F/1193R(V)(16S的V5-V7)扩增可以产生不含Ct16S的扩增产物,而对于大多数植物的Mt16S的干扰则需要通过去除长约750 bp的产物而去掉[3]。此方法对所检植物物种有限制,比如不适用水稻;
2)重新设计细菌16S特异性地引物(包括引入碱基突变)来降低植物源DNA的干扰,可以在一定程度上提高所鉴定出的内生细菌占比[4];
3)采用巢式PCR,第一步用一对引物扩增出部分16S序列,第二步再用另一对引物从PCR产物中扩增出16S的特定可变区如V4,最后进行内生菌多样性分析[5];我们采用过类似的方案进行内生细菌鉴定,可以降低植物源DNA的占比至20~30%,但仍会因植物物种或是植物器官的不同而产生波动;
4)利用CRISPR/Cas9 系统中Cas9 核酸酶和特异性向导RNA(gRNA)在PCR扩增过程中靶向消除植物源DNA序列。此方法可将水稻根部和叶片中的DNA污染分别从63.2%和99.4%降低到2.9%和11.6%[6];
5)采用锁核苷酸(Locked Nucleic Acid,LNA)或肽核酸(Peptide Nucleic Acid,PNA)作为PCR夹钳在PCR扩增过程中阻断植物源DNA的延伸[7,8,9]。
如果将LNA技术与我们采用过的巢式PCR结合起来是否能更大程度地降低植物源序列的干扰,获取更高比例的内生细菌数据,并且还能具有更广泛的物种适用性?
3. 更好的植物内生细菌测序方法
3.1 LNA-16S测序鉴定内生细菌原理
LNA是一种核苷酸类似物,通过在核糖上加入一个“2 '-O,4'-C-亚甲基”桥链,把糖环的空间构象锁定,提高核酸互补链结合的稳定性,进而提高链结合的特异性(图1)[10]。通过使用LNA可对寡核苷酸序列的Tm值进行微调。一般在引物/探针中每加入一个LNA,热稳定性就可以提高3~8 ℃。一条探针中加入多个LNA,可以大幅提高探针与目标区段结合的稳定性。基于以上优势,带有LNA的寡核苷酸序列常应用于PCR引物、双标记探针、原位杂交探针和分子信标等。LNA也有一个劣势,相比于传统引物,每合成一个LNA碱基是合成常规碱基费用的上百倍。
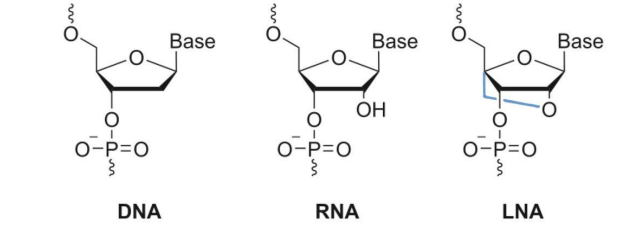
图1. DNA、RNA和LNA核苷酸的结构图
为了验证我们前述的猜想,在巢式PCR鉴定内生菌的方法上,引入LNA引物,是否能进一步降低植物源序列的干扰,并获取更广泛的植物物种适用性,我们进行了如下实验:
参考[8,11]中引物序列和方法,合成了与常规细菌PCR引物有交叠序列,且针对植物细胞器如Ct16S和Mt16S的特异LNA引物。在第一轮PCR反应前,同时加入三对引物,两对是带LNA且分别特异结合Ct16S和Mt16S的引物,一对是不带LNA的常规特异结合细菌16S的引物(可扩增全长16S)。由于LNA引物的Tm值较高,因此在较高温度(70℃)时即可与Ct16S和Mt16S基因退火。之后,由于交叠部分的碱基位置已被LNA引物结合锚定,在到达较低的Tm值时,如54℃时,常规16S引物无法再与植物细胞器基因退火,同时LNA引物的3’端引物是被磷酸化,无法进行序列延伸(图2)。因此,继续进行PCR扩增,只会扩增出细菌的全长16S序列,而抑制植物细胞器基因的扩增。
接着进行第二轮PCR反应,采用常规16S扩增引物(带有测序引物和barcode),如V4或是V3-V4,对第一轮PCR产物进行扩增(即扩增子文库构建)。扩增产物经纯化后,即可用于上机测序。我们称这种16S测序为LNA-16S测序。
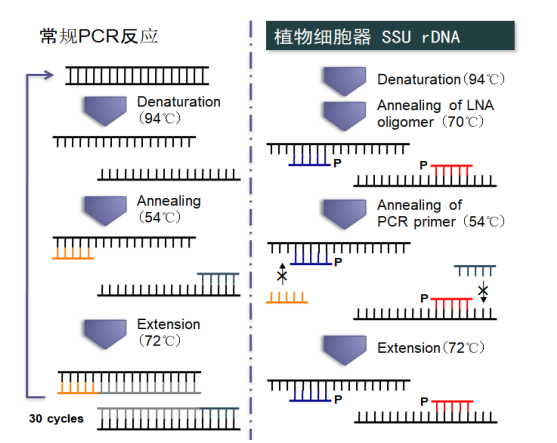
图 2. LNA-PCR工作原理[11]
3.2 LNA-16S测序鉴定内生细菌占比高达99%
LNA-16S测序鉴定内生细菌的表现是否如我们预期的那样?我们采用LNA-16S(V4)测序实测了多种单子叶和双子叶植物的根和叶中的内生细菌,并与非LNA的巢式PCR-16S(V4),常规16S V3-V4以及V5-V6测序的结果进行比较(图3)。
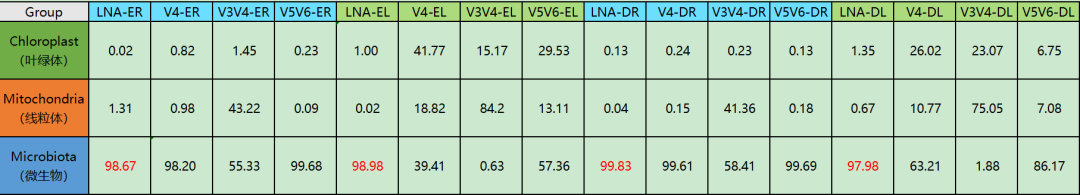
不论是单子叶植物还是双子叶植物,不论是根样本还是叶样本,在LNA-16S测序都能鉴定出极高占比的内生细菌,平均占比为98.86%,最高可达99.83%。相应地,植物源序列的总占比最高仅占2.02%,最低仅占0.17%(表1)。
在我们测试的4种内生细菌鉴定方法中,综合内生细菌和植物源DNA高比率,以及植物物种及器官的广泛适用性和表现,LNA-16S测序都是表现最佳的。因此,我们将LNA-16S测序作为鉴定植物内生细菌的首选方法,为研究者提供检测服务。
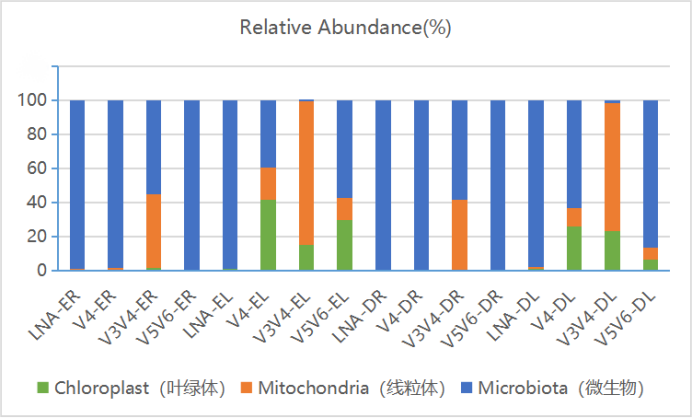
图3. LNA-16S测序与其他16S测序在植物内生细菌鉴定上的比较
LNA:LNA-16S测序(V4),V4:非LNA的巢式PCR-16S测序,V3V4:常规16S V3-V4测序,V5V6:常规16S V5-V6测序,ER:单子叶植物根样品,EL:单子叶植物叶样本,DR:双子叶植物根样本,DL:双子叶植物叶样本
表1. 4种16S测序方法获取的植物内生细菌占比比较(%)
不仅如此,考虑到V3-V4是更常被扩增的16S可变区,我们进一步比较了LNA-16S(V3-V4)和LNA-16S(V4)测序鉴定植物内生细菌的结果(图4)。结果表明,LNA-16S(V3-V4)测序在植物根和叶上鉴定到的内生细菌总占比和优势菌群组成与LNA-16S(V4)测序基本相似,能限制植物源序列干扰在非常小的范围内。此外,LNA-16S(V3-V4)测序鉴定到的各个分类层级上的细菌类别会多一些。
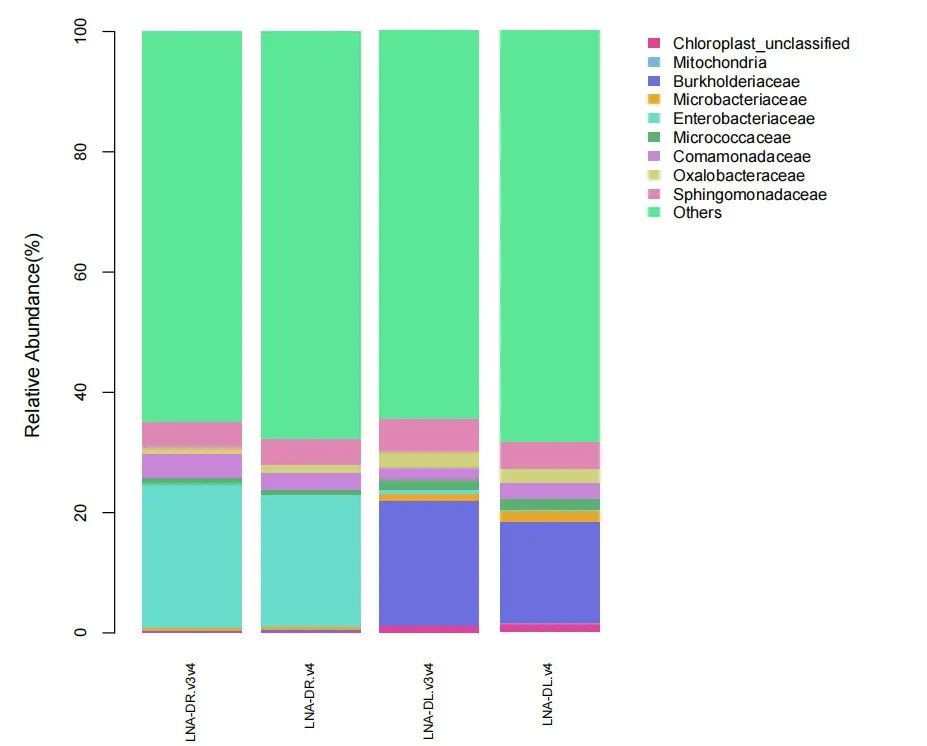
图4. LNA-16S(V3-V4)测序与LNA-16S(V4)测序在植物内生细菌鉴定上的比较(以双子叶植物为例)
4. 植物内生菌研究思路和方法
植物内生菌(包括内生细菌和内生真菌)在植物生长发育,抵抗病毒病菌侵染、抵抗非生物胁迫如干旱、重金属污染等过程中发挥着关键作用,因此对植物内生菌的研究也是植物生物学研究中的重要一环。
以下通过解析一些代表性的植物内生菌研究案例,来帮助读者更好地了解植物内生菌的研究思路和实验设计。
4.1代表性案例精解
案例1:银杏叶片发育期内生细菌群落特征


发表期刊:Front. Microbiol.
影响因子:5.640
发表时间:2021.10.4
研究目的:分析发育期银杏叶片中内生细菌的组成和丰度,以及黄酮类化合物对内生菌群落的影响
研究对象:银杏叶片内生菌
研究方法:16S测序
样本数量:5个时间点×3棵树×3个重复,共有45个

研究设计:
选择三棵野生银杏树(即S1、S2和S3),从4月至8月银杏叶子开始生长时,每月收集叶子样本。将每棵树的树冠分为3个部分,从离地5 ~ 7 m处采集200 g健康叶片作为每个部分的样本。总共在5个时间点从3棵树上采集了45份叶片样本。将样本运到实验室后,每个样本被分成三部分进行植物化学分析、内生菌分离和DNA提取。用于DNA提取的部分需要进行表面灭菌,并在液氮中冷冻,然后在-80°C下储存。采用16S测序对银杏叶片内生细菌进行分析,并讨论黄酮等化合物对内生菌群落的影响。
研究结论:
首次揭示银杏叶片不同生长阶段的细菌群落。银杏叶在不同生长阶段和黄酮类化合物的影响下,不同时期的细菌群落有显著差异。Spearman相关分析表明,内生细菌与黄酮类化合物之间存在较强的相关性。体外培养实验进一步为黄酮类化合物对微生物的影响提供了证据。Tax4Fun2的预测结果表明,黄酮类化合物可能影响银杏叶片不同生长期细菌群落的变化。
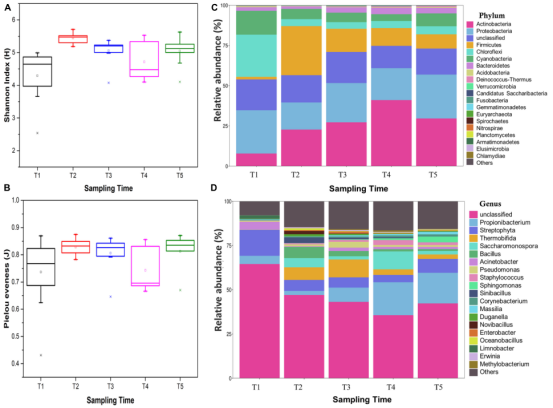
图1 内生细菌群落多样性指数
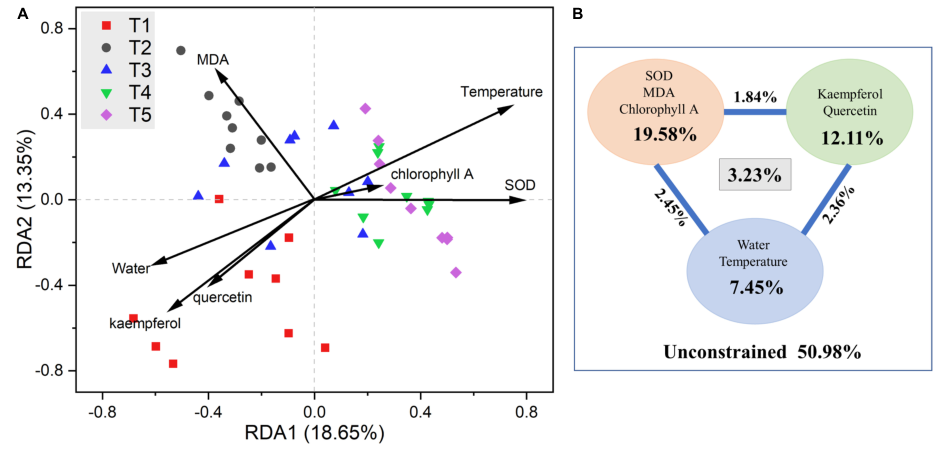
图2. RDA(A)和VPA(B)分析银杏叶片内生细菌群落与环境变量之间的关系
参考文献:
Deng Y, et al. Endophytic Bacterial Communities of Ginkgo biloba Leaves During Leaf Developmental Period. Front Microbiol. 2021 Oct 4;12:698703.
案例2:镉污染稻田不同水稻品种内生细菌群落结构的研究


发表期刊:Front. Microbiol.
影响因子:5.640
发表时间:2021.11.16
研究目的:调查在镉(Cd)污染稻田中生长的不同水稻品种的根内生细菌群落。
研究对象:水稻根内生菌
研究方法:16S测序
样本数量:5个品种×2个时间点×3个重复,共30个

研究设计:
根据籽粒中Cd的积累水平将待测水稻品种分为两组:低Cd积累水平的RBQ、728B和NX1B(LA品种),和高Cd积累水平的BB和S95B(HA品种)。大田试验采用随机区组设计,使用前,将地块充分翻耕,分为15个区(200 ×60 cm),每个区之间的间隔为40 cm。一个区有三栏,一个栏有10个穴,每个穴中种两个苗。每个水稻品种有3个重复(区)。移栽后30 d(营养生长期)和60 d(生殖生长期)分别采集水稻根部样本。对每个水稻品种,在每个区按对角线抽样方法抽样5个单株,并混合根。根部样本分为两部分:一部分用于16S测序,另一部分用于内生细菌分离。处于营养生长期的5个水稻品种的根样本命名为RBQ1、728B1、NX1B1、BB1和S95B1,处于生殖生长期的根样本命名为RBQ2、728B2、NX1B2、BB2和S95B2。
研究结论:
首次详细描述了Cd胁迫下水稻根内生菌组,并为了解内生菌、植物和重金属污染之间的相互作用提供见解。
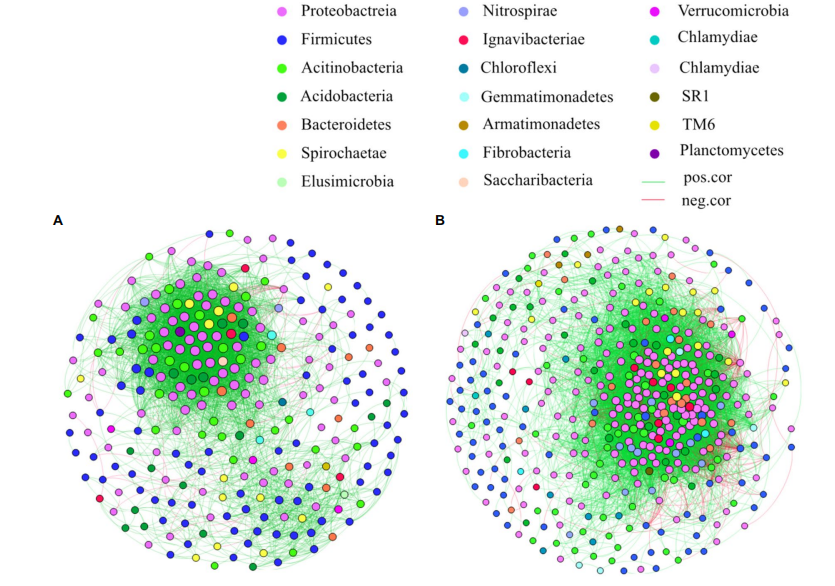
图1. 营养(A)和生殖(B)期样本的共现网络图
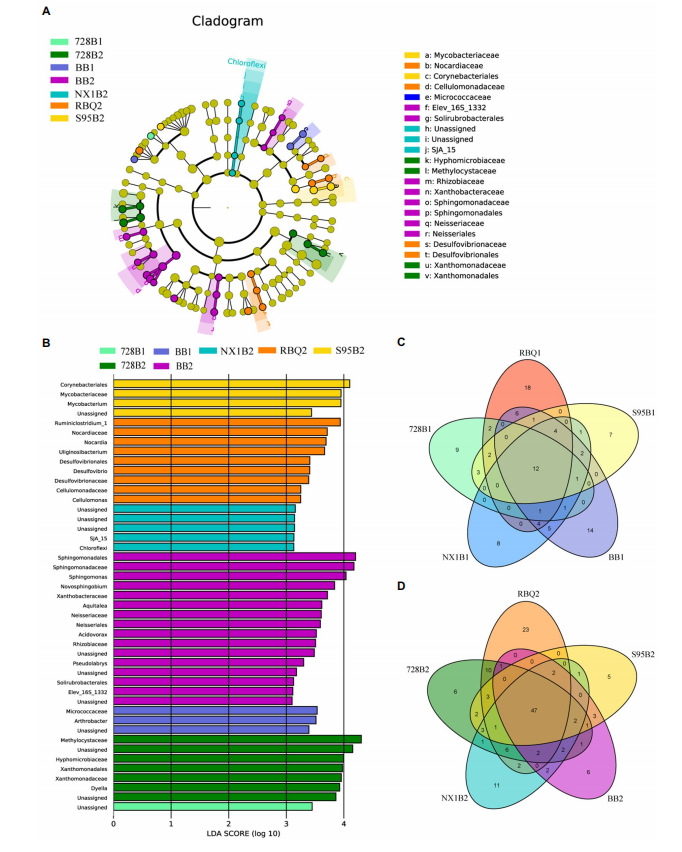
图2. LEfSe和Venn图分析
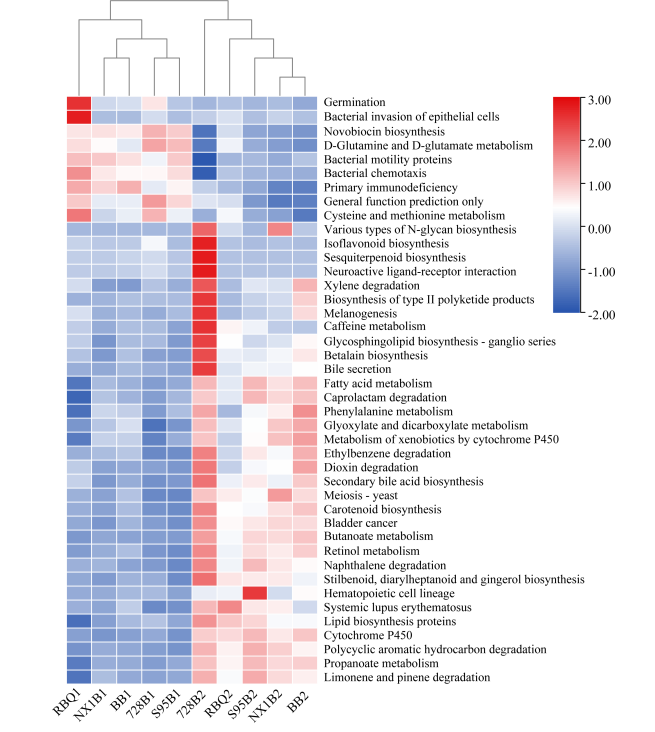
图3. 内生菌群落基因功能预测
参考文献:
Chu C, et al. Unveiling Endophytic Bacterial Community Structures of Different Rice Cultivars Grown in a Cadmium-Contaminated Paddy Field. Front Microbiol. 2021 Nov 16;12:756327.
案例3:根际和内生微生物群落的相互作用对新疆棉花生长的影响


发表期刊:Front. Microbiol.
影响因子:5.640
发表时间:2021.12.06
研究目的:研究棉花根际土壤和根内生菌的相互作用对棉花生长的影响
研究对象:根际土壤+棉花根内生菌
研究方法:16S测序+ITS测序
样本数量:(7个地区×4个时期×3个重复)×2,共336个

研究设计:
在天山北坡和南脚的7个地区的棉花种植区随机采集棉花幼苗、蕾、花和吐絮4个生长时期患病和健康植株的样本。采集的样本包括根和根际土壤(距离根表面0-3 mm)用于16S和ITS测序。
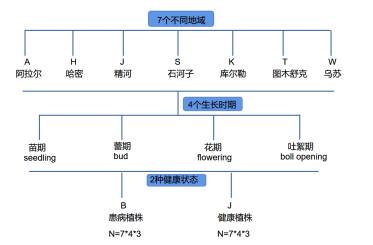
图1 研究设计
研究结论:
发现根际菌落主要为变形菌门、酸杆菌、芽孢菌、放线菌、氯菌、拟杆菌门、子囊菌门、被孢菌门、担子菌门。患病棉花植株中内生细菌和根际细菌和真菌的丰度均高于健康植株。内生细菌和根际细菌没有共同的OTUs。优势菌在不同棉根际土壤和根中的分布不同,优势菌假单胞菌和泛菌属,优势菌链格孢菌和头孢菌数量最多。在不同生态区域的采样点,新疆南至北棉花内生菌和根际微生物群OTUs总数呈上升趋势。在整个棉花生长期中,根际微生物和内生菌的组成和多样性存在显著差异。

图2 根际和内生细菌/真菌属水平群落组成
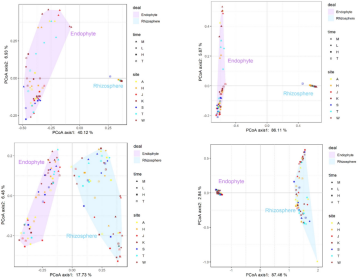
图3 不同采样点和不同生长时期的根际和内生细菌/真菌PCoA分析
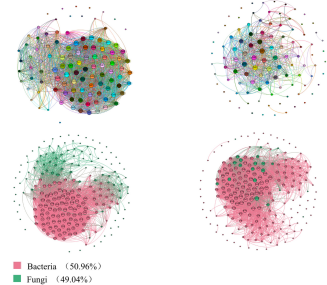
图4 属水平棉根际和内际微生物群落的相关网络分析
参考文献:
Shi Y, et al. Differentiation and Variability in the Rhizosphere and Endosphere Microbiomes of Healthy and Diseased Cotton (Gossypium sp.). Front Microbiol. 2021 Dec 6;12:765269.
案例4:多组学分析揭示内生菌促进冰草生长和耐盐的机制


发表期刊:International Journal of Molecular Sciences
影响因子:5.924
发表时间:2021.10.30
研究目的:探究内生细菌如何影响冰草生长和耐盐
研究对象:根际土壤、冰草根内生细菌 、冰草代谢组
研究方法:16S测序、代谢组检测

研究设计:
采集三个不同地区的土壤,在不同的盐分(50 mM,200 mM和350 mM)下种植冰草。取各个处理的根,经表面消毒后,用16S测序进行内生细菌分析。然后从冰草叶和根分离和培养内生菌。经过两轮筛选促植物生长的内生细菌(PGPE),将它们与冰草种子一起培育,通过测定冰草生长1周后的鲜重和根长,来评价各菌株的促生效果。对选定的菌株测定促植物生长的特征并进行系统发育分析。最后,对接种PGPE 2和4在不同处理下的冰草进行代谢组学分析。
研究结论:
分离得到的两株内生菌都具有促进植物生长的作用。只有4号菌株产生IAA,而2号菌株促进植株生长,但不产生IAA或其他任何供试性状。代谢谱突出了盐胁迫下和接种PGPE后代谢物的差异。特别是,在种植了PGPE的植物中,某些代谢物的丰度增加了,例如那些与TCA循环、ABC转运蛋白和光合作用有关的代谢物(图7)。盐度也影响了冰草的新陈代谢,导致相容溶质和多胺含量的变化。相比之下,模拟接种且盐胁迫的植株中的许多物质含量在接种菌株2或4的盐胁迫植株中都表现出下降的趋势,表明这些菌株缓解了盐胁迫的影响。
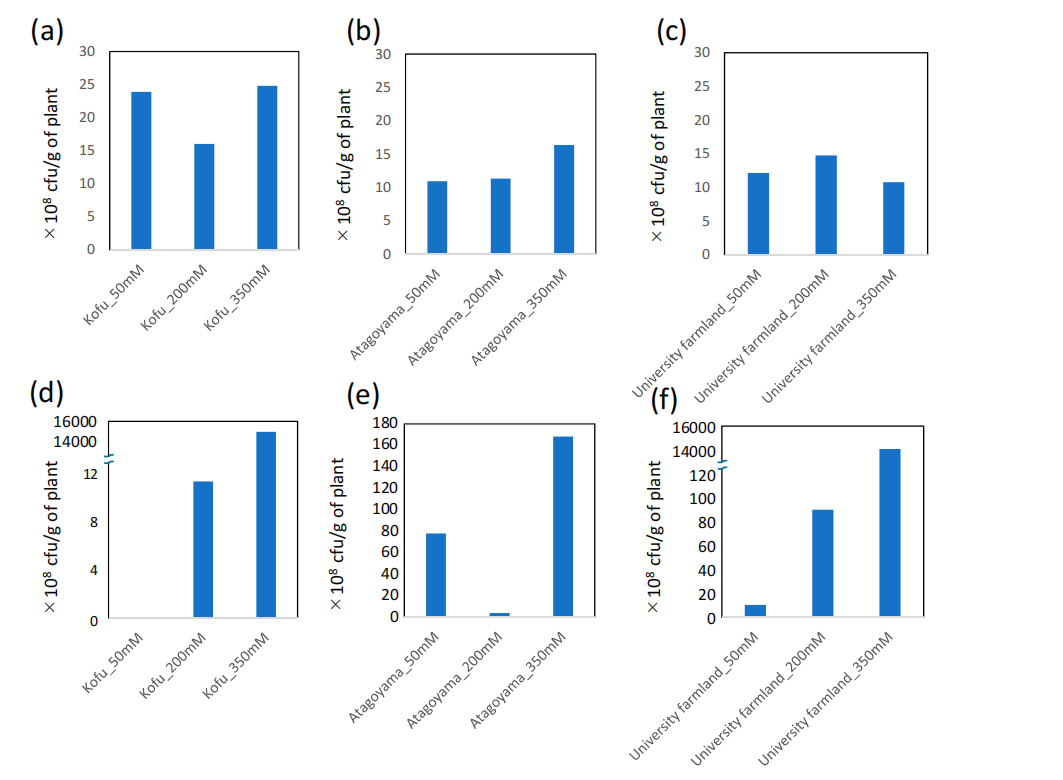
图1. 根际土壤微生物(a-c)和内生细菌(d-f)的数量
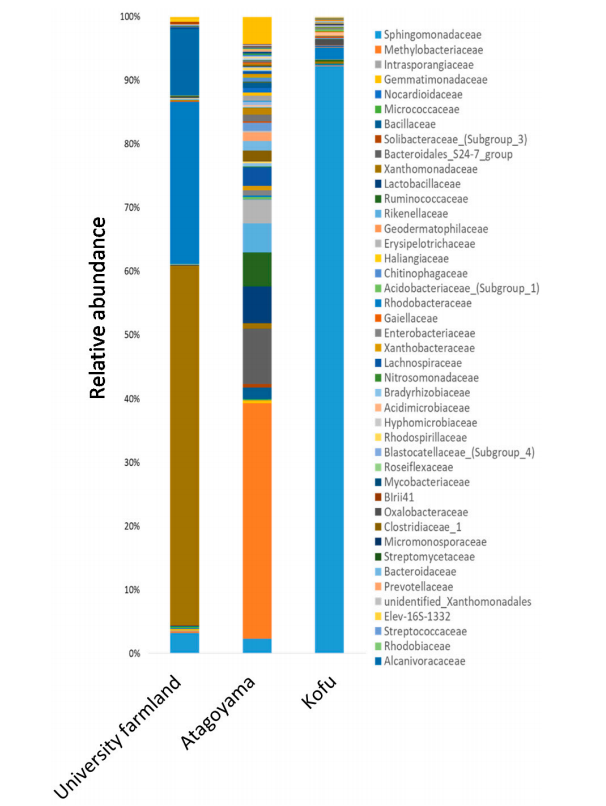
图2. 生长在三种不同土壤里的冰草内生细菌家族
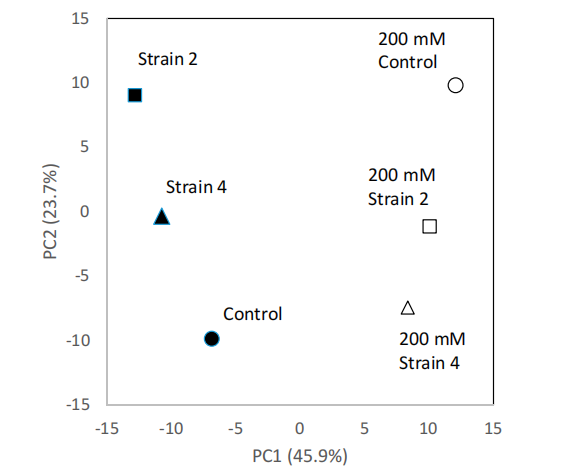
图3. 不同处理的冰草代谢物的PCA分析
参考文献:
Kataoka R, et al. Metabolomics Analyses Reveal Metabolites Affected by Plant Growth-Promoting Endophytic Bacteria in Roots of the Halophyte Mesembryanthemum crystallinum. Int J Mol Sci. 2021 Oct 30;22(21):11813.
4.2 研究思路和方法小结
整体研究思路上并没有与其他环境微生物组的研究思路有明显的不同(>>一文讲透环境微生物组研究怎么做)。植物内生菌的研究内容主要集中在内生菌鉴定、对寄主植物生长发育的影响、对抗逆应答的作用、内生菌与其他环境微生物比如根际土壤微生物联合对寄主植物生长发育和抗逆应答的作用等。
具体地,做单组学研究,建议做多对象研究,可以是研究内生细菌和内生真菌,也可以是研究根、茎、叶中的内生菌,也可以是根际土壤微生物和根内生菌;做多组学研究,仍然是优先和代谢组联合,也可以是联合宿主的转录组或蛋白质组;内生菌一般不建议做宏基因组,因为宿主DNA占比过高。在实验设计上,最常见的是不同处理×不同时间点,不同品种×不同时间点。研究内生菌的一个突出不同点是,有许多研究加入了内生菌的分离培养和功能验证。关于样本重复数,也与其他环境微生物组一样,我们建议每组样本重复数≥ 5个。
参考文献:(滑动查看)
1. Papik J, Folkmanova M, Polivkova-Majorova M, et al. The invisible life inside plants: Deciphering the riddles of endophytic bacterial diversity. Biotechnol Adv. 2020 Nov 15;44:107614.
2. Wu W, Chen W, Liu S, et al. Beneficial Relationships Between Endophytic Bacteria and Medicinal Plants. Front Plant Sci. 2021 Apr 22;12:646146.
3. Bai Y, Müller D B, Srinivas G, et al. (2015). Functional overlap of the Arabidopsis leaf and root microbiota. Nature 528, 364–369.
4. Chen L, et al. Designing specific bacterial 16S primers to sequence and quantitate plant endo-bacteriome. Sci China Life Sci. 2022 May;65(5):1000-1013.
5. Yu J, et al. Design and application of specific 16S rDNA-targeted primers for assessing endophytic diversity in Dendrobium officinale using nested PCR-DGGE. Appl Microbiol Biotechnol. 2013 Nov;97(22):9825-36.
6. Song L, and Xie K. (2020). Engineering CRISPR/Cas9 to mitigate abundant host contamination for 16S rRNA gene-based amplicon sequencing. Microbiome 8, 80.
7. Ikenaga M, Sakai M. Application of Locked Nucleic Acid (LNA) oligonucleotide-PCR clamping technique to selectively PCR amplify the SSU rRNA genes of bacteria in investigating the plant-associated community structures. Microbes Environ. 2014 Sep 17;29(3):286-95.
8. Ikenaga M, et al. Improvements in Bacterial Primers to Enhance Selective SSU rRNA Gene Amplification of Plant-associated Bacteria by Applying the LNA Oligonucleotide-PCR Clamping Technique. Microbes Environ. 2018 Sep 29;33(3):340-344.
9. Matsumoto H, Fan X, Wang Y, et al. (2021). Bacterial seed endophyte shapes disease resistance in rice. Nat Plants 7, 60–72.
10. Singh S. K, et al. (1998). LNA (locked nucleic acids): synthesis and high-affinity nucleic acid recognition. Chem. Commun. 4, 455-456.
11. Yu Z H, Ikenaga M, Sakai M, et al.(2021). Locked Nucleic Acid (LNA) Oligonucleotide PCR Clamping Method for Investigating the Plant Endophyte Bacteria. // Microbiome Protocols eBook. Bio-101: e2003525.
相关阅读
宏基因组测序这么用,让你的微生物研究芜湖起飞 | 微生物专题
万万没想到!FFPE样本居然还能做肿瘤微生物组!|微生物专题
猜你喜欢
iMeta简介 高引文章 高颜值绘图imageGP 网络分析iNAP
iMeta网页工具 代谢组MetOrigin 美吉云乳酸化预测DeepKla
iMeta综述 肠菌菌群 植物菌群 口腔菌群 蛋白质结构预测
10000+:菌群分析 宝宝与猫狗 梅毒狂想曲 提DNA发Nature
一文读懂:宏基因组 寄生虫益处 进化树 必备技能:提问 搜索 Endnote
16S功能预测 PICRUSt FAPROTAX Bugbase Tax4Fun
生物科普: 肠道细菌 人体上的生命 生命大跃进 细胞暗战 人体奥秘
写在后面
为鼓励读者交流快速解决科研困难,我们建立了“宏基因组”讨论群,己有国内外6000+ 科研人员加入。请添加主编微信meta-genomics带你入群,务必备注“姓名-单位-研究方向-职称/年级”。高级职称请注明身份,另有海内外微生物PI群供大佬合作交流。技术问题寻求帮助,首先阅读《如何优雅的提问》学习解决问题思路,仍未解决群内讨论,问题不私聊,帮助同行。
点击阅读原文,跳转最新文章目录阅读
