之前有人在公众号留言问过用R语言如何实现t-SNE降维,今天的推文介绍一下R语言实现的代码,主要内容参考自链接 https://datavizpyr.com/how-to-make-tsne-plot-in-r/
t-SNE 的全称是 t-Distributed Stochastic Neighbor Embedding,具体的计算原理我也不懂,反正就是类似PCA把高维度的数据转换成低维度的数据
示例数据集用到的是企鹅的数据集,名称是penguins,这个数据集来自于R包palmerpenguins,如果要用这个数据还需要安装这个R包,实现t-SNE需要用到R包Rtsne。R包tidyverse是用来做数据整理的,所以先加载这三个R包,如果是第一次使用需要先安装,安装命令是
install.packages("tidyverse")
install.packages("palmerpenguins")
install.packages("Rtsne")
加载需要用到的R包
library(tidyverse)
library(palmerpenguins)
library(Rtsne)
选择数据集中的数值型变量用于后续分析
penguins %>%
select(where(is.numeric),-year,species) %>%
mutate(ID=row_number()) %>%
column_to_rownames("ID") %>%
na.omit()-> df
这里新学到的函数
-
选择数据框中的数值变量 select(where(is.numeric)) -
给数据集添加1:多少行的数字 mutate(ID=row_number()) -
数据集里指定列作为行名(前提是不能有重复) column_to_rownames("ID")
t-SNE降维
tSNE_fit<-df %>%
select(-species) %>%
scale() %>%
Rtsne()
提取降维结果
tSNE_fit$Y %>%
as.data.frame() %>%
rename(tSNE1="V1",
tSNE2="V2") %>%
mutate(Species=df$species) -> tSNE.plot
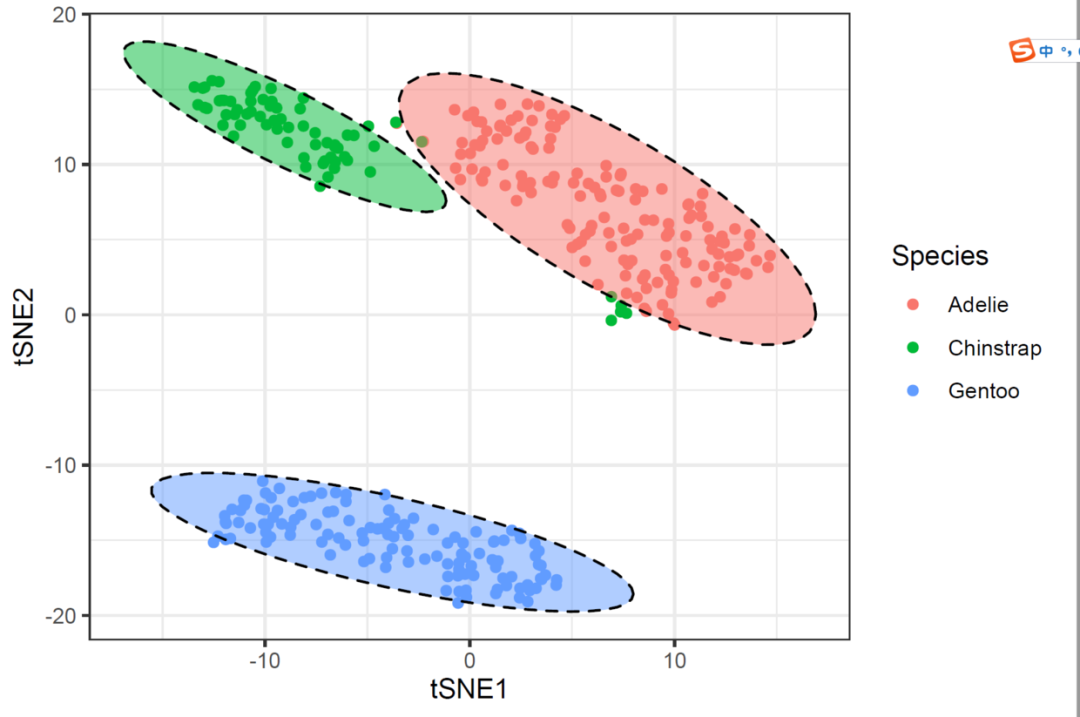
散点图展示结果
library(ggplot2)
ggplot()+
geom_point(data=tSNE.plot,
aes(x=tSNE1,y=tSNE2,color=Species))+
stat_ellipse(data=tSNE.plot,
geom="polygon",
aes(x=tSNE1,y=tSNE2,
group=Species,
fill=Species),
alpha=0.5,
lty="dashed",
color="black",
key_glyph="blank")+
theme_bw()
优雅的修改图例
https://mp.weixin.qq.com/s/I3YnxqulQRu-9i-gZIh7fA
欢迎大家关注我的公众号
小明的数据分析笔记本
留言讨论相关内容
今天推文的示例代码可以在公众号后台留言回复20210827获取
小明的数据分析笔记本 公众号 主要分享:1、R语言和python做数据分析和数据可视化的简单小例子;2、园艺植物相关转录组学、基因组学、群体遗传学文献阅读笔记;3、生物信息学入门学习资料及自己的学习笔记!
本文分享自微信公众号 - 小明的数据分析笔记本(gh_0c8895f349d3)。
如有侵权,请联系 [email protected] 删除。
本文参与“OSC源创计划”,欢迎正在阅读的你也加入,一起分享。
{{o.name}}
{{m.name}}