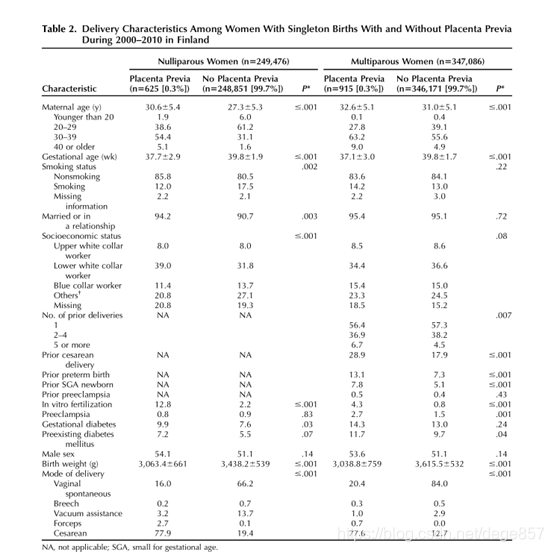
在前面的章节中,我们已经探讨了2种SCI单因素表的制作方法,今天我们来将第三种表,其实这三种表已经涵盖了绝大部分的SCI的单因素表,只要您有心,绝对可以做出来。今天我们来看看这第三种,这种就像是第二种的加强版,先把产妇分为未生育的和已经生育的,再在里面比较两个人群前置胎盘患者的关系。
我们还是使用既往的乳腺癌的数据
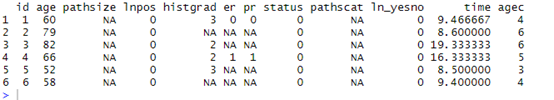
age表示年龄,pathsize表示病理肿瘤大小(厘米),lnpos表示腋窝淋巴结阳性,histgrad表示病理组织学等级,er表示雌激素受体状态,pr表示孕激素受体状态,status结局事件是否死亡,pathscat表示病理肿瘤大小类别(分组变量),ln_yesno表示是否有淋巴结肿大,time是生存时间,后面的agec是我们自己设定的,不用管它。
library(foreign)
library(survival)
bc <- read.spss(“E:/r/Breast cancer survival agec.sav”,
use.value.labels=F, to.data.frame=T)
删除缺失值,查看数据
bc <- na.omit(bc)
head(bc)
这是数据格式
我们先把是否有淋巴结转移,分成两个数据表,再分别进行分析
ln_yesno0<-subset(bc,bcKaTeX parse error: Expected 'EOF', got '#' at position 12: ln_yesno<1)#̲我们告诉subset 函数在l…ln_yesno>=1)
这样我们就得到了ln_yesno0,ln_yesno1这两个分类数据集,我们就可以用它来分别进行统计了。
我们把年龄分为3个类别,小于40岁,40-60岁,大于60岁的,比较在这三个年龄区间孕激素状态对对无淋巴结转移人群对死亡结局的影响
先来做一个小于40岁人群中,孕激素状态(pr)在无淋巴结转移人群对死亡结局的影响
建立模型
f.age1 <- glm(status ~ pr, family = binomial(link = “logit”), data = ln_yesno0,subset = (age<=40))
对模型解析
summary(f.age1)
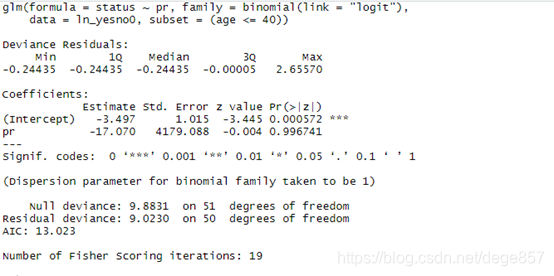
已经得出P值和标准误,已经可以做表了,如果你要求更高一点,还可以继续求95%CI和OR(参看我原来的帖子)
再来来做一个小于40岁人群中,孕激素状态(pr)在有淋巴结转移人群对死亡结局的影响(注意这里就要换一个数据集)
f.age2<- glm(status ~ pr, family = binomial(link = “logit”), data = ln_yesno1,subset = (age<=40))
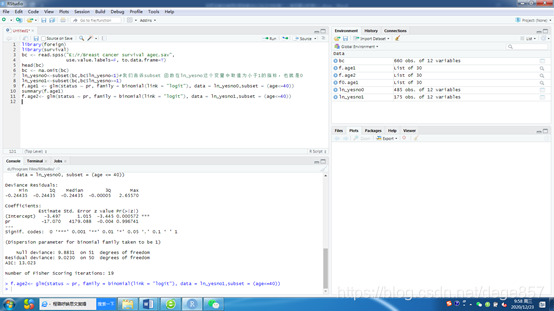
再来一个做一个小于40-60岁人群中,孕激素状态(pr)在无淋巴结转移人群对死亡结局的影响
f.age3 <- glm(status ~ pr, family = binomial(link = “logit”), data = ln_yesno0,subset = (age>40 & age<=60))
再来一个做一个小于40-60岁人群中,孕激素状态(pr)在有淋巴结转移人群对死亡结局的影响(换个数据集而已)
f.age4 <- glm(status ~ pr, family = binomial(link = “logit”), data = ln_yesno0,subset = (age>40 & age<=60))
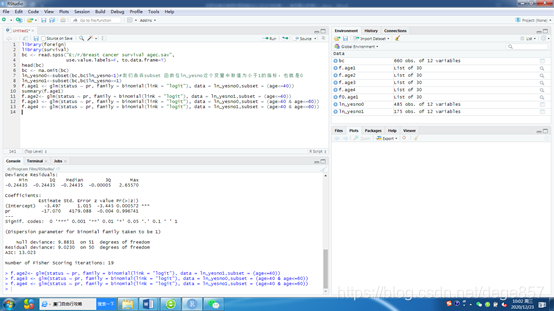
这样就可以依次做出表中所有的数据了,其实代码和原来很多都是重复,也不难做,但是你的思路一定要清晰,明白它是怎么做出来的。我们已经把大部分的单因素表格做出来了,下次将继续介绍怎么做SCI的多因素表。

手把手教你使用R语言做出SCI论文中的表二(单因素分析表)(3)
猜你喜欢
转载自blog.csdn.net/dege857/article/details/111576501
今日推荐
周排行