背景简介
中国是世界上糖尿病患者最多的国家,病人达到1.1亿,每年有130万人死于糖尿病及其相关疾病。每年用于糖尿病的医疗费用占中国公共医疗卫生支出的比例超过13%,超过3000亿元。本次大赛旨在通过糖尿病人的临床数据和体检指标来预测人群的糖尿病程度,以血糖浓度为指标。参赛选手需要设计高精度,高效,且解释性强的算法来挑战糖尿病精准预测这一科学难题。
数据简介
大赛初赛数据共包含两个文件,训练文件d_train.csv和测试文件d_test.csv,每个文件第一行是字段名,之后每一行代表一个个体。文件共包含42个字段,包含数值型、字符型、日期型等众多数据类型,部分字段内容在部分人群中有缺失,其中第一列为个体ID号。训练文件的最后一列为标签列,既需要预测的目标血糖值。
大赛复赛数据共包含两个文件,训练文件f_train.csv和测试文件f_test.csv,每个文件第一行是字段名,之后每一行代表一个个体,部分字段名已经做脱敏处理。文件共包含85个字段,部分字段内容在部分人群中有缺失,其中第一列为个体ID号。训练文件的最后一列为标签列,既需要预测的是否有糖尿病的类别。
一、数据准备与探索
import pandas as pd
import numpy as np
import os
import matplotlib.pyplot as plt
import seaborn as sns
from sklearn.impute import SimpleImputer
from sklearn.ensemble import RandomForestRegressor
from sklearn.preprocessing import StandardScaler
from sklearn.feature_selection import variance_threshold
from sklearn.feature_selection import mutual_info_regression as MIR
from sklearn.feature_selection import SelectKBest
from sklearn.feature_selection import SelectFromModel
from sklearn.feature_selection import RFE
from sklearn.model_selection import train_test_split
from sklearn.model_selection import cross_val_score
os.chdir(r'C:\userMe\jupyter_notebook\项目三医疗数据\精准医疗大赛')
d_train = pd.read_csv('d_train_20180102.csv', encoding = 'gb2312')
d_test = pd.read_csv('d_test_A_20180102.csv', encoding = 'gb2312')
print(d_train.columns) # 查看列名
print(d_train.head())
round(d_train.describe()) # 查看数据分布
d_train.shape
"""
第一阶段:数据准备
"""
d_train = d_train.drop('id', axis=1) # 将id列删除
d_train['性别'] = d_train['性别'].map({'男': 1,'女': 0, '??':1}) # 将男女进行二值转化
d_train.groupby(['性别']).size().plot.pie(subplots=True,autopct='%.2f', fontsize=20)
d_test = d_test.drop('id', axis=1)
d_test['性别'] = d_test['性别'].map({'男': 1,'女': 0, '??':1})
d_test.groupby(['性别']).size().plot.pie(subplots=True,autopct='%.2f', fontsize=20)
| id | 年龄 | *天门冬氨酸氨基转换酶 | *丙氨酸氨基转换酶 | *碱性磷酸酶 | *r-谷氨酰基转换酶 | *总蛋白 | 白蛋白 | *球蛋白 | 白球比例 | ... | 血小板计数 | 血小板平均体积 | 血小板体积分布宽度 | 血小板比积 | 中性粒细胞% | 淋巴细胞% | 单核细胞% | 嗜酸细胞% | 嗜碱细胞% | 血糖 | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| count | 5642.0 | 5642.0 | 4421.0 | 4421.0 | 4421.0 | 4421.0 | 4421.0 | 4421.0 | 4421.0 | 4421.0 | ... | 5626.0 | 5619.0 | 5619.0 | 5619.0 | 5626.0 | 5626.0 | 5626.0 | 5626.0 | 5626.0 | 5642.0 |
| mean | 2866.0 | 46.0 | 27.0 | 28.0 | 87.0 | 39.0 | 77.0 | 46.0 | 31.0 | 2.0 | ... | 253.0 | 11.0 | 13.0 | 0.0 | 57.0 | 34.0 | 7.0 | 2.0 | 1.0 | 6.0 |
| std | 1656.0 | 13.0 | 14.0 | 23.0 | 26.0 | 41.0 | 4.0 | 3.0 | 4.0 | 0.0 | ... | 60.0 | 1.0 | 2.0 | 0.0 | 8.0 | 7.0 | 2.0 | 2.0 | 0.0 | 2.0 |
| min | 1.0 | 3.0 | 10.0 | 0.0 | 23.0 | 6.0 | 57.0 | 30.0 | 7.0 | 1.0 | ... | 37.0 | 7.0 | 8.0 | 0.0 | 14.0 | 8.0 | 3.0 | 0.0 | 0.0 | 3.0 |
| 25% | 1433.0 | 35.0 | 20.0 | 15.0 | 70.0 | 18.0 | 74.0 | 44.0 | 29.0 | 1.0 | ... | 213.0 | 10.0 | 12.0 | 0.0 | 52.0 | 29.0 | 6.0 | 1.0 | 0.0 | 5.0 |
| 50% | 2870.0 | 45.0 | 24.0 | 21.0 | 84.0 | 26.0 | 77.0 | 46.0 | 31.0 | 1.0 | ... | 249.0 | 11.0 | 13.0 | 0.0 | 57.0 | 34.0 | 7.0 | 2.0 | 1.0 | 5.0 |
| 75% | 4303.0 | 54.0 | 29.0 | 32.0 | 100.0 | 44.0 | 80.0 | 48.0 | 33.0 | 2.0 | ... | 289.0 | 11.0 | 15.0 | 0.0 | 62.0 | 38.0 | 8.0 | 3.0 | 1.0 | 6.0 |
| max | 5732.0 | 93.0 | 435.0 | 499.0 | 374.0 | 737.0 | 100.0 | 54.0 | 66.0 | 7.0 | ... | 745.0 | 15.0 | 25.0 | 1.0 | 88.0 | 76.0 | 23.0 | 22.0 | 4.0 | 38.0 |
二、处理缺失值
"""
缺失值处理
"""
#将字段名更改为 var_0-40(避免乱码问题)
for i in range(d_train.shape[1]):
d_train = d_train.rename(columns={d_train.columns[i]:'var_'+ str(i)})
for i in range(d_test.shape[1]):
d_test = d_test.rename(columns={d_test.columns[i]:'var_'+ str(i)})
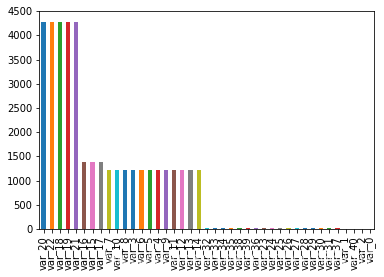
# 画图查看缺失值分布情况
nulls = d_train.isnull().sum().sort_values(ascending=False)
nulls.plot(kind='bar')
(nulls/d_train.shape[0])*100
# 删除缺失值占比超过70%的字段(乙肝检查)
remove1 = ["var_18","var_19","var_20","var_21","var_22"]
train_1 = d_train.drop(remove1, axis=1)
test_1 = d_test.drop(remove1, axis=1)
train_1.shape
(train_1.isnull().sum().sort_values(ascending=False)/train_1.shape[0])*100
# 查看极值
des = train_1.quantile([0, 0.01,0.1,0.5,0.8,0.9,0.99,1])
des
# 画图查看极值
for i in range(des.shape[1]):
# i = 1
plt.plot(des.iloc[:,i],c='b')
plt.ylabel(des.columns[i])
plt.show()
# 查看一下血糖分布
plt.plot(train_1.loc[:, 'var_40'])
plt.hist(train_1.loc[:, 'var_40'], alpha=0.5, bins=50)
# 用随机森林对其他缺失值进行填补
train_1 = train_1.drop('var_2', axis=1)
x_missing = train_1.copy()
x_missing = x_missing.drop('var_0', axis = 1)
missing = x_missing.isnull().sum().sort_values(ascending=False)
for i in missing.index:
# i = 'var_16'
y = x_missing[str(i)]
x = x_missing.drop(str(i), axis=1)
if len(y)==0:
break
# 将其他缺失值用0填充
x_0 = SimpleImputer(missing_values=np.nan, strategy='constant', fill_value=0).fit_transform(x)
ytrain = y[y.notnull()]
ytest = y[y.isnull()]
xtrain = x_0[ytrain.index, :]
xtest = x_0[ytest.index, :]
rfc = RandomForestRegressor(n_estimators=100)
rfc = rfc.fit(xtrain, ytrain)
ypredict = rfc.predict(xtest)
# 将填好的特征返回到原始特征矩阵中
x_missing.loc[x_missing.loc[:,str(i)].isnull(),str(i)] = ypredict
train_2 = x_missing.copy()
train_2.isnull().sum()缺失值占比: var_20 75.841900 var_22 75.841900 var_18 75.841900 var_19 75.841900 var_21 75.841900 var_16 24.423963 var_15 24.423963 var_17 24.423963 var_7 21.641262 var_10 21.641262 var_8 21.641262 var_3 21.641262 var_6 21.641262 var_5 21.641262 var_4 21.641262 var_9 21.641262 var_11 21.605814 var_12 21.605814 var_13 21.605814 var_14 21.605814 var_32 0.407657 var_33 0.407657 var_34 0.407657 var_35 0.283587 var_38 0.283587 var_39 0.283587 var_36 0.283587 var_23 0.283587 var_24 0.283587 var_25 0.283587 var_26 0.283587 var_27 0.283587 var_28 0.283587 var_29 0.283587 var_30 0.283587 var_31 0.283587 var_37 0.283587 var_1 0.000000 var_40 0.000000 var_2 0.000000 var_0 0.000000 dtype: float64
三、特征分析与筛选
"""
特征分析与筛选
"""
# 方差及相关性查看(相关性还可以用F检验现行相关性)
train_2.var().sort_values(ascending=False)
train_2.corr()
# 互信息法(最终保留27个变量)
result = MIR(train_2.drop('var_40',axis=1), train_2['var_40'])
result1 = pd.concat([pd.DataFrame(result),pd.DataFrame(train_2.drop('var_40',axis=1).columns)], axis=1)
col_name = list(result1[result1.iloc[:,0]!=0].iloc[:,1])
k = result.shape[0] - sum(result <= 0)
train_3 = SelectKBest(MIR, k = 27).fit_transform(train_2.drop('var_40',axis=1), train_2['var_40'])
# 将数据标准化
train_3 = StandardScaler().fit_transform(train_3)
train_3 = pd.DataFrame(train_3)
train_3.columns = col_name四、模型选择
1、线性回归+嵌入法进行变量筛选及建模
2、用sklearn的RandomForestRegressor随机森林的回归树建模
3、用sklearn的xgboost进行建模