Review综述:3D Unet vs 2D Unet
[email protected]
TL;DR 太长不读版:
2D U-Net:
数据量比较小,3维数据的z轴slice数比较少
病理在三维上是稀疏的,2D网络更好
3D U-Net:
数据量比较大,3维数据的z轴slice数比较多时
专注于细节和局部特征,用于小目标的分割(参考脑肿瘤分割的论文)
病理在2D上没有明显征象,3D有。
几种经典的卷积
卷积神经网络(CNN)已显示出实现多种计算机视觉任务的能力(例如图像分割)。分割任务主要有两种: 语义分割和实例分割。语义分割是对图像中的每个像素都划分出对应的类别,即实现像素级别的分类; 而类的具体对象,即为实例,实例分割不但要进行像素级别的分类,还需在具体的类别基础上区别开不同的实例。比如说图像有多个人甲、乙、丙,那边他们的语义分割结果都是人,而实例分割结果却是不同的对象.
在ResNet以后使用深度学习进行分类任务的性能已经超过了人类,但是目标检测任务和分割任务的准确度却一直较低,其中分割任务的准确度是最低的。因为分割任务需要对每个像素点进行精确的分类,其难度最大。针对语义分割的需求,最早由Jonathan Long等人提出的Fully Convolutional Networks(FCN)成为了语义分割领域的基本框架之一,后续很多框架都多少参考了FCN的思想。但是FCN的准确度较低,不如U-net用的广泛. 目前已经有不少其他好用的分割网络:Mask RCNN, DeepLabv3+, FCIS.
由于语义分割需要输入输出都是图像,所以与之前经典的图像分类和目标检测网络在分割任务上就不大适用了。在此前的经典网络中,经过多层卷积和池化之后输出的特征图尺寸会逐渐变小,所以对于语义分割任务我们需要将逐渐变小的特征图给还原到输入图像的大小。为了实现上述目标,现有的语义分割等图像分割模型的一种通用做法就是采用编码和解码的网络结构,此前的多层卷积和池化的过程可以视作是图像编码的过程,也即不断的下采样的过程。那解码的过程就很好理解了,可以将解码理解为编码的逆运算,对编码的输出特征图进行不断的上采样逐渐得到一个与原始输入大小一致的全分辨率的分割图。在介绍u-net之前,我们来看一下卷积网络的基础,几种经典的卷积。
1D Convolutions 一维卷积
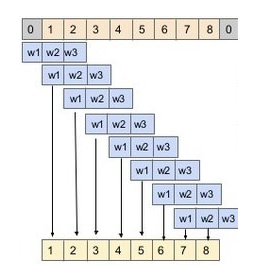
2D Convolutions 二维卷积
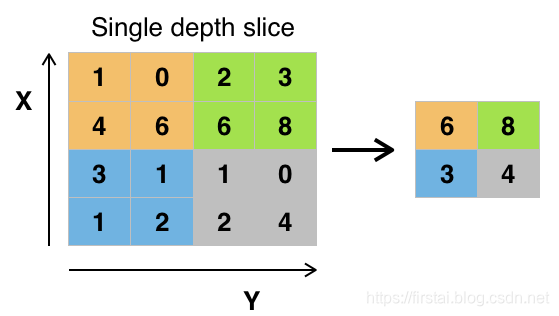
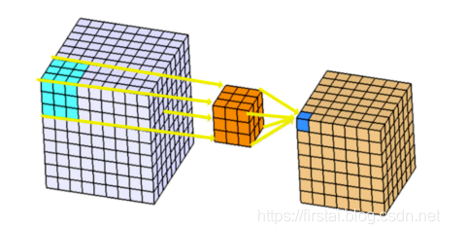
3D Convolutions 三维卷积
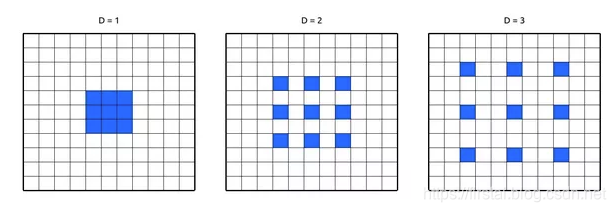
Dilated Convolutions 膨胀卷积/空洞卷积
U-Net
U-Net 是基于FCN的改进和发展,我们先看一下FCN。
FCN
FCN是对图像进行像素级分类的代表,是深度学习在图像分割的开山之作,率先给出了语义级别的图像分割解决方案。总的而言,FCN遵循编码解码的网络结构模式,使用 AlexNet 作为网络的编码器,采用转置卷积(反卷积)对编码器最后一个卷积层输出的特征图进行上采样直到特征图恢复到输入图像的分辨率,因而可以实现像素级别的图像分割。FCN的一个好处是可输入任意尺寸的图像进行语义分割。
U-Net
作为 FCN 的一种改进和发展,Ronneberger 等人通过扩大网络解码器的容量来改进了全卷积网络结构,并给编码和解码模块添加了收缩路径(contracting path)来来实现更精准的像素边界定位。u-net 的结构如下图所示:
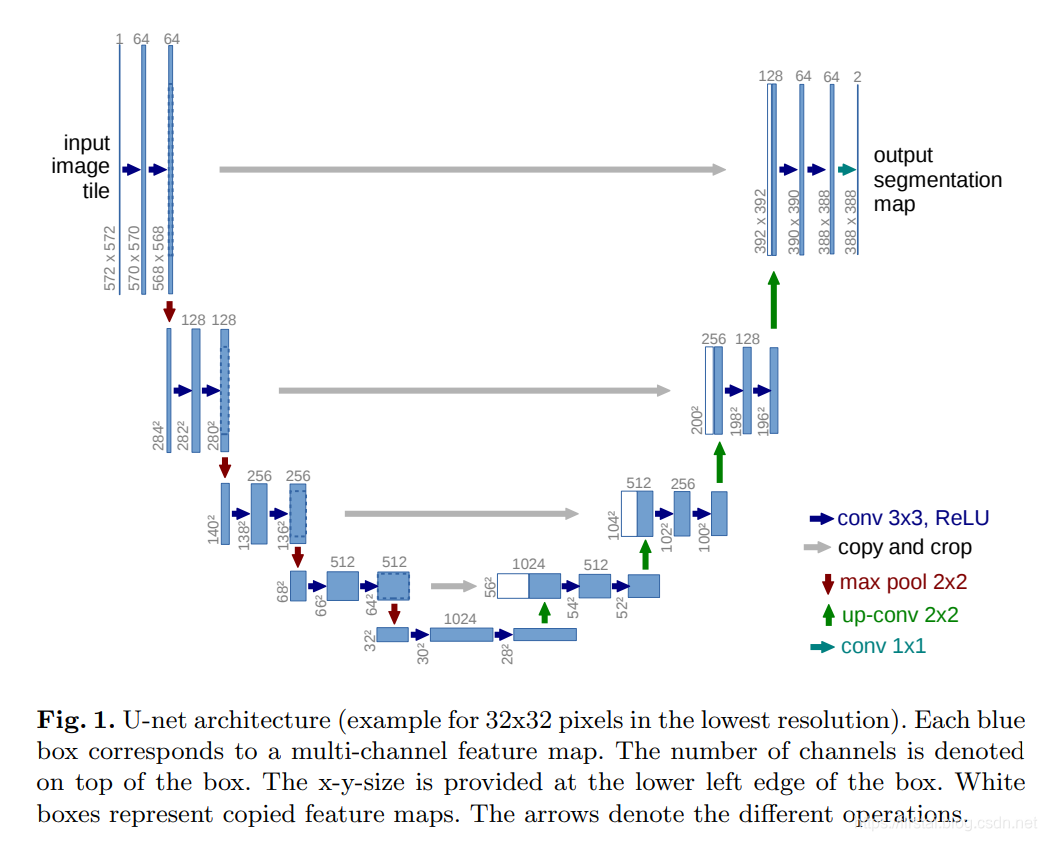
Unet包括两部分,第一部分,特征提取,VGG类似。第二部分上采样部分。由于网络结构像U型,所以叫Unet网络。特征提取部分,每经过一个池化层就一个尺度,包括原图尺度一共有5个尺度。上采样部分,每上采样一次,就和特征提取部分对应的通道数相同尺度融合,但是融合之前要将其crop。这里的融合也是拼接。
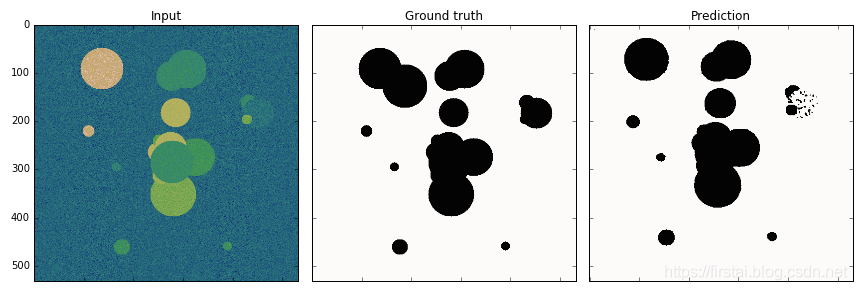
u-net 在大量医学影像分割上的效果使得这种语义分割的网络架构非常流行,近年来在一些视觉比赛的冠军方案中也随处可见 u-net 的身影。U-Net分割例子:
3D U-Net
在生物医学图像领域,3D数据是很常见的,一层一层转化为2D数据去标注训是不现实的,而且用整个3D体积的全部数据去训练既低效又极有可能过拟合。3D Unet只需要少部分2D的标注切片就可以生成密集的立体的分割。此网络主要有两个不同的作用方式,一是在一个稀疏标注的数据集上训练并在此数据集上预测其他未标注的地方,另一个方法是在多个稀疏标注的数据集训练,然后预测新的数据。
3D-Unet的结构基本上和2D一模一样,只是增加了一个维度。

3D vs 2D U-Net
以下参考https://www.zhihu.com/question/271497684/answer/611486904
3D数据和2D数据相比,多了一个方向的信息。举个例子,如果将一根黄瓜切成300片,随便抽一片出来让你辨认这是什么,可能会和其他的蔬果搞混,但是如果把这300片拼起来组成它原本黄瓜的样子,你一定会认识这是黄瓜。2D数据的表达为(x,y),3D数据的表达为(x,y,z)。医疗影像的大部分数据都是3D的,也就是多层slice叠加而成的。但是由于z轴上的像素间距(pixel spacing)不同,3D的数据也被为薄层数据和厚层数据。薄层数据层厚较薄,所以z轴slice数比较多,比如眼底OCT图片的z轴slice数为128层;厚层数据的层厚比较厚,z轴slice数就相对较少,比如脑平扫CT一般层厚为5mm,z轴slice数在20-40层不等。这样显而易见,薄层的数据相较于厚层数据,在z轴方向的信息更加丰富。
3D卷积可以对3D数据从(x,y,z)三个方向上进行编码,而2D卷积只能对3D数据从(x,y)两个方向进行编码,这是3D卷积的优点。一般来讲,3D卷积的参数量更大,所以我们常用的3D-UNet都不是像2D-UNet那样降采样16倍,而是降采样8倍。但是由于数据量和模型参数量的匹配问题,3DUNet可能需要更多的数据去训练,否则可能会导致过拟合(over-fitting)。
有些病理在2D上没有明显征象,3D就会比较合适。比如动脉瘤的检测,由于血管和动脉瘤在CTA上都是高信号,无法根据灰度值去判断动脉瘤。而是根据3D上血管的走向,发现3D形状上的一些异常,从而进行动脉瘤的诊断。这个病症的特点决定了,从任务背景上2D网络大概率不如3D网络。 有些病理在三维上是稀疏的。如果是在3维上比较稀疏的影像,可能2D网络就要更好。3D网络显存占用很大,这导致不可能将整个3D体素作为输入,必须做crop,裁成一系列3D patch作为输入。裁块会限制网络所能达到的最大感受野,导致丢失一定的全局信息,如果待分割目标本身比裁的块大很多的情况下,网络难以学习目标的整体结构信息。
3D网络适用于coarse to fine的两级分割结构,用于小目标的分割(参考脑肿瘤分割的论文)。第一级用检测网络,或者下采样过的图像做粗定位选bounding box,第二级裁剪出bounding box作为3d网络的输入。
因此,首先需要根据实际问题(临床病理和数据特点),去设计2D或者3D的网络。其次general的结论是,数据量比较小,3维数据的z轴slice数比较少时,2D网络可能更好。数据量比较大,3维数据的z轴slice数比较多时,3D网络可能更好。
2D U-Net:
数据量比较小,3维数据的z轴slice数比较少
病理在三维上是稀疏的,2D网络更好
3D U-Net:
数据量比较大,3维数据的z轴slice数比较多时
专注于细节和局部特征,用于小目标的分割(参考脑肿瘤分割的论文)
病理在2D上没有明显征象,3D有。
