代码实现如下:
data3.3<-read.csv("C:/Users/Administrator/Desktop/data3.3.csv",head=TRUE)
datas<-data.frame(scale(data3.3[,1:6]))
pr3.3<-princomp(~x1+x2+x3+x4+x5,datas,cor=T)
# 对5个变量做主成分分析,其中cor=T表明是用相关系数矩阵进行主成分分析
summary(pr3.3,loadings=TRUE) # 输出主成分分析的结果
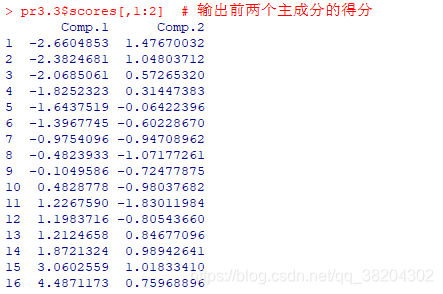
pr3.3$scores[,1:2] # 输出前两个主成分的得分
输出结果为:

summary()的输出结果中Inportance of components部分第一行是5个主成分的标准差,即主成分所对应的特征跟的算术平方根
;第二行是各主成分方差所占的比例,反映了主成分所能解释数据变异的比例,也就是包含原数据信息的比例;第三行是累积比例。
第一个主成分Comp.1的方差百分比为79.826%,含有原始5个变量近80%的信息量;前面两个主成分累积百分比为98.468%,几乎包含了5个变量的全部信息,因此取前两个主成分已经足够。
另外,Loadings部分输出的矩阵为各主成分表达式中
的系数,其中空白部分为默认的为输出的<0.1的值。
现在,我们仅保留前两个主成分对其进行最小二乘回归。
代码实现如下:
pre3.3<-pr3.3$scores[,1:2] # 将前两个主成分的得分保存在变量pre3.3中
datas$z1<-pre3.3[,1] # 将第一主成分的得分添加在数据框datas中,变量名为z1
datas$z2<-pre3.3[,2] # 将第二主成分的得分添加在数据框datas中,变量名为z2
pcr3.3<-lm(y~z1+z2-1,datas)
summary(pcr3.3)
输出结果为:
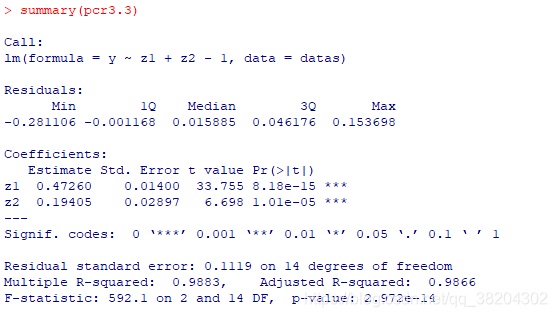
由输出结果可以知道,主成分的回归方程为:
由于主成分是标准化后自变量的线性组合,如果想要得到
关于自变量
的回归方程,只需要分别将下面两个式子
代入上式即可。