Pulse Sequence Resilient Fast Brain Segmentation
抽象
从T1加权(T1-w)磁共振图像(MRI)精确自动分割大脑解剖结构一直是神经成像管道中计算密集的瓶颈,通过基于无监督强度建模的方法和多种方法获得最先进的结果-atlas注册和标签融合。随着基于强大的监督卷积神经网络(CNN)的学习算法的出现,现在可以在几秒钟内产生高质量的脑分割。然而,这些方法的监督性很强,因此难以将它们推广到与训练过程不同的数据上。现代神经影像学研究必然是具有多种采集方案的多中心计划。尽管协议协调实施严格,但不可能将影响图像对比度的扫描仪,场强,接收线圈等的整个MRI成像参数标准化。在本文中,我们提出了一种基于CNN的分割算法,该算法除了高度准确和快速外,还能够适应输入T1 -w采集的变化。我们的方法依赖于构建T1 -w脉冲序列的近似正演模型,其产生典型的测试图像。我们使用正向模型来使用特定于测试数据的训练示例来增强训练数据。这些增强数据可用于更新和/或构建更健壮的分割模型,其更适合于测试数据成像属性。我们的方法在几秒钟内生成高度准确的,最先进的分割结果(整体Dice重叠= 0.94),并且在各种协议中都是一致的。
1简介
全脑分割是神经图像处理管道中最重要的任务之一。分割输出由白质,皮质和皮质下结构的标记组成,例如丘脑,海马,杏仁核等。依赖于体积分割的结构体积和形状统计数据经常用于量化健康人群和患病人群之间的差异[3]。理想的分割算法当然需要高度准确,但是,关键的是,它需要对输入数据的变化具有鲁棒性。大型现代MRI数据集必然是多中心计划,以获得更多的主题。由于扫描仪制造商,场强,接收线圈,脉冲序列,可用对比度和分辨率的变化,很难在不同的站点实现完美的采集协调。场地特定参数的变化引入偏差并增加下游图像处理的变化,包括分割[5,7]。低计算负荷是分割算法的另一个期望特性。快速的记忆光分割算法可以更快地处理大型数据集并广泛采用。
现有的分割算法可大致分为三种类型:(a)无监督,(b)基于多图谱登记,以及(c)监督。无监督算法[3,12]将输入图像的观察强度拟合到基于图谱的基础模型,并执行最大后验标记。它们假定强度分布的函数形式(例如高斯分布),并且如果输入分布不同于该假设,则结果可能降级。已经努力开发混合方法[10],其对输入序列是鲁棒的并且还利用手动标记的训练数据。无监督方法通常是计算密集型的,需要0.5-4小时才能运行。多图册配准和标签融合(MALF)算法实现了最先进的[1,14]分割精度。然而,它们需要多个计算上昂贵的注册,然后是标签融合。如果测试图像对比度属性与图谱图像显着不同,则注册质量也会受到影响。最近,随着医学成像中深度学习方法的成功,基于3D CNN的监督分割方法产生了精确的分割,运行时间为几秒到几分钟[8,13]。尽管这些模型提供了强大的局部和全球背景,但它们容易受到多扫描仪MRI研究中不可避免的微妙对比差异的影响。但是,通过使用测试数据特定增强的适当培训,我们将在Sect中展示。 3,这些差异可以基本上消除。
我们提出了PSACNN脉冲序列增强卷积神经网络; 基于CNN的多标签分割方法,其采用增强方案,即在运行中生成训练图像块,看起来好像它们已经使用测试数据的脉冲序列成像。 PSACNN训练包括三个主要步骤:(1)估计测试数据脉冲序列参数,(2)将测试数据脉冲序列正演模型应用于训练数据核磁共振(NMR)参数,以创建测试数据特定训练特征,(3)训练 深度CNN使用增强来预测分割。 我们将在Sect中详细描述每个步骤。2。
2方法
设A = {A(1),A(2),...,A(M)}是M T1 -w图像的集合,具有相应的专家手动标记图像集Y = {Y(1),Y(2) ,... Y(M)}。配对集合{A,Y}被称为图集图像集或训练图像集。我们假设使用脉冲序列ΓA获取A,其中ΓA可以是MPRAGE(磁化准备梯度回波)[9]或FLASH(快速低角度拍摄),SPGR(损坏梯度回波)等。另外,令B = {ββ(1),ββ(2),...,ββ(M)}为相应的NMR参数图。对于每个i∈{1,...,M},我们得到ββ(i)= [ρ(i),T1(i),T2(i)],其中ρ(i)是质子密度图和T1( i)和T2(i)分别存储纵向(T1)和横向(T2)弛豫时间图。大多数图集数据集不会获取或生成ββ图。我们使用先前采集的数据集[4]使用图像合成为我们的图集数据生成它们,该数据集估计来自多回波FLASH图像的ρ和T1图。我们将在补充材料中描述如何使用这些地图补充现有的地图集。
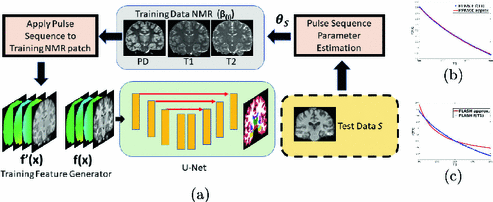
图。1。
(a)用于训练的测试数据特定增强的工作流程,(b)在MPRAGE方程(蓝色)和我们的近似(红色)中的T1分量的拟合,(c)T1分量(蓝色)与我们的近似(红色)的拟合 对于FLASH。
步骤1:估计测试数据采集参数:设S是我们想要通过脉冲序列ΓS获得的分割的测试图像。 我们假设在训练之前我们可以访问测试图像S(或测试数据集),以便我们可以设计特定于测试数据的扩充。 我们想直接从图像S估计ΓS的脉冲序列参数。体素x处的强度S(x)由ΓS的成像方程给出。 等式(1)示出了FLASH序列的成像方程。 S(x)是采集参数重复时间(TR),回波时间(TE),翻转角(α),增益(G)和组织核磁共振参数ββ(x)= [ρ(x),T1的函数 (x),T * 2(x)]。

MPRAGE序列对于模型[15]来说更复杂。通常,难以导出大多数脉冲序列的成像方程,并且给定ββ(x)和采集参数集,有必要运行完整的布洛赫方程模拟以获得体素强度。另外,在现代MRI扫描仪上可能影响信号强度的扫描仪参数的数量非常大,并且对于许多数据集,可能无法获得详细的参数集。因此,我们想直接从图像强度估计这些。然而,通常不可能仅从图像强度本身鲁棒地估计所有脉冲序列参数。即使当方程式被充分理解时,强度对成像参数的高度非线性依赖性使得它们的估计不稳定。因此,我们已经制定了具有较少数量参数的MPRAGE和FLASH方程的近似值,这些参数可以从图像强度直接且稳健地估计。我们对FLASH序列的近似表示在方程式中。 (2) - (3)。

其中θθFLASH= {θ0,θ1,θ2}构成我们的参数集。 对于1.5T时人脑中T1值(500,3000)ms的范围,我们的近似拟合T1井的信号依赖性(见图1(b)和1(c))。 等式(4)是我们对Wang等人提供的MPRAGE成像方程的近似。[15]。
![]()
给定测试图像S,我们通过类似于Jog等人的策略来估计θθS。[6]。 设Sc,Sg,Sw分别为脑脊液(CSF),灰质(GM)和白质(WM)的平均强度。 这些可以通过将高斯混合模型拟合到强度分布来通过三类分类方案获得。 设ββc,ββg,ββw分别是CSF,GM和WM类的平均NMR值,它们是从先前获得的数据中获得的[4]。 因此,我们有一个三个线性方程组

第2步:特征提取和增强:我们使用U-Net CNN架构[11]作为我们的预测器。 GPU内存限制阻止我们使用256×256×256脑图像作为输入。对于A中的每个A(i),我们在体素x处采样32×32×32尺寸的贴片p(x)。我们观察到,训练纯粹基于补丁的U-Net会导致由于不正确的本地化导致的分段错误。为了提供更多的全局上下文,我们使用FreeSurfer管道[4]产生的非线性扭曲信息,将FreeSurfer图集与测试图像S对齐。对于S中的每个体素x,我们提取FreeSurfer图集3D坐标w(x) ,扭曲到x,并产生wp(x)-a 32×32×32×3的扭曲的FreeSurfer图集坐标。最终特征向量f(x)= [p(x),wp(x)],是强度补丁和坐标的串联(参见图1(a)所示的2D补丁)。通过从第i个NMR图中提取每个i∈{1,...,M},ββ(i)∈B的NMR参数补片来生成测试数据特定的增强补片,其中体素x处的NMR补片由[ρρ(i)表示。 )(x),T1(i)(x),T2(i)(x)]。增强补丁由下式给出:
![]()
此外,我们还对θθS的3D参数空间进行采样,并生成非测试数据特定的增强补丁,以增加可用训练的广度。 增强的补丁也与扭曲的FreeSurfer atlas坐标补丁wp(x)连接,以生成增强特征f'(x)= [p'(x),wp(x)]并添加到训练中(图1( 一个))。
具有相应的手动标记图像块的原始和增强特征用于训练U-Net,其具有三个池化和相应的上采样阶段(参见图1(a))。每个橙色块由两个3D卷积层组成,后跟批量标准化层。第一个块具有32个3×3×3大小的滤波器,后续编码块的滤波器数量是前一个块的两倍。红色箭头表示跳过连接,其中来自编码层的数据被连接作为解码层的输入。除了具有softmax激活的最后一层之外,所有激活都是ReLu。输出为32×32×32×L,其中L = 44是标签数。我们使用Adam优化算法来最小化在所有标签上计算的基于软件Dice的损失。批量大小为32,并在增强和原始训练功能之间平均分配。未增强训练的总大小是来自A的16名受试者的106个补丁。验证数据来自三个科目。在预测期间,我们从测试图像S中提取特征并应用训练的U-Net以生成标签概率的重叠片。通过在重叠区域中求平均将它们组合在一起以产生最终概率图。选择具有最高概率的标签作为硬分割中的标签。
3 Experiments and Results
3.1:相同的扫描数据集。 在本实验中,我们使用与训练数据相同的采集协议,在测试数据上比较PSACNN与未增强CNN(CNN),FreeSurfer分割(ASEG)[3],SAMSEG [10]和MALF [14]的性能。 完整的数据集由39名受试者组成,具有1.5 T Siemens Vision MPRAGE采集(TR = 9.7 ms,TE = 4 ms TI = 20 ms,体素大小= 1×1×1.5 mm 3),根据协议完成专家手册标签 在[2]中描述。 我们选择了16个受试者的子集作为CNN,PSACNN的训练数据,以及作为MALF的图集。 我们使用3个主题来验证CNN,20个用于测试。 图2显示了所有算法的结果以及PSACNN生成的示例分段。 对于大多数结构,CNN(红色)和PSACNN(蓝色)具有显着高于使用配对t检验(p <0.01)测试的下一个最佳方法的骰子重叠(ALL Dice = 0.9376)。
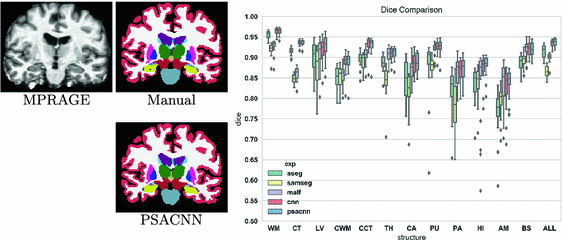
图2。
缩略语:白质(WM),皮质(CT),侧脑室(LV),小脑白质(CWM),小脑皮质(CCT),丘脑(TH),尾状核(CA),壳核(PU),苍白球(PA) ),海马(HI),杏仁核(AM),脑干(BS),所有结构重叠(ALL)。
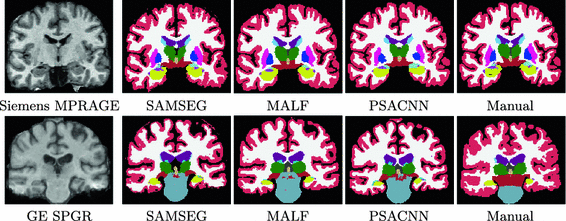
图3。
使用SAMSEG,MALF和PSACNN的分段结果以及手动分段输入Siemens和GE采集。
3.2:不同的扫描数据集。在本实验中,我们比较了两个数据集上PSACNN与其他方法的准确性; (a)西门子数据集:在1.5T Siemens SONATA扫描仪上采集的14个主题MPRAGE扫描,其脉冲序列与训练数据相同;(b)GE数据集:在1.5T GE上采集的13个主题SPGR(损坏的梯度调用)扫描Signa(TR = 35ms,TE = 5ms,α=45∘,体素尺寸= 0.9375×0.9375×1.5mm 3)扫描仪。两个数据集都具有使用与训练数据相同的协议生成的专家手动分段。图4显示了所有方法的Dice系数的比较。在西门子数据集CNN和PSACNN明显优于其他数据(图4(a)),正如预期的那样,由于它与训练数据的相似性。然而,在GE扫描显示出明显不同的组织对比度(图3),所有方法显示重叠减少(图4(b)),但CNN具有最差的性能,因为它无法概括。另一方面,PSACNN(所有骰子= 0.7636)对于由于基于脉冲序列的增强引起的对比度变化是稳健的,并且产生与诸如SAMSEG(所有骰子= 0.7708)的最新算法相当的分割。 )和MALF(所有骰子= 0.7804)的准确性,处理时间减少一个数量级。
3.3:多扫描仪一致性。 在这个实验中,我们测试了从15个受试者获得的四个数据集上各种方法产生的分割的一致性; MEF:在Siemens 3 T TRIO扫描仪上获得的多回波FLASH,TRIO:MPRAGE在西门子TRIO上获得,GE:MPRAGE在1.5 T GE Signa上获得,西门子:MPRAGE在1.5 T Siemens SONATA上获得。 西门子扫描参数最接近训练数据集的参数,我们计算了在数据集MEF,TRIO,GE上使用不同分割方法获得的结构体积的绝对差异。 在表1中,我们仅报告由于空间不足导致的WM体积差异,并且还因为总白质体积是脉冲序列变化累积效应的良好指标。 MALF表现出最高的GE一致性,其余为第二好。 PSACNN为TRIO提供最佳性能,为GE数据集提供第二好的性能。
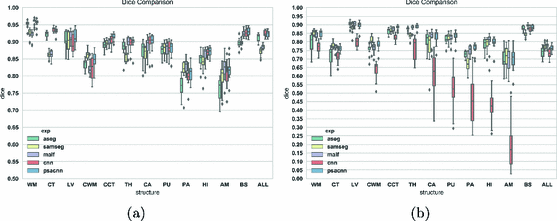
图4。
关于(a)西门子数据集,(b)GE数据集的骰子评估。 对于首字母缩略词,请参见图2标题。
Tú 4.
表格1。
数据集MEF,TRIO,GE与西门子之间针对不同算法的绝对WM体积差异的平均值和标准偏差(标准偏差)。

4讨论和结论
我们已经描述了PSACNN,一种基于CNN的分割算法,其可以通过使用MRI图像形成的近似正演模型生成测试数据特定增强来使其自身适应测试数据。 增强可用于稳健地训练或微调任何潜在的预测器。 我们在不同的数据集上展示了最先进的分割性能。 预测速度很快,在单个进程上运行不到一分钟。 在未来,我们打算使用更精确的成像方程并模拟其他脉冲序列以增加应用范围。