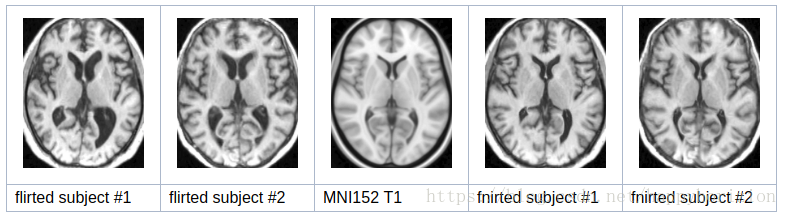
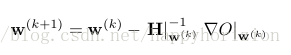非线性配准 fnirt
为了排除个体差异, 进而使得个体间脑组织的对比得以实施, 常需要将不同的大脑配准到公共的模板/空间. 这样做是为了后续用TBSS等统计方法进行组间分析(compare fractional anisotropy or tissue composition). 配准算法根据适用的问题不同分为线性和非线性两种, flirt采用了线性配准算法, 所谓的线性配准就是采用坐标旋转\平移\缩放\剪切将两个图像匹配到一起.但是线性配准也有局限性,很多时候仅仅用线性配准是不够的. 下面的例子就展示了线性配准和非线性配准后的结果:
算法建模:
1) 仿射变换
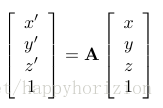
其中A是一个4*4的矩阵where A is a 4x4 matrix that is uniquely defined by 12 values (3 translations, 3 rotations, 3 zooms and 3 shears).
加上非线性修正部分:
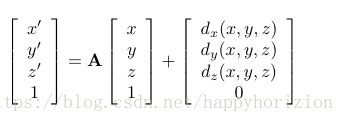
在fnirt算法中, d_i(x,y,z)是四阶或者三阶B样条曲线.
2) 代价函数:
fsl的代价函数表示为二次方求和的形式. 其中f()指参考图像(例如MNI152模板), g()指需要配准到参考图像上的原始图像. 代价函数的这个形式在某种程度上也约束了fnirt的使用条件,那就是g()和f()必须要比较接近才行. 如果代价函数采用了correlation-ratio或者互信息这类对图像之间的对比差异不那么敏感的函数, 就可以放宽这个适用条件.
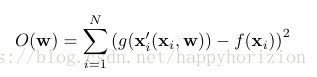
但是这种”最小二乘”形式的代价函数, 在最优化的时候比较有优势. 另外, fnirt选项: 调整图像密度参数f可以在全局和局部同时调节寻优搜索空间, 从而解决了对比度差异的问题, 使得算法在f()和g()不那么接近时也具备一定的鲁棒性.
3) 优化算法
配准问题(不管是线性还是非线性)实际上都是一个非线性优化问题, 所谓的非线性优化问题是指目标函数或者代价函数是待优化变量w的非线性形式. 如何寻找到使得目标函数最小的w有很多方式. 有些算法中需要计算目标函数的一阶, 甚至二阶导数. 下式给出Gauss-Newton算法的寻优迭代形式, 中H和 分别代表目标函数的Hessian矩阵和梯度(Jacobian矩阵). Gauss-Newton算法在迭代过程中保证每个迭代步都朝着局部最优化的方向前进,并且可以保证比较快的收敛速度.
fnirt在Gauss-Newton迭代中采用了Levenberg-Marquardt修正, 并且在算法设计上保证了Hessian矩阵计算的效率.
4) “微分同胚”的概念
从空间U到空间V之间微分同胚隐射是说空间U中的点与空间V中的点肯定是一一对应的, 而且这种映射关系还是可逆的. 除此之外, 这种映射的Jacobian矩阵在每个位置处都是大于零的. 这一点在非线性配准运算中特别重要.
fnirt命令行调用方法
fnirt命令行基本的调用方式是:
fnirt –ref=target_image --in=input_image但是这样简单的命令,肯定不可能得到期望的结果,因为fnirt需要用户指定要做什么,怎么做. 如果没有指定这些参数, 运算结果肯定不好. fnirt支持很多设置选项, 而且这些选项之间是有联系的. 这让fnirt的使用有些复杂, 要求用户比较了解非线性配准的具体原理. 为了降低调用难度, 官方提供了一些配置文件, 初级用户可以直接调用配置文件, 不用逐一设置选项参数.
1) 配置文件
配置文件中包含了fnirt可以设置的选项参数, 配置文件的文件名可以通过—config参数传递, 要求是ascii格式的文件, 要求每行一个参数, 例如:
# name of reference image 模板, 全脑,包括颈部
--ref=/usr/local/fsl/data/standard/MNI152_T1_2mm.nii.gz然后将这个文件保存为 my_config.cnf, fnirt命令行调用, 如下命令:
fnirt --in=my_brain --config=my_config --warpres=6,6,6如果用户需要自定义config文件, 比较好的方式是从官方的例子入手, 然后逐渐根据自己的情况修改参数以适应需求. 如果问题特别具有普遍性, 还可以直接和官方邮件沟通,他们会给出一些配置方面的建议.
下面给出官方提供的cnf文件T1_2_MNI152_2mm.cnf , 在FSL安装路径下的src/fnirt/fnirtcnf:
# =======FSLDIR/src/fnirt/fnirtcnf/T1_2_MNI152_2mm.cnf=====
# name of reference image
# --ref=/usr/local/fsl/data/standard/MNI152_T1_2mm.nii.gz
--ref=MNI152_T1_2mm
# name of file with mask in reference space
# --refmask=/usr/local/fsl/data/standard/MNI152_T1_2mm_brain_mask_dil.nii.gz
--refmask=MNI152_T1_2mm_brain_mask_dil
# If =1, use implicit masking based on value in --ref image. Default =1
--imprefm=1
# If =1, use implicit masking based on value in --in image, Default =1
--impinm=1
# Value to mask out in --ref image. Default =0.0
--imprefval=0
# Value to mask out in --in image. Default =0.0
--impinval=0
# sub-sampling scheme, default 4,2,1,1
--subsamp=4,4,2,2,1,1
# Max # of non-linear iterations, default 5,5,5,5
--miter=5,5,5,5,5,10
# FWHM (in mm) of gaussian smoothing kernel for input volume, default 6,4,2,2
--infwhm=8,6,5,4.5,3,2
# FWHM (in mm) of gaussian smoothing kernel for ref volume, default 4,2,0,0
--reffwhm=8,6,5,4,2,0
# Weigth of membrane energy regularisation, default depending on --ssqlambda and --regmod switches. See user documetation.
--lambda=300,150,100,50,40,30
# Estimate intensity-mapping if set, deafult 1 (true)
--estint=1,1,1,1,1,0
# Apply the mask if set, default 1 (true)
--applyrefmask=1,1,1,1,1,1
# Apply the mask if set, default 1 (true)
--applyinmask=1
# (approximate) resolution (in mm) of warp basis in x-, y- and z-direction, default 10,10,10
--warpres=10,10,10
# If set (=1), lambda is weighted by current ssq, default 1
--ssqlambda=1
# Model for regularisation of warp-field [membrane_energy bending_energy], default bending_energy
--regmod=bending_energy
# Model for intensity-mapping [none global_linear global_non_linear local_linear global_non_linear_with_bias local_non_linear]
--intmod=global_non_linear_with_bias
# Order of poynomial for mapping intensities, default 5
--intorder=5
# Resolution (in mm) of bias-field modelling local intensities, default 50,50,50
--biasres=50,50,50
# Weight of regularisation for bias-field, default 10000
--biaslambda=10000
# If =1, ref image is used to calculate derivatives. Default =0
--refderiv=0
# ==============end===============2) 参数列表:
输入参数
–ref=filename 模板文件名称, 例如MNI152_T1_2mm.nii.
–in=filename 要配准的NifTI文件
–config=config_file 配置文件, 文件类型为.cnf
–aff=mat_fname 初始猜测的仿射变换矩阵. 通常是flirt命令的输出结果.
–inwarp=coef/filed_fname 非线性优化迭代的初始猜测. 可以是之前fnirt计算得到的warp
–intin=fname_of_file_with_global/local_intensity_mapping 还是之前fnirt计算的结果,用于进一步优化的.描述intensity_mapping文件名称
–refmask=filename 目标FOV的mask文件名. 例如MNI152_T1_2mm_brain_mask_dil.nii
–inmask=filename 目标FOV的mask文件, 这里是用户自生成的mask文件
输出参数
–cout=filename 定义warp文件的系数
–iout=filename 输入NIfTI配准到模板上的结果
–fout=filename 输出的warp文件
–jout=filename 输出Jacobian文件. 对后续分析(diagnosis)和VBM有用
–refout=filename 模板经过intensity modulation后的输出, 对后续diagnosis有用.
–intout=filename 输出文件, 内含intensity modulation信息
全局设置参数 parameter that are specified “once for all”
–warpres=xres,yres,zres 指定warp文件的精度(单位是mm), 例如10, 10, 10
–splineorder=n B样条的次数(order), n=2或者3
–imprefm(=1/0) 如果设置为1, 算法将认为在实际的FOV外侧还有1个体素. 通常保留默认值
–impinval(=1/0) 类似上一条设置, 如果是1的话认为输入文件在FOV外侧再多取1个体素. 通常保留默认值.
–ssqlambda=0/1, lambda是否需要乘上差值的平方和. 和内部算法直接相关的一个设置. 最好保留默认值.
–regmod=model 设置算法中用的正则化模型 (regularisation-model), 例如bending_energy
–regderiv 设置是否需要根据模板来计算梯度.
–intmod=model 指定要用到的intensity-nomalisation模型
–intoder=n 指定intensity modulation用到的多项式阶数
不同阶段降采样参数
–subsamps=level1, level2, … 配准操作时降采样阶数, 例如4, 2, 1
–miter=level1, level2, … 每一层的迭代次数,例如5,5,10
–reffwhm=level1, level2, … 每一层对模板的平滑操作, 例如8,4,0
–infwhm=level1, level2, … 每一层对输入图像的平滑, 例如10,6,2
–lambda=level1, level2,… 每一层对于差值平方和的相对加权以及正则化设置, 例如300,75, 30
–estint=level1, level2, …设置每层的intensity模型是否需要estimate.
–applyrefmask=level1, level2, …设置每层是否需要mask参考空间, 例如1,1,0
–applyinmask=level1, level2, …设置每层是否需要mask用户自定义参考空间,例如0,0,1
下面给出一个原图\模板和配准后大脑NifTI的对比图, 图中灰白色的原NifTi, 蓝色透明及其背景的灰白色不透明图像分别是配准后的图像和参考的MNI152图像. FSL的registration效果还是不错的.