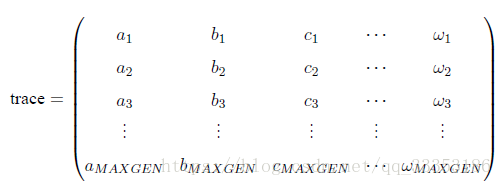上一篇讲了Geatpy的快速入门:https://blog.csdn.net/qq_33353186/article/details/82014986
但是光是几个例子是远远不能熟练掌握python遗传和进化算法编程的,得进一步了解其原理以及API。
Geatpy是简单封装的开放式进化算法框架,可以方便、自由地与其他算法以及实际项目相结合。其层次结构图如下:
其中函数接口是自定义的目标函数以及罚函数(可以没有罚函数),比如要解决一个约束优化问题,那么一般把目标函数写在aimfuc.py里(文件名自定义),把约束条件写成一个罚函数放在punishing.py里(文件名自定义)。当然有时候也可以出于方便把约束条件直接写在目标函数里面。
上图的”编程模板“即为解决实际优化问题而编写的模板函数。比如实现了一个自适应的遗传算法,那么就可以把算法流程全部写在编程模板里面。Geatpy自带许多已完善的编程模板,用于解决常见的单目标和多目标优化问题。在后面我们也会详细讲解这种模板怎么编写。
上图的”高级操作函数“和”低级操作函数“就是进化算法的基本算子。比如适应度计算算子、选择算子、重组和交叉算子、变异算子、重插入算子等等。这些算子被写成了Geatpy的库函数,详细API文档和案例可以参见https://github.com/geatpy-dev/geatpy/tree/master/geatpy/doc
这些库函数具体有:
1. 初始化种群
- crtbp(创建简单离散种群、二进制编码种群)
- crtip(创建整数型种群)
- crtpp(创建排列编码种群)
- crtrp(创建实数型种群)
2. 进化迭代相关函数
这部分是在进化迭代过程中要用到的:

- ranking、scaling、indexing、powing:计算种群个体的适应度。
- selecting :选择操作(它将调用低级选择函数)。
- recombin :重组操作(它将调用低级重组函数)。
- mutate :变异操作(它将调用低级变异函数)。
- reins:重插入,把育种个体重插入到父代,生成新一代种群。
- migrate:多种群下的种群迁移(可以增加种群的多样性)。
3. 适应度计算
- ranking(基于等级划分的适应度分配计算)
- scaling(线性尺度变换适应度计算)
- indexing(指数尺度变换适应度计算)
- powing(幂尺度变换适应度计算)
4. 选择
selecting 是高级选择函数,它调用下面的低级选择函数:
- sus(随机抽样选择)
- rws(轮盘赌选择)
- tour(锦标赛选择)
5. 重组(包括交叉)
交叉是重组的一部分。
- recdis(离散重组)
- recint(中间重组)
- reclin(线性重组)
- xovdp(两点交叉)
- xovdprs(减少代理的两点交叉)
- xovmp(多点交叉)
- xovpm(部分匹配交叉)
- xovsh(洗牌交叉)
- xovshrs(减少代理的洗牌交叉)
- xovsp(单点交叉)
- xovsprs(减少代理的单点交叉)
6. 突变
mutate 是高级的突变函数,它调用下面的低级突变函数:
- mut(简单离散变异算子)
- mutbga(实数值变异算子)
- mutbin(二进制变异算子)
- mutgau(高斯突变算子)
- mutint(整数值变异算子)
- mutpp(排列编码变异算子)
7. 重插入
reins 是重插入函数,它将育种个体重插入到父代种群中,生成新一代种群。
8. 种群迁移
当使用多种群设计时,可用migrate 函数实现种群中的个体迁移。
9. 染色体解码
对于二进制/格雷编码的种群,我们要对其进行解码才能得到其表现型。
- bs2int(二进制/格雷码转整数)
- bs2rv(二进制/格雷码转实数)
10. 数据可视化
- trcplot(单目标进化跟踪器绘图)
- frontplot(多目标优化帕累托前沿绘图函数)
11. 多目标相关
- awGA(适应性权重法多目标聚合函数)
- rwGA(随机权重法多目标聚合函数)
- ndomin(简单非支配排序)
- ndomindeb(Deb 非支配排序)
- ndominfast(快速非支配排序)
- upNDSet(更新帕累托最优集)
12. 模板相关
- sga_real_templet(单目标编程模板(实值编码))
- sga_code_templet(单目标编程模板(二进制/格雷编码))
- sga_permut_templet(单目标编程模板(排列编码))
- sga_new_real_templet(改进的单目标编程模板(实值编码))
- sga_new_code_templet(改进的单目标编程模板(二进制/格雷编码))
- sga_new_permut_templet(改进的单目标编程模板(排列编码))
- awGA_templet(基于适应性权重法(awGA) 的多目标优化编程模板)
- i_awGA_templet(基于交互式适应性权重法(i-awGA) 的多目标优化编程模板)
- nsga2_templet(基于改进NSGA-Ⅱ 算法的多目标优化编程模板)
- q_sorted_templet(基于快速非支配排序法的多目标优化编程模板)
详细API文档和案例可以参见https://github.com/geatpy-dev/geatpy/tree/master/geatpy/doc
下面讲一下Geatpy中重要的数据结构:
Geatpy 的大部分数据都是存储在numpy 的array 数组里的,numpy 中另外还有matrix的矩阵类型,但Geatpy不使用它,于是可以默认array 就是“矩阵”(也可以存储一维向量,接下来会谈到)。
其中有一些细节需要特别注意:numpy 的array 在表示行向量时会有2 种不同的结构,一种是1 行n 列的矩阵,它是二维的;一种是纯粹的一维行向量。因此,在Geatpy 教程中会严格区分这两种概念,我们称前者为“行矩阵”,后者为“行向量”。Geatpy 中不会使用超过二维的array。例如有一个行向量x,其值为1 2 3 4 5 6,那么,用print(x.shape) 输出其规格,可以得到(6,),若x 是行矩阵而不是行向量,那么x 的规格就变成是(1,6) 而不再是(6,)。
在编程中,如果对numpy 的array 感到疑惑,你可以用” 变量.shape” 语句来输出其维度信息,以确定其准确的维度。
- 种群染色体的数据结构:
Geatpy 中,种群染色体是一个二维矩阵,简称“种群矩阵”。一般所说的“种群”是特指种群染色体矩阵。
种群矩阵一般用Chrom 命名,是一个是numpy 的array 类型的,每一行对应一条染色体,同时也对应着一个个体。染色体的每个元素是染色体上的基因。
一般把种群的规模(即种群的个体数) 用Nind 命名;把种群个体的染色体长度用Lind 命名。其结构如下图所示:
对于多种群,各个子种群被合成在一个大的矩阵中,每个子种群的规模是相同的,结构如下图所示:
比如如下种群:
假设它要表示2 个子种群,那么,前两个个体(前两行) 就是1 号子种群,后两个个体(后两行) 就是2 号子种群。种群个体的染色体为:1234, 2341, 3142 和4231。
- 种群表现型的数据结构:
种群表现型的数据结构跟种群染色体基本一致,也是numpy 的array 类型。我们一般用Phen 来命名(也可以命名为variable)。它是种群矩阵Chrom 经过解码操作后得到的基因表现型矩阵,每一行对应一个个体,每行中每个元素都代表着一个控制变量,并用Nvar 表示控制变量的个数。如下图:
- 目标函数值的数据结构:
Geatpy 采用numpy 的array 类型变量来存储种群的目标函数值。一般命名为ObjV ,每一行对应种群矩阵的每一个个体。因此它拥有与Chrom 相同的行数。每一列代表一个目标函数值。因此对于单目标函数,ObjV 会只有1 列;而对于多目标函数,ObjV 会有多列。例如ObjV 是一个二元函数值矩阵:
其第一列就代表目标函数f1的目标函数值,第二列代表目标函数f2的目标函数值。
- 个体适应度的数据结构:
个体适应度的数据结构与上面的ObjV类似,不过它只有一列。每一行对应种群每个个体的适应度值。
- 区域描述器的数据结构:
区域描述器是用来描述种群染色体的特征,比如染色体中基因所表达的控制变量的范围、是否包含范围的边界、采用什么编码方式,是否使用对数刻度等等。
1) 对于二进制/格雷编码的种群,使用7 行n 列的矩阵FieldD 来作为区域描述器,n是染色体所表达的控制变量个数。FieldD 的结构如下:
其中,lens 包含染色体的每个子染色体的长度。sum(lens) 等于染色体长度。
lb 和ub 分别代表每个变量的上界和下界
codes 指明染色体子串用的是标准二进制编码还是格雷编码。codes[i] = 0 表示第i个变量使用的是标准二进制编码;codes[i] = 1 表示使用格雷编码。
scales 指明每个子串用的是算术刻度还是对数刻度。scales[i] = 0 为算术刻度,scales[i] = 1 为对数刻度。对数刻度可以用于变量的范围较大而且不确定的情况,对于大范围的参数边界,对数刻度让搜索可用较少的位数,从而减少了遗传算法的计算量。
lbin 和ubin 指明了变量是否包含其范围的边界。0 表示不包含边界;1 表示包含边界。
2) 对于实值编码(即前面所说的不需要解码的编码方式) 的种群,使用2 行n 列的矩阵FieldDR 来作为区域描述器,n 是染色体所表达的控制变量个数。FieldDR 的结构如下:
区域描述器FieldD 和FieldDR 都是numpy 的array 类型。可以直接用代码创建,比如:
FieldDR=np.array([[-3, -4, 0, 2],
[2, 3, 2, 2]])也可以用Geatpy 内置的crtfld库函数来方便地生成区域描述器。详细用法请看https://github.com/geatpy-dev/geatpy/blob/master/geatpy/doc/crtfld/crtfld.pdf
- 进化追踪器的数据结构:
在使用Geatpy 进行进化算法编程时,常常建立一个进化追踪器(如pop_trace) 来记录种群在进化的过程中各代的最优个体,尤其是采用无精英保留机制时,进化追踪器帮助我们记录种群在进化的“历史长河”中产生过的最优个体。待进化完成后,再从进化追踪器中挑选出“历史最优”的个体。这种进化记录器也是numpy 的array 类型,结构如下:
其中MAXGEN 是种群进化的代数。trace 的每一列代表不同的指标,比如第一列记录各代种群的最佳目标函数值,第二列记录各代种群的平均目标函数值……trace 的每一行对应每一代,如第一行代表第一代,第二行代表第二代……
- 全局最优集的数据结构:
在使用Geatpy 进行多目标进化优化编程时,常常建立一个全局的帕累托最优集(NDSet) 来记录帕累托最优解。它也是numpy 的array 类型,结构如下:
f、g、h、φ 等表示不同的目标函数值。NDSet 的每一行都是一个帕累托非支配解对应的各个目标函数值。
熟悉Geatpy库函数和数据结构是熟练地进行python进化算法编程的前提。在下一篇,我们将详细讲述如何编写编程模板来实现一个解决约束优化问题的遗传算法: https://blog.csdn.net/qq_33353186/article/details/82021750
欢迎继续跟进,感谢!