Fastq文件的获取
author:CYH-BI
date:2023.10.25
Fastq文件得获取 ,可以从NCBI获取,也可以自己测序得到结果,下面介绍从网上获取数据。
linux 下安装SRA Toolkit工具
2023.3.24
1、NCBI 官网查看工具地址,并下载安装包
具体操作步骤如下:
-
打开NCBI 官网
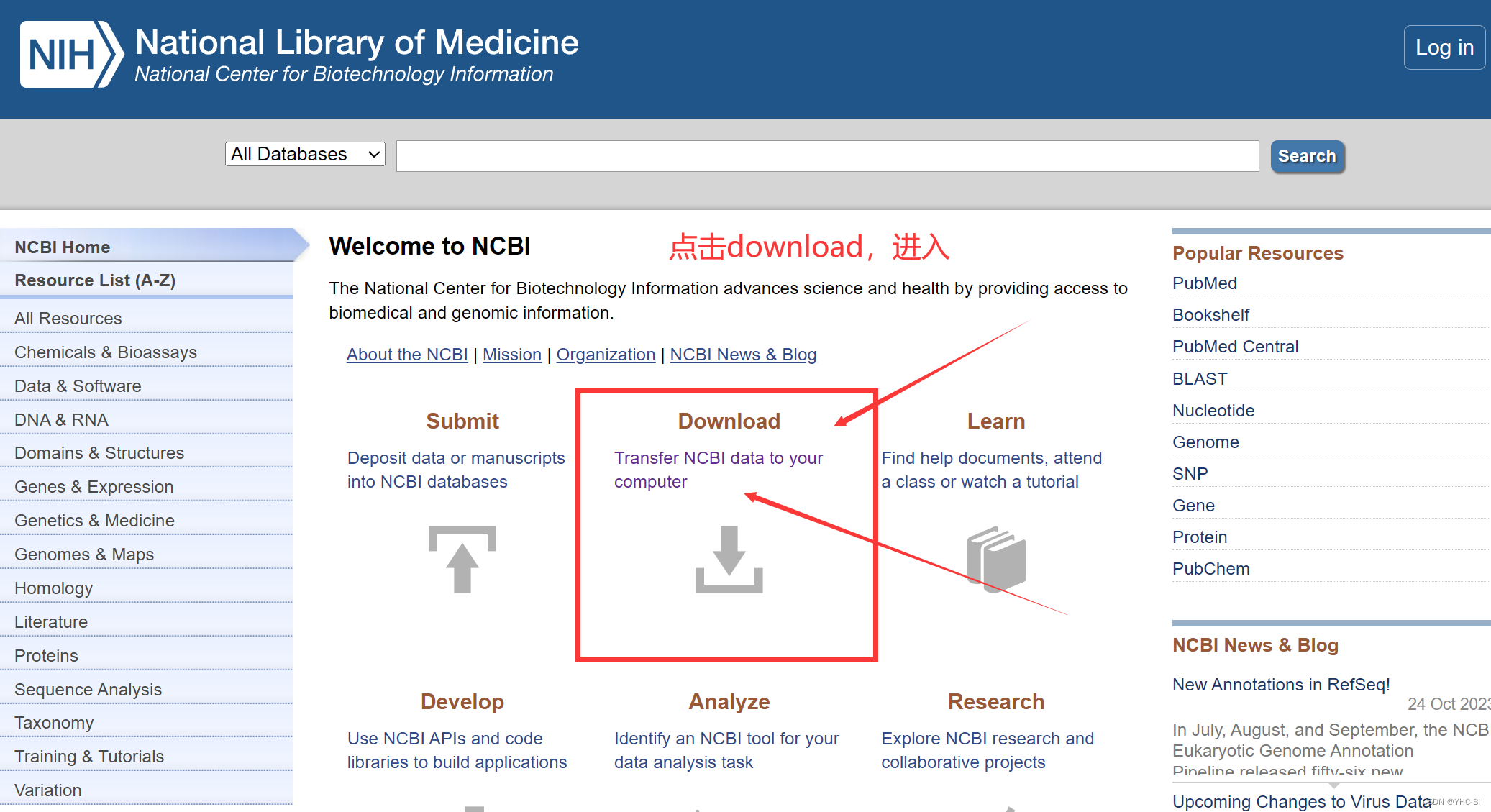
-
点击Download Tools并进入
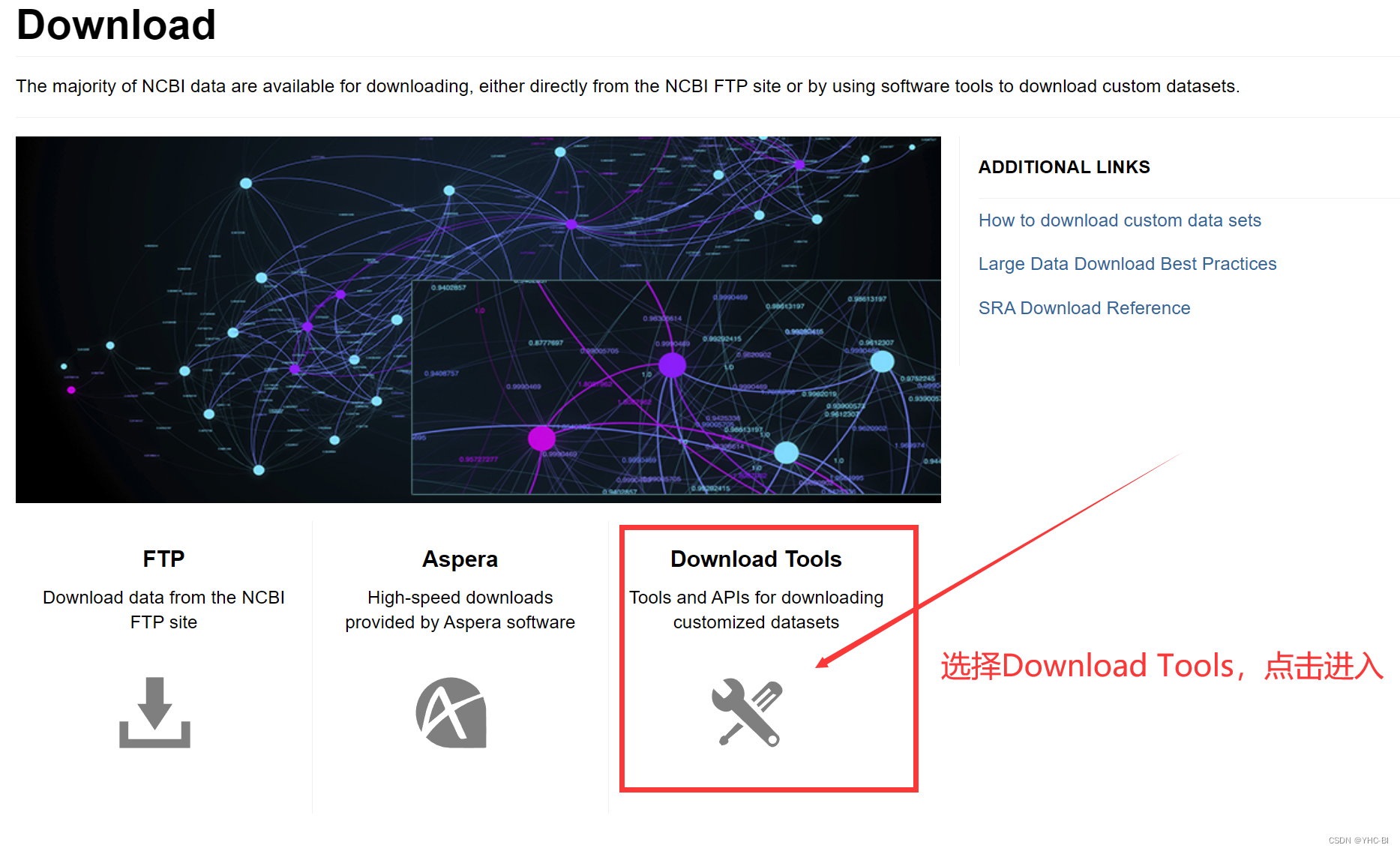
-
找到SRA Toolskit

-
会转跳到github ,复制下载连接,后续使用Linux wget 命令下载
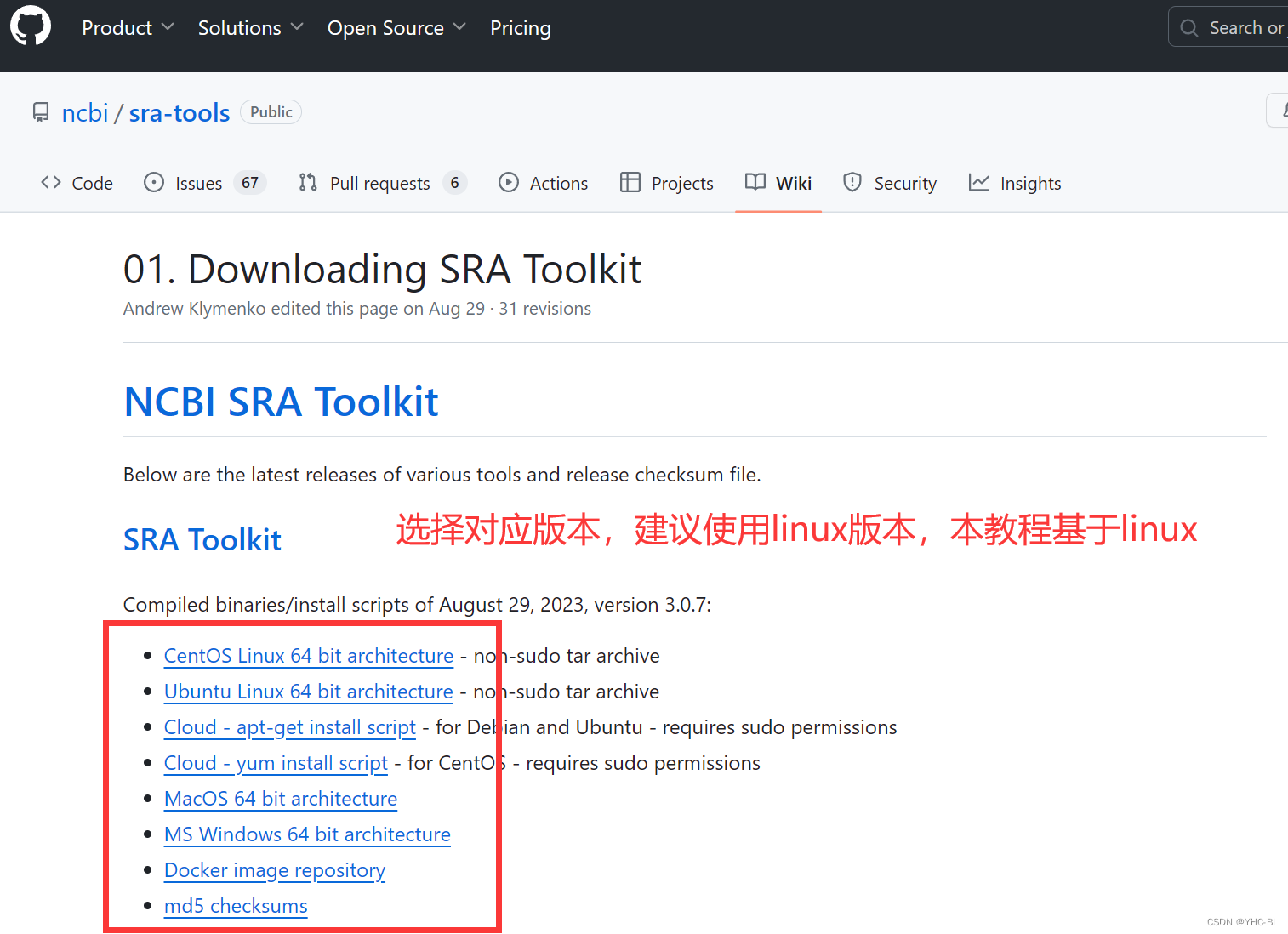
-
打开Linux 总端,使用wget 命令下载
wget https://ftp-trace.ncbi.nlm.nih.gov/sra/sdk/3.0.7/sratoolkit.3.0.7-centos_linux64.tar.gz
2、解压sratoolkit.3.0.7 安装包.。注意包名称,我们的不一定一样(不同版本不一样)
tar xvf sratoolkit.3.0.7-centos_linux64.tar.gz
3、查看有无安装成功(如果软件含有bin目录,大多情况下配置环境时路径需要精确到bin)
cd /home/cyh/sratoolkit/sratoolkit.3.0.7-centos_linux64/bin #看看能不能打开bin
4、配置SRA Toolkit工具环境
nano ~/.bashrc #编辑.bashrc文件 或者是 vim /etc/.bashrc
export PATH="/home/cyh/sratoolkit/sratoolkit.3.0.7-linux64/bin:$PATH" #最后一行输入
#编辑结束后按CTRL+C保存,按提示后按回车
# 格式:export PATH="<路径>:$PATH" ;路径要精确到bin
#注意:你的全路径。是你的,不是我的。
#注意:你的全路径。是你的,不是我的。
#注意:你的全路径。是你的,不是我的。
source ~/.bashrc #重启文件
SRA Toolkit 工具的目的是下载NCBI未处理的SRA数据,还能将SRA文件处理成fastq文件
使用SRA Toolkit工具下载SRA数据
1、使用命令下载感兴趣的数据,SRA Toolkit 工具有个命令可以稳定下载数据,就是有点慢,但是稳定。
[root@localhost sra_data]# prefetch SRR12415658 ##以SRR12415658为例
以SRR12415658数据为例:
建议使用后台下载:(如果是服务器,后台下载不会因为连接中断而中断,除非服务器网断了)
nohup prefetch SRR12415658 &
对于多个数据下载:(方法多种多样,可以自己写循环)
2、下载后,使用fastq-dump拆分sra数据,转变为fastq格式数据
fastq-dump --split-3 SRR12415658.sra
--split-3表示双端测序
加以加上 -O 参数,不加就会下到sra 里面(好像在SRA Toolkit里,忘记了,上次找了好久)。
fastq-dump --split-3 SRR12415658.sra -O /home/cyh/Desktop/sra
将结果输出到 /home/cyh/Desktop/fastq ,(文件你得先有)
3、还需了解其他参数,可以使用 -h参数。比如,如果你需要将文件输出到其他地方,加个 -O参数。
fastq-dump -h
后续可以得到两个fastq文件 ,用于后续FastQC 质控。