上期我们介绍了序列比对中的一些基本概念,包括匹配(match)、不匹配(mismatch)、替换(substitution)、间隔(gap)、插入(insertion)、删除(deletion)、全局和局部对齐(global and local alignments),这期我们来对这些概念进行更为详细的描述。
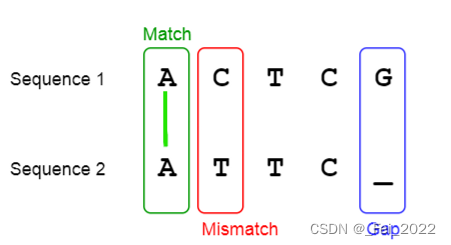
- 匹配(match):指两个序列中的相应位置上的碱基或氨基酸相同的情况。匹配通常用相同的字母或符号表示,如DNA序列中,A与A、C与C、G与G、T与T之间的匹配表示为“|”。
- 不匹配(mismatch):指在两个序列中相应位置上的碱基或氨基酸不同的情况。不匹配通常用不同的字母或符号表示。
- 间隙(gap):指序列中的缺失区域,即一个序列中某个位置缺少了一个碱基或氨基酸。在比对序列时,可以在一条序列中插入一个破折号或其他符号来表示间隙,以便将两条序列对齐。
- 替换(substitution):指在两个序列中相应位置上的碱基或氨基酸发生了不匹配的情况。替换通常用一个字母或符号替换另一个字母或符号表示。
- 插入(insertion):指在一个序列中插入一个或多个碱基或氨基酸的过程,导致与另一个序列不匹配。插入通常用一个字母或符号表示。
- 删除(deletion):指从一个序列中删除一个或多个碱基或氨基酸的过程,导致与另一个序列不匹配。删除通常用一个字母或符号表示。

- 局部对齐(local alignment):是在两个序列中找到一段最佳的匹配区域进行比对,以找到最佳的匹配方案。局部对齐通常用于比对相似性较低的序列。
- 全局对齐(global alignment):是将两个序列的整个长度进行比对,以找到最佳的匹配方案。全局对齐通常用于比对相似性较高的序列。

(虚线以上是局部对齐,下面序列仅和上面序列部分比对,局部序列比对减少了不同序列比对所需的缺口数量,因此能够揭示在全局序列比对中通常检测不到的短保守或相似区域。;虚线以下是全局对齐,两条序列的比对是从头到尾的,也就是比对从一个序列的第一个碱基开始,在另一个序列的最后一个碱基停止比对。)
扫描二维码关注公众号,回复:
16906759 查看本文章


下面我来看两条序列 seq1:ATGAAGCGTGC和seq2:ATGAAGAGTGCA,seq1的长度是11,seq2的长度是12。我们可以如下图一样对两条序列进行对齐,但是这对齐方式只有5个碱基匹配上了(也就是位于第1 2 3 4 10位置上的碱基),如果使用其他方式,匹配上的碱基数目尽会不会变多?

这里我们可以在两条序列上引入间隔(gap)(翻译成“空位”似乎听起来更适合),如下图,
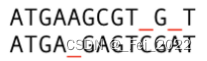
seq1在序号10和12位置处插入了空位,seq2在序号5位置处插入了空位,这样,两条序列的长度变得相同,同时匹配上的碱基数目也变多了,由原来的5个碱基变成了现在的9个碱基。
同时,插入空位的方式不是唯一的,在不同位置插入,也可能得到一样的匹配效果。下图显示了另外一种方式:
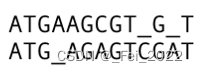
仔细对比上面两图,我们发现第二中方式中seq2插入空位的位置是序号4,而第一种方式中seq2插入的空位位置是序号5。下图更详细地说明了两种方式的区别,但最后的结果都是一样的。
