【NC 2023】Macformer-人工智能大环药物分子生成方法

主要标注了关键氢键
【NC 2022】Generative deep learning enables the discovery of a potent and selective RIPK1 inhibitor
A图
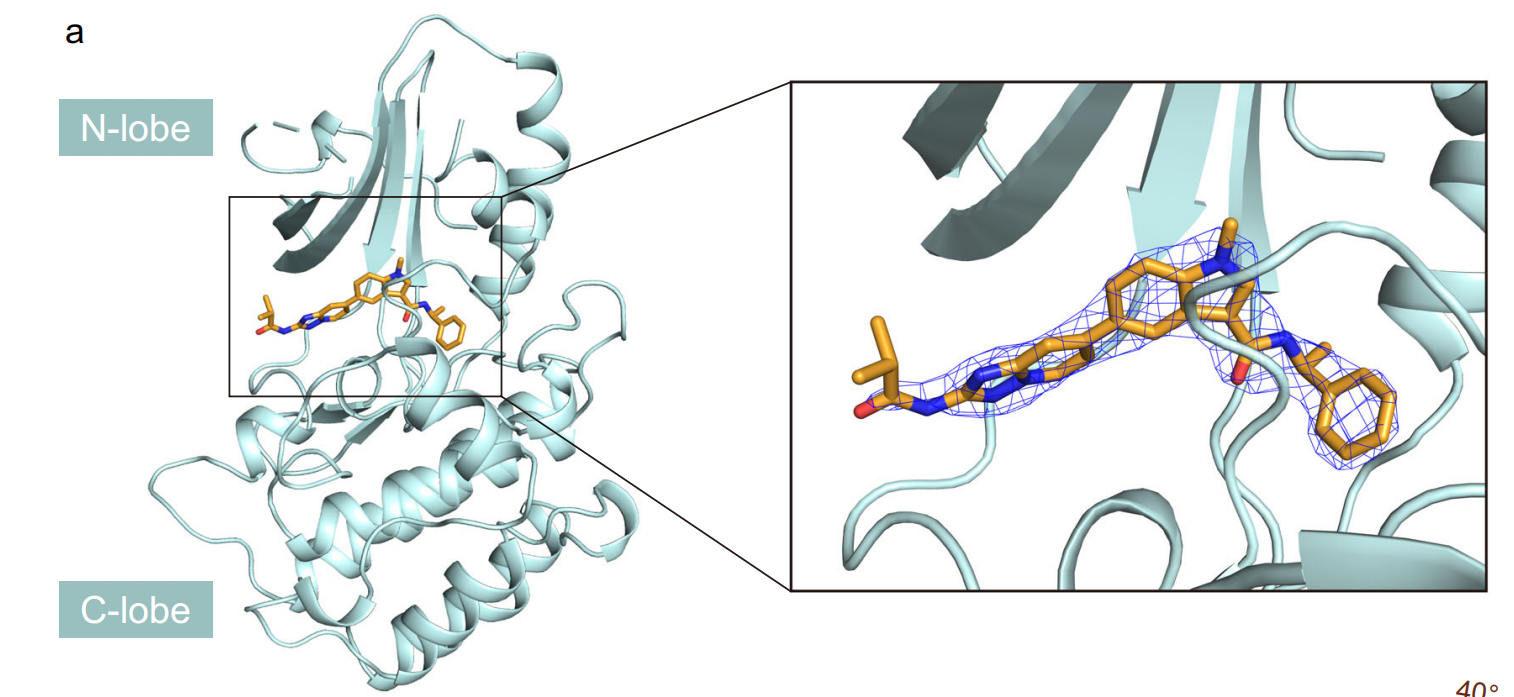
a. The binding site of RI-962. 2Fo-Fc electron density map (contoured at 1.0 σ, blue mesh) is shown for RI-962 (in orange)
-
RI-962的结合部位(The binding site of RI-962):这是指RI-962与某个较大分子(例如蛋白质)结合的特定区域。
-
2Fo-Fc电子密度图(2Fo-Fc electron density map):在X射线晶体学中,2Fo-Fc图是一种常用的电子密度图,用于表示观察到的电子密度与计算出的电子密度之间的差异。Fo代表观察到的结构因子,Fc代表计算出的结构因子。2Fo-Fc图用于验证结构模型的准确性,并有助于识别分子的确切结构。
-
轮廓在1.0σ处(contoured at 1.0 σ):这是一个描述电子密度图的细节。电子密度图的轮廓线表示了特定电子密度的区域。1.0σ表示图中的轮廓线代表标准偏差(σ)的1倍,有助于确定电子密度的确切水平。
-
蓝色网格(blue mesh):这是电子密度图的可视化表示,通常以三维网格形式呈现。
-
RI-962(以橙色表示)(RI-962 (in orange)):RI-962的分子结构在图中用橙色表示。
所以,整体来说,这段文字描述了化合物RI-962在与其结合的分子中的结合部位。这个结合部位通过使用2Fo-Fc电子密度图(以蓝色网格形式显示,并在1.0σ处轮廓)和橙色表示RI-962来进行可视化。
B图
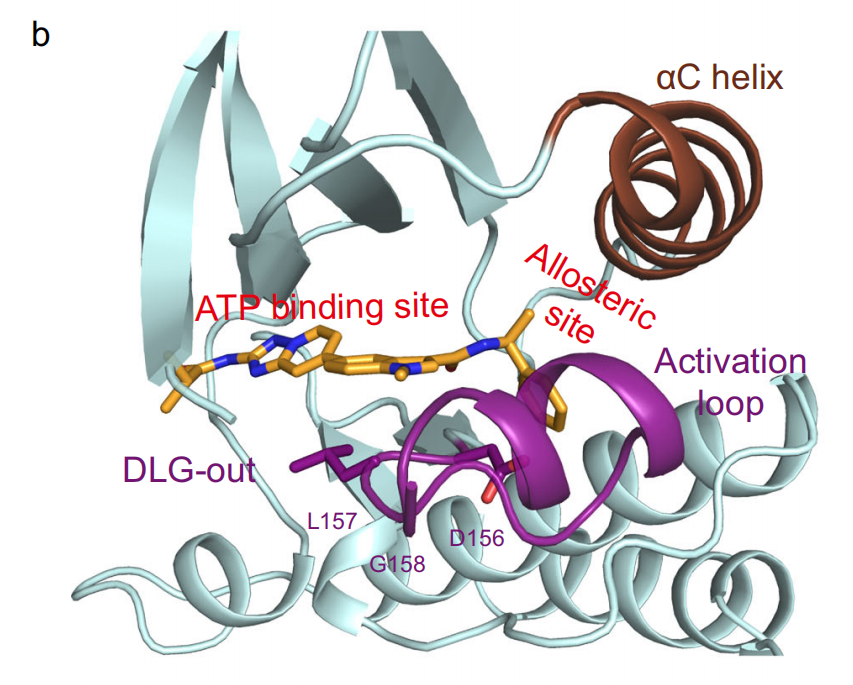
b. RI-962-bound RIPK1 adopts an inactive conformation. The αC-helix is colored in chocolate, and the activation loop is colored in deep purple. Position of RI-962 (in orange) in DLG-out structure (sphere, in deep purple) is shown.
-
RI-962-bound RIPK1 adopts an inactive conformation: 这意味着当化合物RI-962与RIPK1蛋白结合时,RIPK1采取了一种非活性的构象。换句话说,这种结合可能阻止了RIPK1的功能。
-
The αC-helix is colored in chocolate: 这句话描述了RIPK1中特定的αC-螺旋的颜色编码。在许多蛋白激酶中,αC-螺旋是一个重要的结构元素,参与调控激酶的活性。
-
The activation loop is colored in deep purple: 激活环(activation loop)是蛋白激酶中的另一个关键结构,通常通过磷酸化或去磷酸化来调控激酶活性。在此可视化中,它被着色为深紫色。
-
Position of RI-962 (in orange) in DLG-out structure (sphere, in deep purple) is shown: 这句话描述了RI-962(用橙色表示)在所谓的DLG-out结构中的位置。DLG-out结构可能是RIPK1中的一个特定构象,其中RI-962以深紫色的球体形式表示。
总的来说,这段文字提供了关于RI-962与RIPK1蛋白结合后的结构和构象的详细描述,包括不同结构元素的可视化颜色编码
C图
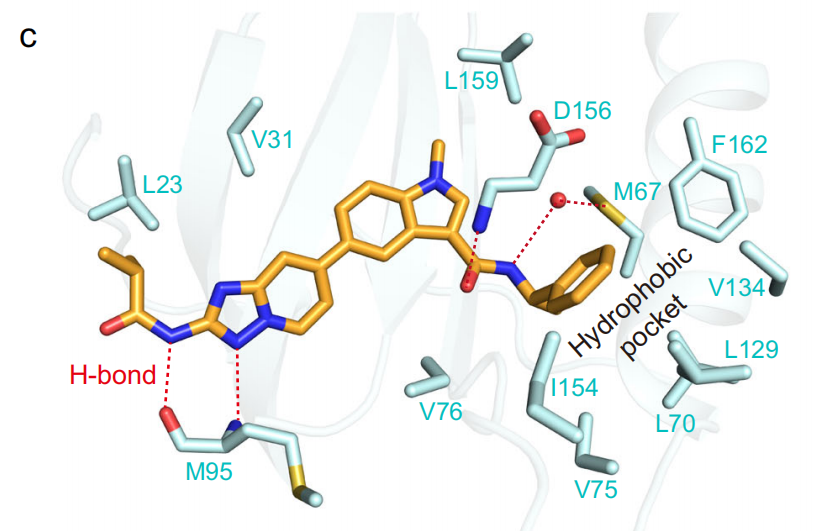
c. Interactions between RI-962 and the RIPK1 kinase domain (key residues are shown as pale cyan sticks)
Hydrogen bonds are represented as red dashed circles. The steric clash is represented as red circle. Salt bridge is represented as positive-negative symbol.
-
Interactions between RI-962 and the RIPK1 kinase domain (key residues are shown as pale cyan sticks):这句描述了RI-962与RIPK1激酶结构域之间的相互作用,其中关键的氨基酸残基以淡青色棒状表示。这些关键残基可能在RI-962与RIPK1之间的结合中起着关键作用。
-
Hydrogen bonds are represented as red dashed circles:氢键是分子间相互作用的一种形式,涉及氢原子与电负性较强的原子(如氧或氮)之间的吸引。在这里,氢键用红色虚线圆圈表示。
-
The steric clash is represented as red circle:如前所述,"steric clash"指的是分子中原子或基团间的空间拥挤,可以通过红色圆圈来表示。这种空间拥挤可能会影响分子的构象和功能。
总体来说,这段描述提供了RI-962与RIPK1激酶结构域之间相互作用的详细视觉表述,包括不同类型的化学键合和空间关系,以及如何在图形表示中描绘这些相互作用。这样的描述通常用于科学论文或报告,以详细解释分子结构和功能的特定方面。
D图

d. Superimposition of the cocrystal structures of RIPK1 (in salmon)-Cpd8 (in salmon) and RIPK1 (in pale cyan)-RI-962 (in orange). The sidechains of K45 and E63 are shown in sticks.
Salt bridge is represented as positive-negative symbol:盐桥是一种特殊类型的分子间相互作用,涉及正负电荷之间的吸引。这里将其表示为正负符号,通常涉及氨基酸的带电侧链之间的吸引。
-
共晶结构(cocrystal structures): 共晶结构涉及两种或更多分子在单一晶体结构中的排列。在这种情况下,共晶结构涉及RIPK1与两种不同化合物的结合。
-
RIPK1 (in salmon)-Cpd8 (in salmon): 这部分描述了与RIPK1结合的化合物Cpd8的结构,这两者都用鲑鱼色表示。
-
RIPK1 (in pale cyan)-RI-962 (in orange): 这部分描述了与RIPK1结合的化合物RI-962的结构,其中RIPK1以淡青色表示,RI-962以橙色表示。
-
叠加(Superimposition): 叠加是一种将两个或多个结构对齐的过程,以便更容易比较它们之间的相似性和差异。在这种情况下,叠加用于对比RIPK1与这两种化合物的结合方式。
-
The sidechains of K45 and E63 are shown in sticks: 这部分提到K45和E63这两个氨基酸残基的侧链以棒状形式表示。这些残基可能在RIPK1与这些化合物结合中起着关键作用。
总而言之,这句描述在图形上展示了RIPK1与两种化合物Cpd8和RI-962的共晶结构叠加,突出了其中关键残基的侧链,以便于比较和分析这些结构之间的相似性和差异。
E图
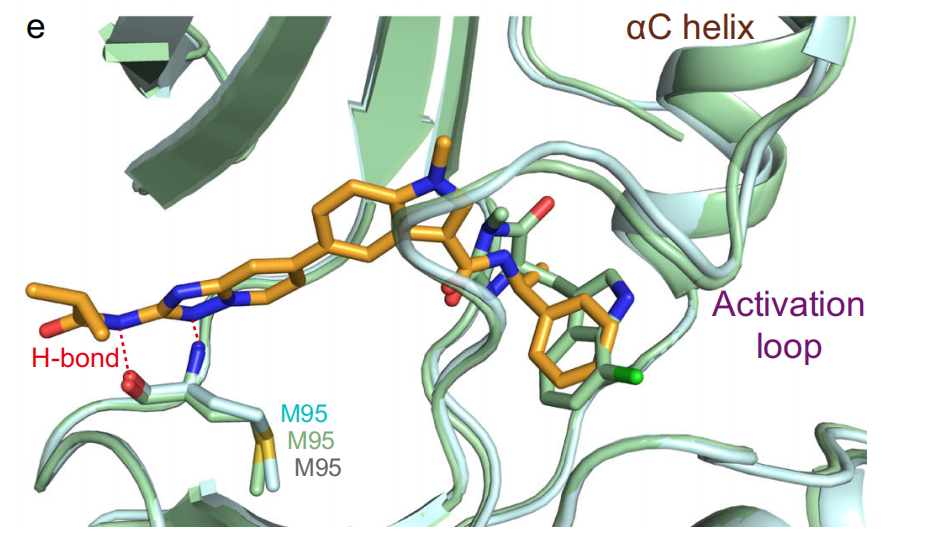
e. Superimposition of the co-crystal structure of RIPK1 (in pale green)-Nec-1a (in pale green) and RIPK1 (in pale cyan)-RI-962 (in orange).
【JMC 2023】通过目标导向分子…ATM抑制剂联合化疗治疗癌症

其中这个“(B)”比较奇特
-
ATM蛋白:ATM是一种与DNA损伤响应有关的蛋白质。当DNA受损时,ATM会被激活,并参与许多关键的信号传导途径,有助于损伤修复。
-
电势表面(electrostatic potential surface):这是一种用来描述分子表面上电荷分布的方法。通过对电势表面的可视化,科学家们可以了解分子如何与其周围环境相互作用,尤其是与其他带电粒子的相互作用。
-
代表性构象(representative conformation):蛋白质等大分子常常可以存在多种不同的空间构象。代表性构象是指在某种分析或模拟过程中,特定构象被选为代表整个构象集合的结构。
-
PCA(主成分分析):主成分分析是一种统计技术,用于减少数据的维度,同时保留其主要的变化模式。在分子动态模拟中,PCA常用于识别和分析分子运动的主要模式。
-
10r的对接结构(docking structure of 10r):这部分可能是指某种称为10r的小分子与ATM蛋白进行分子对接的结构。分子对接是一种计算技术,用于预测小分子如何与较大的生物分子(如蛋白质)相互作用。
综合上述解释,这句话可能描述的是:通过主成分分析(PCA)得到ATM蛋白的代表性构象,并研究了其中与10r小分子的对接结构,然后通过电势表面分析了该构象的电荷分布特征。