写在前面
最近在作图,一直迷恋上TBtools了,真是感觉上手后,都不想自己写代码来分析或画图了。TBtools确实牛X,再一次发自内心的!!!!昨晚,使用TBtools来做共线分析后做Circos图。关于使用TBtools做Circos图,CJ也做了很多的教程,以及也有很的同学或公众号推了相关的教程。具体可以看,定制Circos图:使用TBtools,从数据准备到可视化,视频手把手 | 用户教你Advanced Circos 画最好看的象形图~,还有很多。这个教程也只是自己粗糙的记录自己操作过程(是真的很粗…)。
–Du
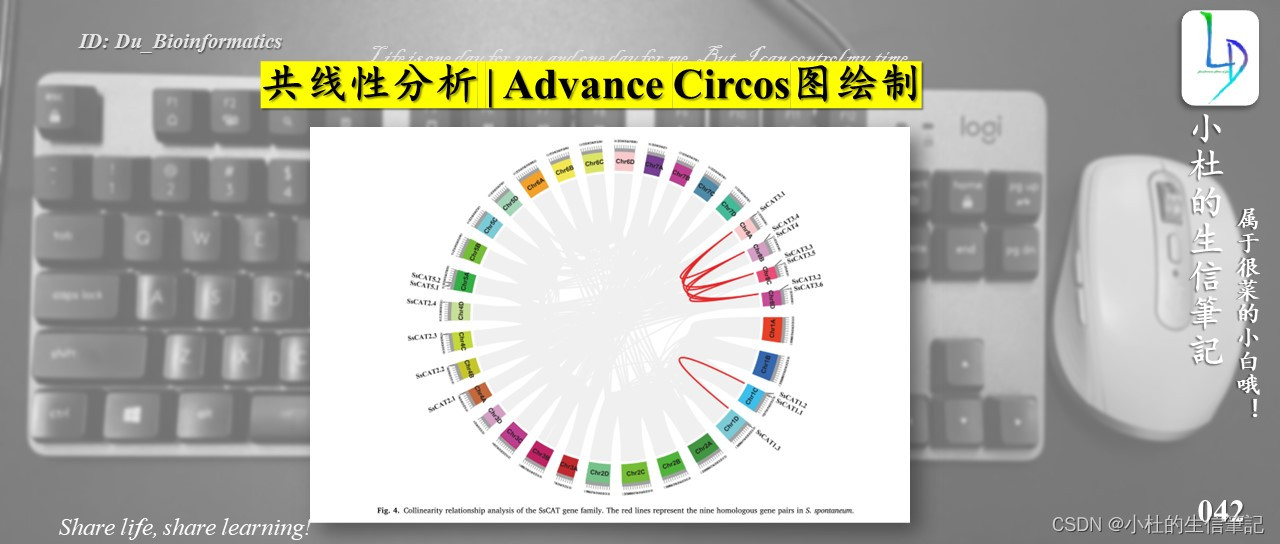
所需文件
-
基因组fa文件
-
基因组注释文件(gtf or gff)
一、获得染色体长度信息

获得结果

二、获得基因位置信息文件
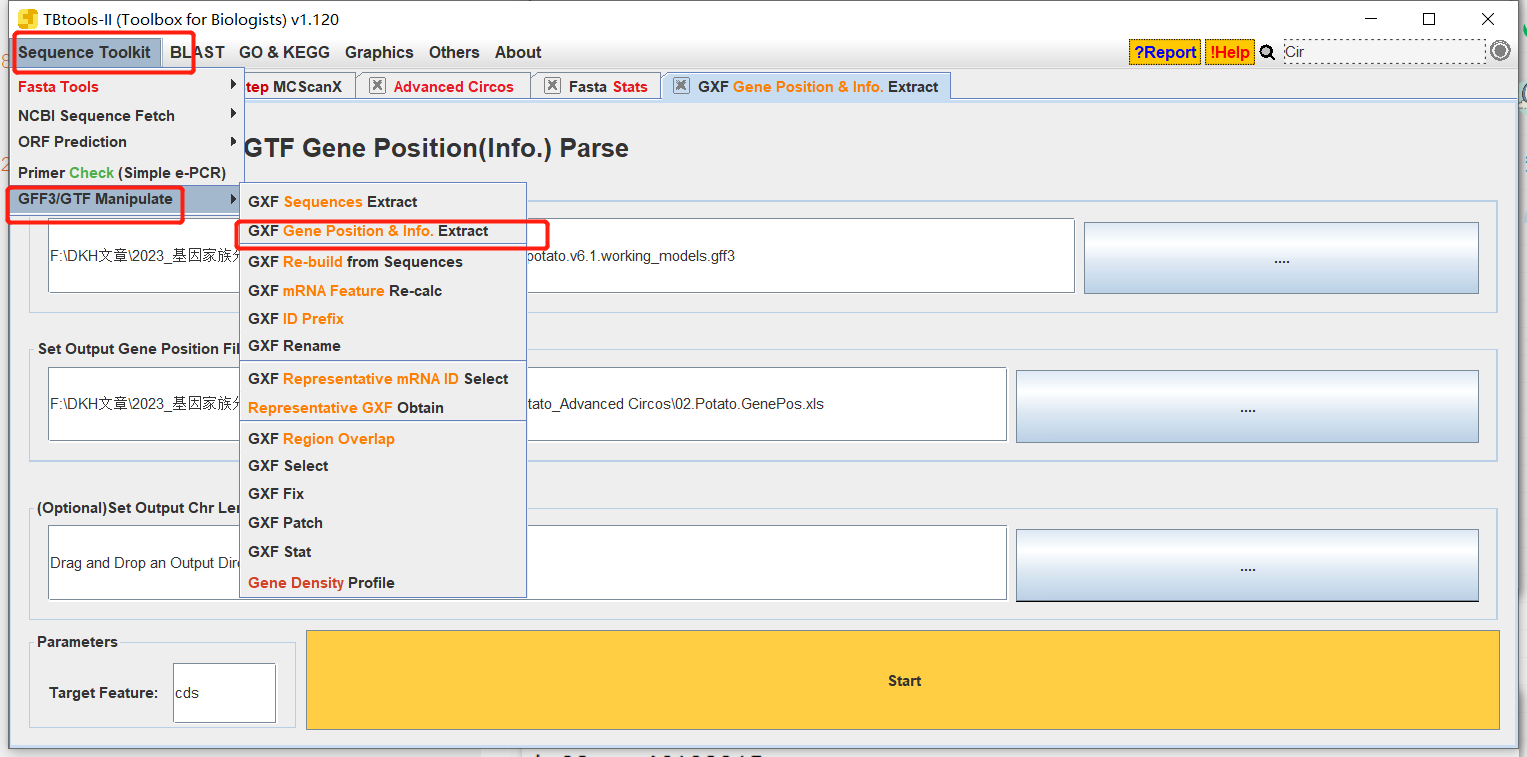
拖拽gff文件到TBtools软件,点击Start。
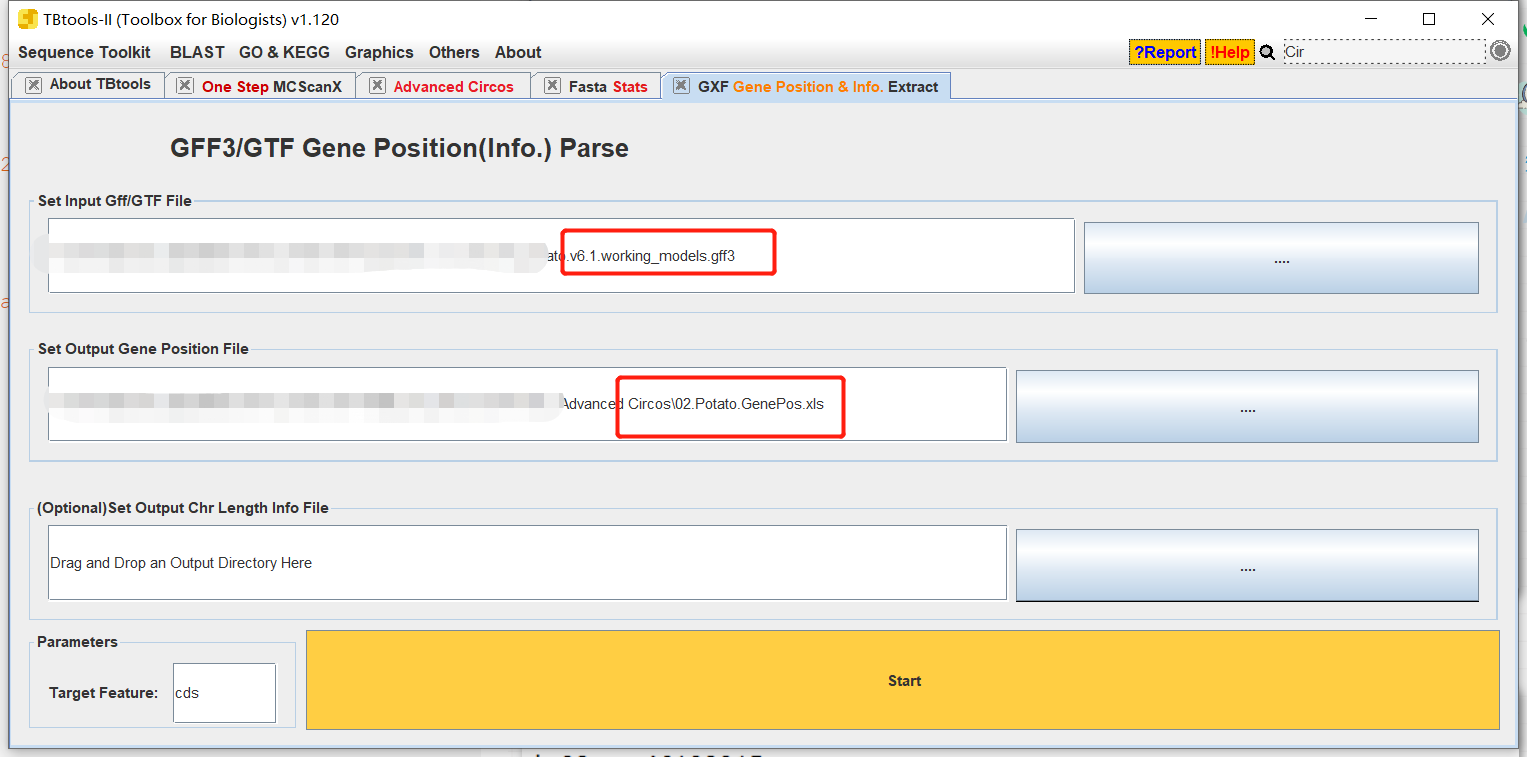
删除不必要的信息
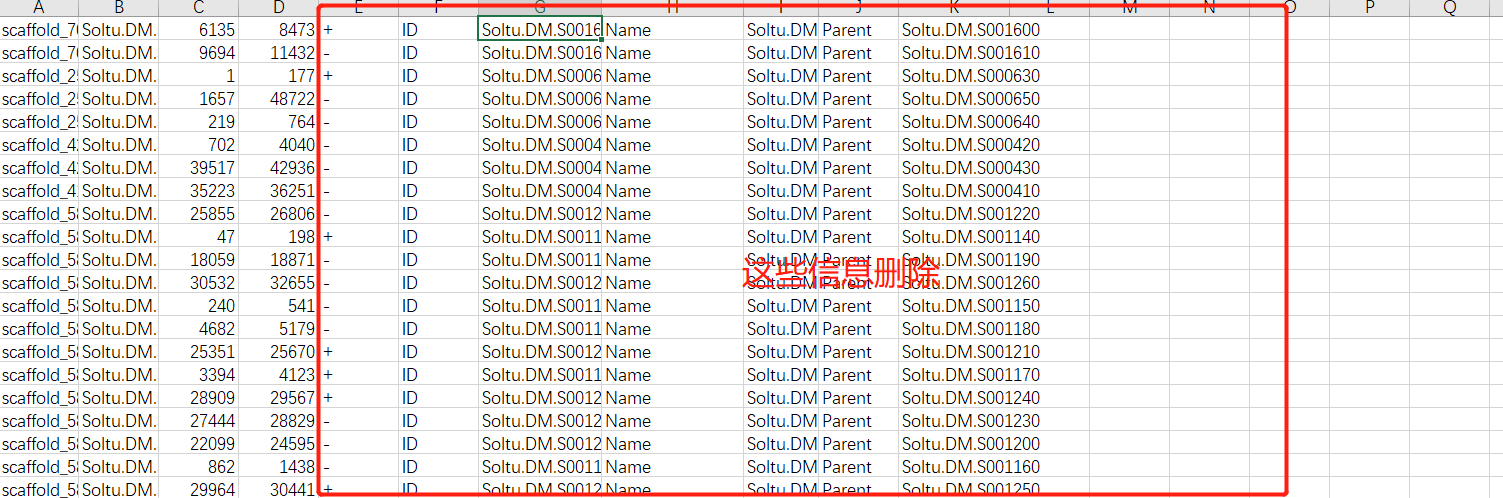
三、提取目标基因的信息


拖拽文件文件时,Selected Coluumn选择我们要match的列

四、基因组内的同源复制信息制作
使用MCScanx,分别输入物种的fa和gtf信息。

结束以后,可以获得geneLinkedRegion.tab.xls的文件,

五、选择目的基因关联信息
使用Table Row Extract or Filter进行match。
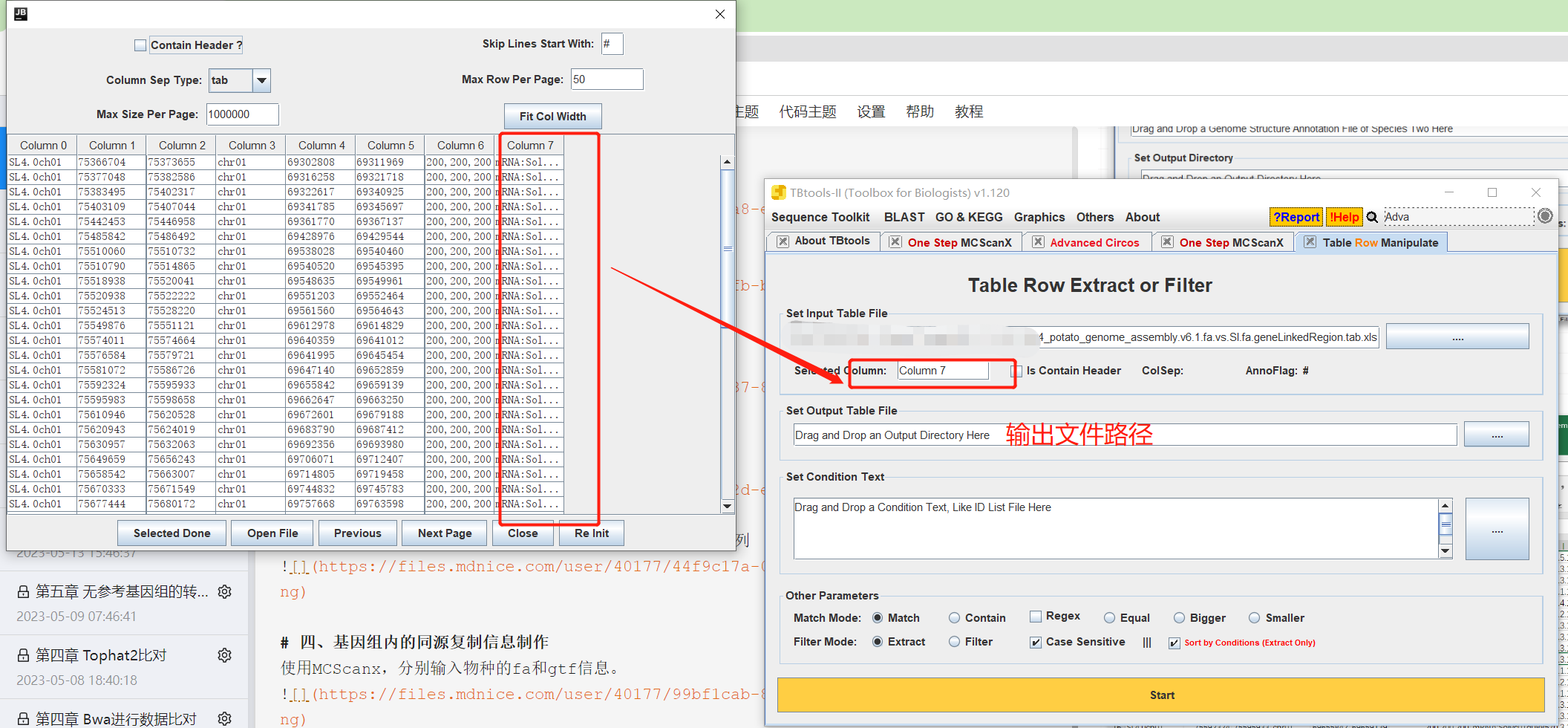
下面的操作为重点,不然匹配出来的文件是空。
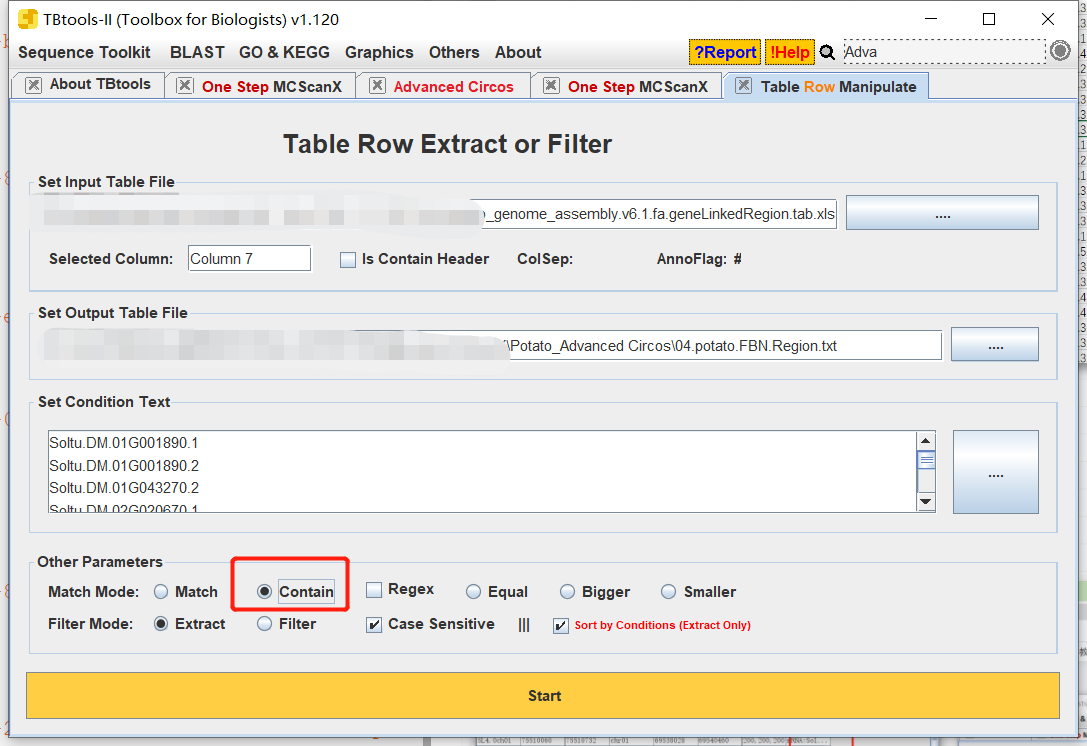
这样就可以获得次作物中,基因组复制的信息位点。
将获得的信息复制到geneLinkedRegion.tab.xls的文件中,并修改颜色。

六、出图
将以上的信息文件输入到Advanced Circos中即可出图

七、调整
出图后,我们自己需要调整很多的参数,如颜色,字体大小,距离…等等,具体可以根据自己的需求来调整。
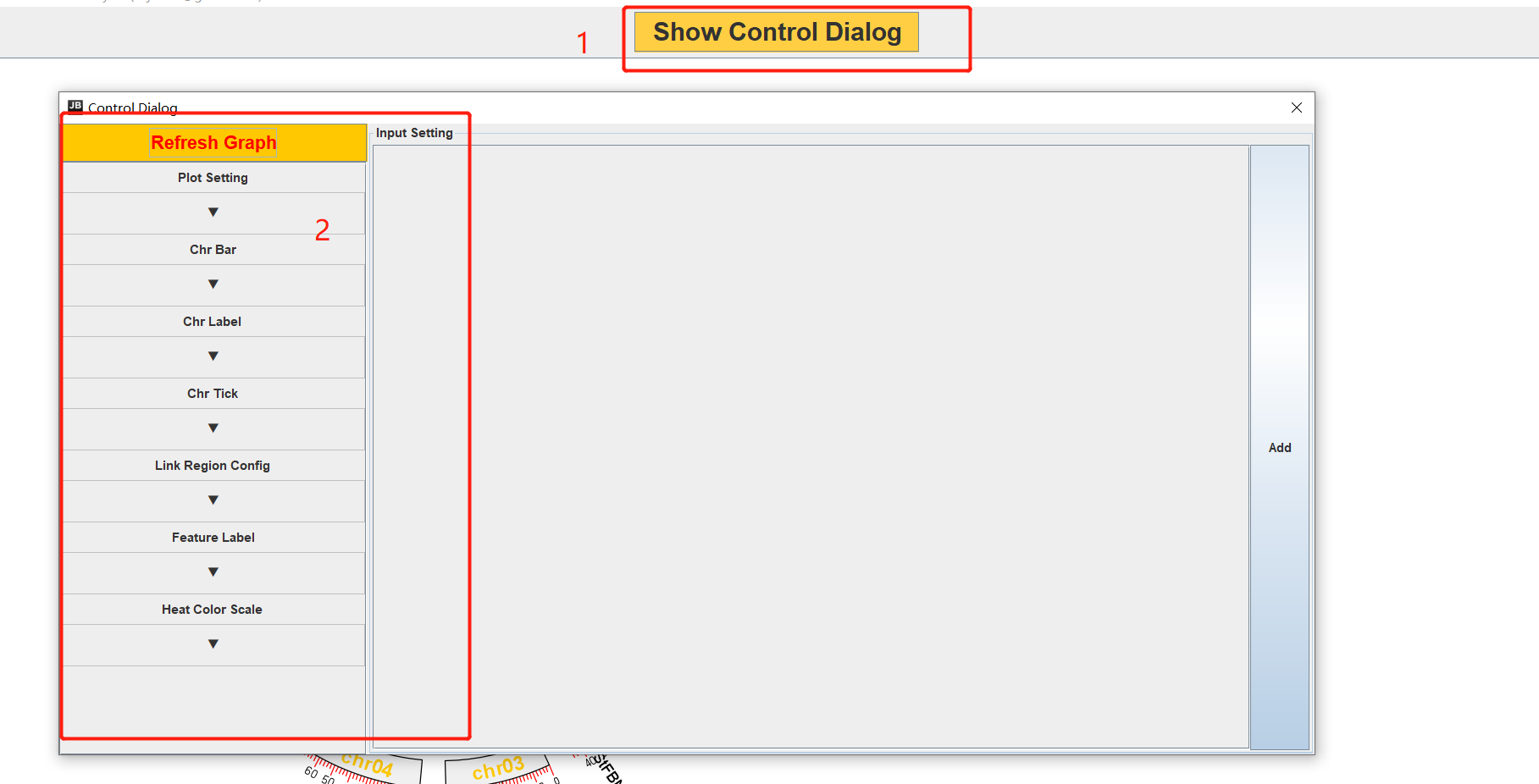
在这里可以调整图形整体的参数,如chr、距离等。
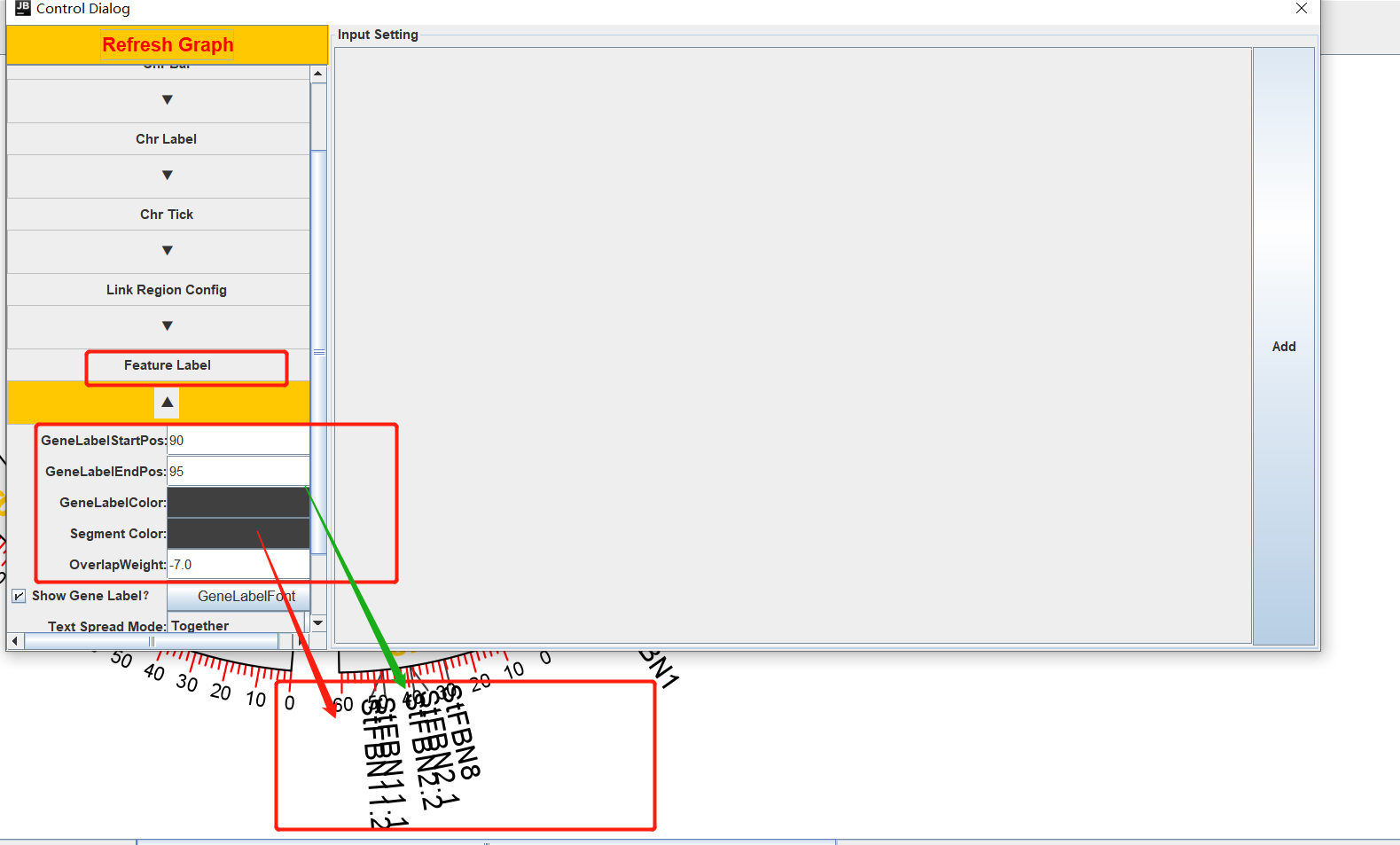
比如,调整Feature Label 可以调整显示基因的间距。

调整染色体的颜色
更多的参数调整,我们可以更具那些优秀的教程一步一步的操作。以及,需要我们自己结合自己实际进行操作。

对于第一次绘制类似图形的童鞋,我还是建议找一张自己看着好看的图形作为参考模仿着绘制。自己作图时一直这样做的,模仿着并优化着,你可以将多张图形整合到自己图形中。
ENDING !!!
往期文章:
1. 最全WGCNA教程(替换数据即可出全部结果与图形)
2. 精美图形绘制教程
3. 转录组分析教程
- 1.课程介绍
- 2.第一章 Linux基础
- 3.第一章 生信软件安装
- 4.第二章 转录组数据的下载
- 5.第三章 参考基因组序列和注释文件的下载
- 6.转录组上游分析教程 | 第四章 数据过滤
- 7.第四章 Hisat2进行数据比对
- 8.第四章 Bowtie2进行数据比对
- 9.第四章 BWA进行数据比对
- 10.第四章 Tophat2比对
- 11.第五章 无参考基因组的转录组分析
- 12.第六章 转录本定量分析
话说公众号需要标星,这样公众号的内容你才不会错过。那么,我们也动手标一下吧。
小杜的生信筆記,主要发表或收录生物信息学的教程,以及基于R的分析和可视化(包括数据分析,图形绘制等);分享感兴趣的文献和学习资料!!