古代玻璃制品的成分分析与鉴别
摘 要
对于问题一:判断文物是否风化,现逐级对玻璃类型、玻璃纹饰、玻璃颜色进行分析,绘制出流程图,即可得到玻璃表面风化与玻璃的类型、纹饰、颜色之间的关系;分析玻璃表面有无风化的化学成分含量统计规律,通过查阅文献了解到统计规律为随机事件在大量的试验中出现的频率,常会在某个固定的常数附近摆动,即选用期望值来表示统计规律,将玻璃类型与有无风化之间进行结合分类,得到高钾玻璃风化、高钾玻璃未风化、铅钡玻璃风化、铅钡玻璃未风化四种类别,对得到的四种类别数据进行归一化处理,求出期望值;对风化前的化学成分含量量进行预测,利用上一小问所求得四种数据类别的期望值进行风化前后求差值,对求得差值与表单2中分化后的化学成分相结合,对无效数据进行剔除,余值即为所预测的化学成分含量。
对于问题二:分析不同类别玻璃的分类规律,对问题一中所求得四种数据类别的期望值利用MATLAB软件进行相关性分析,得到相关分类依据,高钾玻璃的分类依据为氧化钾的含量,铅钡玻璃的分类依据为氧化钠、氧化铅、氧化钡、二氧化硫的含量;选择合适的化学成分进行亚类划分,根据类型,纹饰,表面风化可分为7个类别,将所得7个类别再根据颜色的不同分为22个亚类,然后根据颜色找到相对应颜色的化学成分得到四个化学成分,分别为氧化铁,氧化铜,氧化铅,氧化锡,所求分类结果合理,用单因素灵敏度分析对所求结果进行分析后得到分析前后变化不大说明模型是可靠的。
对于问题三:鉴别未知类别的文物,对其所属类型进行分类。借鉴问题一的统计规律计算方法,对所给8种不同文物,对其化学成分进行累加求和,归一化处理,借鉴问题二的分类规律,找出对高钾、铅钡玻璃影响最大的化学成分为氧化钾和氧化铅,对于表单3比较氧化钾和氧化铅的化学成分占比,当氧化钾的化学成分占比大于氧化铅的化学成分占比时所求的玻璃类型为高钾玻璃(若氧化钾的占比=氧化铅的占比时,则看二氧化硫有值时为高钾玻璃),反之则为铅钡玻璃。得到的结果为A1、A6、A7为高钾玻璃,A2、A3、A4、A5、A8为铅钡玻璃。对分类结果进行敏感性分析,发现变化不大,说明氧化铅(PbO),氧化钾(K2O)比较稳健。
对于问题四:为分析不同类别的玻璃样品化学成分之间的关联关系,根据问题二中的亚划分方法将玻璃样品分为7个类别和选择4种化学成分进行求期望值,将所求值拟合出不同类别下的4种化学成分图,由关联关系的定义可知,对于不同类别下的化学成分之间举有较强的依赖关系。分析其差异性,运用SPSS软件对所求期望值进行T检验,得到氧化铁、氧化铜的差异性小,氧化铅、氧化锡的差异性大。
关键词:归一化处理,单因素灵敏度分析,T检验
- 问题重述
玻璃的出现对于古代百姓生活产生了巨大的影响。风化现象是古代玻璃的一种最常见现象,由于古代玻璃长期被埋藏于地下,且因当时制造生产条件的限制会导致玻璃出现不同程度的腐蚀和风化。因此探究古代玻璃的化学成分含量来反映制作工艺及流程具有很重要的意义。
生产玻璃的主原料为石英砂,主成分为二氧化硅。为降低石英砂的熔点,在炼制时添加草木灰、天然泡碱、硝石和铅矿石等助熔剂。并添加石灰石作为稳定剂。铅钡玻璃的氧化铅、氧化钡的含量较高,钾玻璃是以含钾量高的物质烧制而成的。现有部分古代玻璃制品的相关数据,附件表1给出了该文物的分类信息,附件表2给出了该文物主成分所占的比例。所有数据的化学成分求和都为100%,但因科技检测的影响导致部风化学成分求和比例不为100%,故规定相应化学成分累加求和位于85%~105%之间均为有效数据。
对相关数据进行分析建立数学建模,解决下列问题:
对于问题一:分析玻璃表面风化与玻璃类型、纹饰和颜色的关系;根据玻璃的类型,分析玻璃表面有无风化的化学成分含量统计规律,并且根据对风化点的检测数据,预测风化前化学成分的含量。
对于问题二:依据附件给出的相关数据,对高钾玻璃和铅钡玻璃的分类规律进行分析;对于每个不同类别,选择合适的化学成分对其进行亚类划分并给出具体的分析方法和划分结果,并分析分类结果的合理性和敏感性。
对于问题三:分析附件表3中未知类别玻璃文物的化学成分,鉴别其所属类型,对分类结果进行敏感性分析。
对于问题四:对于不同类别的玻璃样品,分析各化学成分之间的相互关联关系,并且对不同类别间化学成分的关联关系进行差异性比较。
- 问题分析
2.1 问题一
为分析出玻璃表面风化与各影响因素的关系,需对各影响因素结合附件表单1进行逐一归类分析后,得出表面风化与各影响因素的相关关系;将玻璃的类型与玻璃表面有无风化情况进行分类,为方便计算,将得到的几种类别进行统一单位,找出能合理反映出玻璃表面风化前后化学成分含量的变化规律的判别条件,最终求出其相应的统计规律;为预测出风化前的化学成分含量,将表单2中已知风化前后化学成分含量进行求差值处理,对所求差值变化量与风化前化学成分相结合,即可判别出风化前的化学元素含量。
2.2 问题二
对于问题二,需分析不同类型玻璃的分类规律,利用问题一第2小问对玻璃所分的不同类别,简单分析是何种元素对玻璃的分类有影响,然后结合MATLAB软件对可能影响的要素进行相关性判断。
2.3 问题三
对所给的未知类别的文物进行化学成分求和处理,分别表示出各化学成分的占比,由于问题二中已求出不同类别玻璃的分类规律是由相关化学成分决定,结合不同类别玻璃的分类规律,对文职类别的文物类别进行填充。
2.4 问题四
分析二者之间的关联关系,依据问题二中的分类方法,对不同类别下的化学成分找出其中的关联关系进行分析,的到期相关关系,利用SPSS软件进行差异性分析。
- 模型假设
1.假设进行单因素分析时其他因素不变;
2.假设文物没有受到其他因素的影响如天灾,人祸等;
3.假设文物在实验时间内不会被空气氧化影响实验结果;
4假设文物表面清洗干净没有污渍影响颜色,纹饰的判断;
- 模型的建立与求解
4.1 问题一
分析玻璃表面风化与玻璃类型、纹饰和颜色的关系;根据玻璃的类型,分析玻璃表面有无风化的化学成分含量统计规律,并且根据对风化点的检测数据,预测风化前化学成分的含量。
4.1.1 问题一的分析
对于第1小问,若要判断文物是否风化,首先,对玻璃的类型进行分类,其次,对分类后的玻璃类型进行纹饰分类,最后,对分类后的纹饰进行颜色分类。即可分析出文物表面是否风化与玻璃类型、纹饰和颜色的关系。
对于第2小问,为分析风化前后化学成分含量的变化规律,结合玻璃类型及玻璃表面文物的风化状况对化学成分含量变化进行分析,易知玻璃类型与玻璃表面有无风化共有四类组合,分别为高钾玻璃风化、高钾玻璃未风化、铅钡玻璃风化、铅钡玻璃未风化四种。为统一数据指标,便于计算,将这四组数据进行分类后的各自化学成分进行归一化处理,对于处理后的数据求期望值。
对于第3小问,为预测风化前的化学成分含量,将表单1的玻璃类型填充在表单2中并与表单2中的文物采样点进行匹配,由于第2小问中已求出玻璃表面有无风化的情况下化学成分含量的期望值,现对不同的玻璃类别进行风化前后的期望值求差值(未风化—风化),将求得的差值与文物采样点的数据相结合,剔除掉玻璃风化后不含相应元素的数据值以及相结合后数据值为负数的值,剩余值即为所预测的风化前化学成分含量。
4.1.2 问题一模型的建立和求解
对于第1小问,分析玻璃表面风化与各因素的关系,结合表单1分别对玻璃类型,纹饰以及颜色进行分类,找出玻璃表面风化与各因素的关系,为更直观反映出相关关系,绘制出图1关系图。
图 1文物表面风化与玻璃类型、纹饰及颜色的关系
由图1可知,玻璃类型为高钾玻璃,纹饰为A、C的情况下,无论为何颜色玻璃其表面均风化。纹饰为B的情况下,仅有蓝绿一种颜色且玻璃表面风化;玻璃类型为铅钡玻璃,纹饰为A的情况下,颜色为深蓝色玻璃表面无风化,颜色为黑、蓝绿两种玻璃表面风化,颜色为浅蓝色玻璃表面是否风化无法判断(浅蓝色既有风化部分,也有未风化部分)。纹饰为C的情况下,颜色为绿色玻璃表面无风化,颜色为浅蓝、蓝绿两种玻璃表面风化,颜色为浅绿色、深绿色、紫色玻璃表面是否风化无法判断(既有风化部分,也有未风化部分)。
对于第2小问,由题知,各化学成分比例累加和在85%-105%之间为有效值,通过对附件中的表单二进行分析发现采样点为15和17的各化学成分比例累加和分别为79.47,71.89不满足题目要求,故对采样点15和采样点17进行数据剔除。
对玻璃的类型及玻璃表面有无风化情况分析后得到以下四种类型:高钾玻璃风化、高钾玻璃未风化、铅钡玻璃风化、铅钡玻璃未风化。对以上四种情况所列数据进行归一化处理[1]:
归一化处理:为了数据处理方便提出来的,把数据映射到0~1范围之内处理,更加便捷快速,归到数字信号处理范畴之内
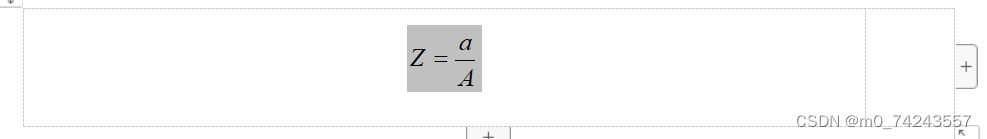
Z为归一化处理的结果,a为已分类玻璃文物的化学成分比例,A为该文物采样点所采集的化学元素成分比例总和。
运用MATLAB软件对数据进行归一化处理,下表为部分归一化数据处理
| 文物采样点 |
类型 |
表面风化 |
二氧化硅 |
氧化钠 |
氧化钾 |
| 01 |
高钾 |
无风化 |
0.6858 |
0 |
0.1006 |
| 02 |
铅钡 |
风化 |
0.1994 |
0 |
0 |
| 03部位1 |
高钾 |
无风化 |
0.6380 |
0 |
0.1134 |
| 03部位2 |
高钾 |
无风化 |
0.8170 |
0 |
0.0968 |
| 04 |
高钾 |
无风化 |
0.7102 |
0 |
0.1023 |
| 05 |
高钾 |
无风化 |
0.6012 |
0.0291 |
0.1276 |
| 06部位1 |
高钾 |
无风化 |
0.6623 |
0.0213 |
0.1475 |
| 06部位2 |
高钾 |
无风化 |
0.7783 |
0 |
0 |
| 07 |
高钾 |
风化 |
0.9383 |
0 |
0 |
| 08 |
铅钡 |
风化 |
0.2328 |
0 |
0 |
| 08严重风化点 |
铅钡 |
风化 |
0.3160 |
0 |
0 |
| 09 |
高钾 |
风化 |
0.9469 |
0 |
0.0101 |
| 10 |
高钾 |
风化 |
0.9290 |
0 |
0 |
| 11 |
铅钡 |
风化 |
0.2748 |
0 |
0 |
| 12 |
高钾 |
风化 |
0.9523 |
0 |
0.0059 |
(完整的归一化数据处理见附录)。
对数据进行归一化处理后,直观地反映出各化学成分在累加和成分中的占比,同时减小了不同文物之间计算的误差。
为更好地反映出各成分的统计规律,通过查阅相关文献了解到统计规律是随机事件在大量的试验中出现的频率常会在某个固定的常数附近摆动,这种规律性们称之为统计规律性。[2]现用期望值来反映各化学成分之间的统计规律。下图为不同类型的玻璃及是否风化情况下的数学期望值。
表2不同类型下玻璃表面有无风化情况下的数学期望
| 表面风化 |
类型 |
二氧化硅 |
氧化钠 |
氧化钾 |
氧化钙 |
| 风化 |
高钾 |
94.33 |
0.00 |
0.54 |
0.87 |
| 无风化 |
高钾 |
69.23 |
0.71 |
9.52 |
5.44 |
| 风化 |
铅钡 |
25.75 |
0.22 |
0.14 |
2.80 |
| 无风化 |
铅钡 |
55.83 |
1.72 |
0.23 |
1.34 |
| 表面风化 |
类型 |
氧化铝 |
氧化铁 |
氧化铜 |
氧化铅 |
| 风化 |
高钾 |
1.94 |
0.27 |
1.57 |
0.00 |
| 无风化 |
高钾 |
6.74 |
1.97 |
2.50 |
0.42 |
| 风化 |
铅钡 |
3.06 |
0.61 |
2.33 |
44.89 |
| 无风化 |
铅钡 |
4.55 |
0.76 |
1.48 |
22.58 |
| 表面风化 |
类型 |
五氧化二磷 |
氧化锶 |
氧化锡 |
二氧化硫 |
| 风化 |
高钾 |
0.28 |
0.00 |
0.00 |
0.00 |
| 无风化 |
高钾 |
1.43 |
0.04 |
0.20 |
0.11 |
| 风化 |
铅钡 |
5.48 |
0.43 |
0.07 |
1.38 |
| 无风化 |
铅钡 |
1.10 |
0.27 |
0.05 |
0.16 |
由表2可知,对所求的期望值进行分析得出,高钾玻璃在风化前后仅有化学成分为二氧化硅的含量升高,其余化学成分的含量均降低。铅钡玻璃在风化前后化学成分为氧化钙、氧化铜、氧化铅、五氧化二磷、氧化锶、氧化锡、二氧化硫的含量升高,二氧化硅、氧化钠、氧化钾、氧化铝、氧化铁的含量降低。
为更直观表示出各成分含量的统计规律,将各玻璃风化前后化学成分含量绘制出折线统计图,如下图:
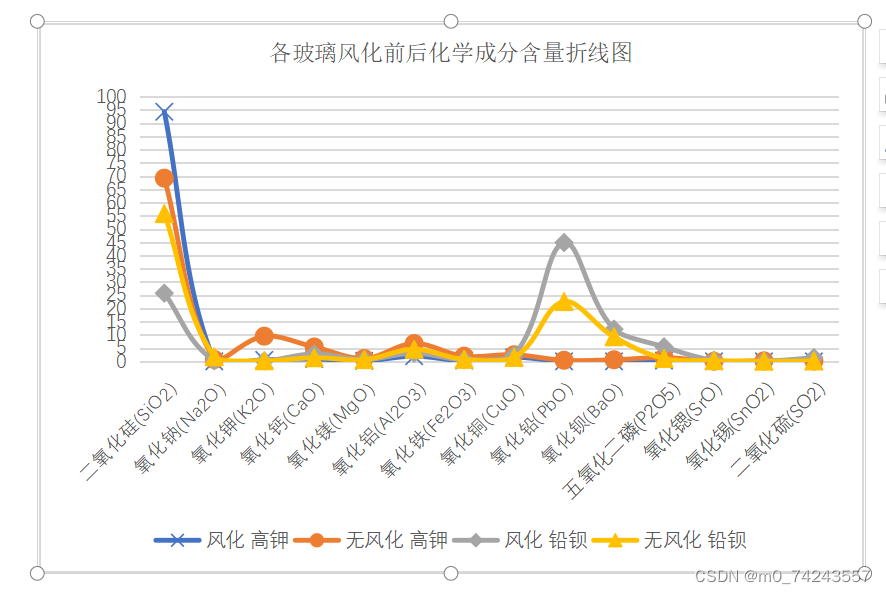
图 2各玻璃风化前后化学成分含量
对于第3小问,为预测风化前的化学成分含量,可利用第2小问中已求出的期望值进行预测。现对表单1中的玻璃类型和表面是否风化进行匹配填充到表单2中。
分别对玻璃类型及是否风化进行分类讨论,对玻璃类别为高钾玻璃时,利用第2小问中求得期望值,求出高钾玻璃风化前后化学成分含量的差值,同理,也可求出铅钡玻璃风化前后化学成分含量的差值。部分数据如下表所示(完整数据见附录):
表 3不同类型玻璃风化前后化学成分含量的差值
| 类型 |
氧化钠 |
氧化钾 |
氧化铅 |
氧化钡 |
二氧化硫 |
| 高钾 |
0.71 |
8.97 |
0.42 |
0.61 |
0.11 |
| 铅钡 |
1.5 |
0.09 |
-22.31 |
-2.87 |
-1.22 |
对于上表所求出的差值与表单2进行相加求和,即可预测出风化前的化学成分含量。
将所求差值结合表单2对文物采样点所给出的化学成分含量进行填充,部分结果如下表所示(完整数据见附录):
| 文物采样点 |
类型 |
二氧化硅 |
氧化钾 |
氧化钙 |
氧化铝 |
氧化铅 |
| 2 |
铅钡 |
65.13 |
1.02 |
1.19 |
7.23 |
28.64 |
| 7 |
高钾 |
62.42 |
6.19 |
6.23 |
||
| 8 |
铅钡 |
48.99 |
0.33 |
2.84 |
9.89 |
|
| 08 |
铅钡 |
33.46 |
2.04 |
2.61 |
13.66 |
|
| 9 |
高钾 |
64.81 |
9.29 |
5.74 |
5.57 |
|
| 10 |
高钾 |
66.56 |
9.62 |
5.33 |
5.06 |
|
| 11 |
铅钡 |
62.44 |
0.18 |
2.36 |
4.19 |
6.6 |
| 12 |
高钾 |
64.08 |
9.71 |
5.84 |
5.71 |
表 4预测风化前化学成分含量
上表为预测到的风化前化学成分含量,对于风化后无相关元素数据和进行数据填充后数值为负的情况,为避免异常数据对其余化学成分含量造成影响,现对异常数据进行剔除处理。
4.2 问题二
依据附件给出的相关数据,对高钾玻璃和铅钡玻璃的分类规律进行分析;对于每个不同类别,选择合适的化学成分对其进行亚类划分并给出具体的分析方法和划分结果,并分析分类结果的合理性和敏感性。
4.2.1 问题二的分析
对于第1小问,为分析出不同类型玻璃的分类规律,即分析颜色、纹饰和不同化学成分对于玻璃分类的影响,由问题1可知颜色和纹饰对于玻璃的分类无较强的区分。则分析玻璃的分类规律即分析各化学成分的期望值与玻璃类型和是否风化的关系,利用MATLAB软件对各化学成分的期望值与玻璃类型及玻璃表面风化的关系进行判断,通过判断即可得出高钾玻璃、铅钡玻璃的分类规律。
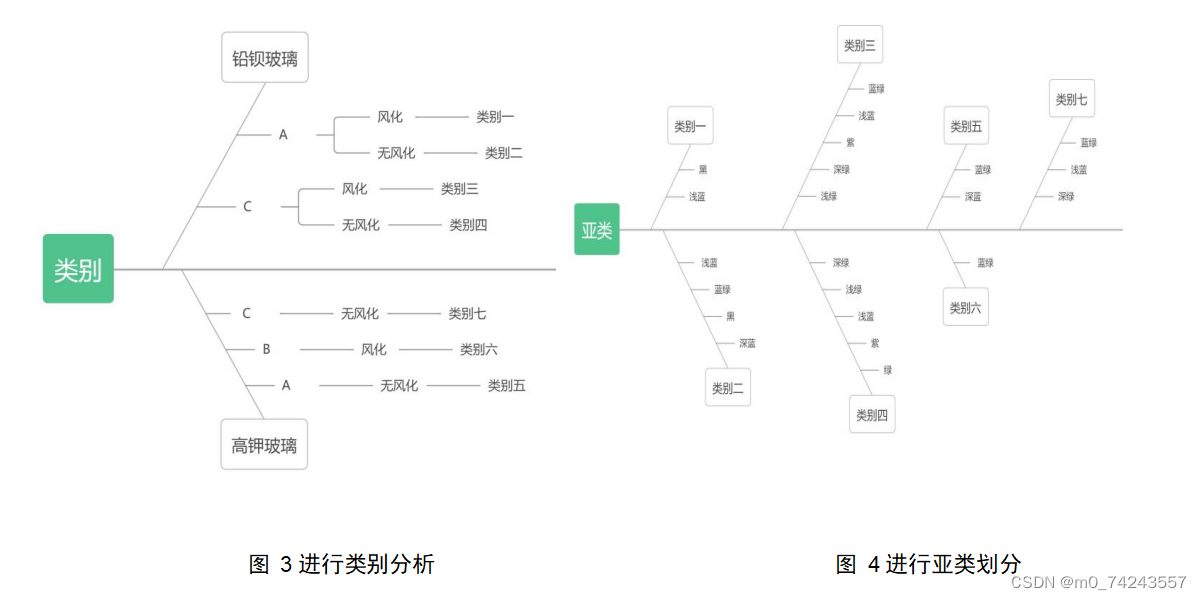
对于第2小问,首先先根据类型,纹饰,表面风化可分为7个类别,7个类别再根据颜色的不同分为22个亚类,然后根据颜色找到相对应颜色的化学成分得到四个化学成分分别为氧化铁(Fe2O3),氧化铜(CuO),氧化铅(PbO),氧化锡(SnO2)。求22个亚类,4个化学成分的期望,最后按颜色分为8类对分类结果进行合理性分析和敏感性分析。
4.2.2 问题二模型的建立模型求解
对于第1小问,由问题1可知,纹饰、颜色对于玻璃的分类影响不是最大的,故排除对纹饰、颜色对于玻璃的分类。
对表单2文物采样点的化学成分数据进行累加求和,由题知,有效数据区间介于85%—105%。对无效数据进行剔除,剔除无效数据后,将剩余数据进行归一化处理。
对高钾玻璃、铅钡玻璃和玻璃表面有无风化为依据,可分四种类型(高钾玻璃风化、高钾玻璃未风化、铅钡玻璃风化、铅钡玻璃未风化)。
分别求各类型下的化学成分期望值,判断各化学成分的期望值与玻璃表面风化的关系,现运用MATLAB软件进行判断。假定1表示该化学成分对于高钾玻璃、铅钡玻璃的影响大,即可求出各化学成分与玻璃类别的相关性,结果如下图所示:
表 5各化学成分与不同类别玻璃的相关性
| 二氧化硅 |
氧化钠 |
氧化钾 |
氧化钙 |
氧化镁 |
氧化铝 |
氧化铁 |
|
| 风化 |
0 |
1 |
0 |
1 |
1 |
1 |
1 |
| 无风化 |
0 |
1 |
0 |
0 |
0 |
0 |
0 |
| 氧化铜 |
氧化铅 |
氧化钡 |
五氧化二磷 |
氧化锶 |
氧化锡 |
二氧化硫 |
|
| 风化 |
1 |
1 |
1 |
1 |
1 |
1 |
1 |
| 无风化 |
0 |
1 |
1 |
0 |
1 |
0 |
1 |
由上表易知,氧化钠、氧化钾、氧化铅、氧化钡、二氧化硫这五种化学元素对于玻璃的类型分类有影响。对于高钾玻璃的分类依据主要由化学成分为氧化钾的含量决定,对于铅钡玻璃的分类依据主要由化学成分为氧化钠、氧化铝、氧化钡、二氧化硫的含量决定。由于二氧化硅为玻璃的主要成分,所以对于二氧化硅不做结果判断。
对于第2小问,首先先根据类型,纹饰,表面风化可分为7个类别,7个类别再根据颜色的不同分为22个亚类,然后根据颜色找到相对应颜色的化学成分得到四个化学成分,分别为氧化铁(Fe2O3),氧化铜(CuO),氧化铅(PbO),氧化锡(SnO2)。
对不同类型的玻璃进行类别分析及亚类划分的依据如下图所示:
| 氧化铅(PbO) |
0.0000 |
0.4229 |
0.2750 |
||
| 34.11% |
1.0000 |
1.4229 |
1.2750 |
||
| 1 |
34.11% |
33.77% |
33.89% |
||
图 3进行类别分析 图 4进行亚类划分
最后按颜色分为八类,应用单因素敏感度分析法[3]来进行敏感性分析见部分数据(余下见附录)
表 6对黑色氧化铅进行灵敏度分析
表 7对黑色氧化铜进行灵敏度分析
| 氧化铜(CuO) |
0.0000 |
0.0099 |
0.0059 |
|
| 34.11% |
1.0000 |
1.0099 |
1.0059 |
|
| 1 |
34.11% |
34.10% |
34.10% |
表 8对紫色氧化铅进行灵敏度分析
| 氧化铅(PbO) |
0.0000 |
0.0233 |
|
| 25.57% |
1.0000 |
1.0233 |
|
| 1 |
25.57% |
25.57% |
表 9对紫色氧化铜进行灵敏度分析
| 氧化铜(CuO) |
0.0000 |
0.3032 |
0.2314 |
|
| 25.57% |
1 |
1.3032 |
1.2314 |
|
| 1 |
25.57% |
25.57% |
25.57% |
由单元素敏感度分析法分析得知各个分类的敏感性分析前后变化不大说明模型是可靠的。
4.3 问题三
分析附件表3中未知类别玻璃文物的化学成分,鉴别其所属类型,对分类结果进行敏感性分析。
4.3.1 问题三的分析
为鉴别未知类别的玻璃文物所属类型,可利用第二问所得出的分类规律进行分析,首先,对未知类别文物所给化学成分数据进行累加求和,其次,进行归一化处理,最后,利用问题2的分类规律对附件表单3中的未知类别玻璃进行类别填充。通过查阅相关文献,利用单因素敏感度分析对数据进行灵敏度分析,选择氧化钾和氧化铅分别做单因素敏感性分析,利用Excel预测工作表相关功能进行敏感性分析。
4.3.2 问题三模型的建立和求解
对每种文物的化学成分数据进行累加求和,再对各化学成分进行归一化处理。由第二问所求的分类规律可知高钾玻璃的主要分类依据由化学成分氧化钾决定,铅钡玻璃的主要分类依据由化学成分氧化钠、氧化铅、氧化钡、二氧化硫决定且氧化铅占比最高。比较氧化钾和氧化铅的化学成分占比,当氧化钾的化学成分占比大于氧化铅的化学成分占比时所求的玻璃类型为高钾玻璃,反之则为铅钡玻璃。(当氧化钾的占比=氧化铅的占比时,比较二氧化硫,若二氧化硫存在时认为玻璃类型为高钾玻璃,反之则为铅钡玻璃)所填充部分数据如下表所示(完整表格见附录):
表 10对未知类别玻璃鉴别其属性
| 文物编号 |
类型 |
二氧化硅 |
氧化钾 |
氧化铅 |
氧化钡 |
二氧化硫 |
| A1 |
高钾 |
0.789 |
0.005 |
|||
| A2 |
铅钡 |
0.392 |
0.356 |
|||
| A3 |
铅钡 |
0.323 |
0.014 |
0.400 |
0.047 |
|
| A4 |
铅钡 |
0.369 |
0.008 |
0.253 |
0.087 |
|
| A5 |
铅钡 |
0.645 |
0.004 |
0.123 |
0.022 |
|
| A6 |
高钾 |
0.940 |
0.014 |
|||
| A7 |
高钾 |
0.912 |
0.010 |
0.001 |
||
| A8 |
铅钡 |
0.511 |
0.002 |
0.212 |
0.113 |
0.023 |
由上表可知,文物编号为A1、A6、A7的文物玻璃类别为高钾玻璃,文物编号为A2、A3、A4、A5、A8的文物玻璃类别为铅钡玻璃。
将上表结果进行敏感性分析,利用单因素敏感度分析和Excel预测工作表进行敏感度分析[4],结果如下表所示:
表 11对氧化铅和氧化钾进行灵敏度分析
| 氧化铅(PbO) |
0.0000 |
0.3563 |
0.3999 |
0.2529 |
0.1228 |
0.0000 |
0.0000 |
0.2124 |
||||||
| 16.16% |
1.0000 |
1.3563 |
1.3999 |
1.2529 |
1.1228 |
1.0000 |
1.0000 |
1.2124 |
||||||
| 1 |
16.16% |
22.15% |
25.27% |
22.71% |
19.03% |
16.16% |
16.16% |
19.73% |
||||||
| 氧化钾(K2O) |
0.0000 |
0.0000 |
0.0137 |
0.0082 |
0.0037 |
0.0136 |
0.0098 |
0.0023 |
||||||
| 16.16% |
1.0000 |
1.0000 |
1.0137 |
1.0082 |
1.0037 |
1.0136 |
1.0098 |
1.0023 |
||||||
| 1 |
16.16% |
16.16% |
16.39% |
16.30% |
16.22% |
16.39% |
16.33% |
16.20% |
||||||
敏感度随着氧化铅(PbO),氧化钾(K2O)所占比例的提高而提高,氧化铅(PbO),氧化钾(K2O)所占比例能够体现玻璃文物的敏感性。因为敏感性分析前后变化不大说明氧化铅(PbO),氧化钾(K2O)比较稳健。
4.4 问题四
对于不同类别的玻璃样品,分析各化学成分之间的相互关联关系,并且对不同类别间化学成分的关联关系进行差异性性比较。
4.4.1 问题四的分析
分析不同类别玻璃样品的化学成分之间的关联关系,由问题二知,将玻璃文物样品分为7个类别,根据颜色得到4个化学成分,分别为氧化铁、氧化铜、氧化铅、氧化锡。对表单2中的化学成分依据所分的七个类别进行求期望值,将所求期望值根据类别及各自属性绘制出折线图,分析其关联性。对不同类别之间关于化学成分的关联关系比较其差异性,通过查阅相关文献,利用SPSS软件进行T检验可得出其关联关系的差异性。
4.4.2 问题四模型的建立和求解
通过查阅相关文献,得到关联关系是一种强依赖的关系,分为单项依赖和双向依赖。
为得出化学成分之间的关联关系,分别对七个类别和四种化学成分进行分析绘制出折线统计图如下图:
由上图及关联关系的定义可知,七个不同类别下四种化学成分之间具有较强的依赖性。
对不同类别求其化学成分之间的差异性,运用SPSS软件进行T型检验,将 图 5导入到SPSS软件中进行差异性分析。结果如下表所示:
表 12对差异性进行T检验
| 单样本检验 |
||||||
| 检验值 = 0 |
||||||
| t |
自由度 |
Sig.(双尾) |
平均值差值 |
差值 95% 置信区间 |
||
| 下限 |
上限 |
|||||
| 氧化铁(Fe2O3) |
3.929 |
6 |
.008 |
1.08539% |
0.4094% |
1.7614% |
| 氧化铜(CuO) |
6.768 |
6 |
.001 |
1.88718% |
1.2049% |
2.5695% |
| 氧化铅(PbO) |
2.566 |
6 |
.043 |
19.43692% |
0.9013% |
37.9725% |
| 氧化锡(SnO2) |
1.359 |
6 |
.223 |
0.05740% |
-0.0459% |
0.1607% |
由表 12可知对化学成分的差异性进行T检验,得到氧化铁、氧化铜的差异性小,氧化铅、氧化锡的差异性大。
- 模型评价
5.1 模型优点
(1)统计规律可以保持各组内,统计资料的相同和组间资料的不同,有利于正确地认识事物的本质及其规律。
(2)统计规律是随机事件的整体性规律,它不是单个随机事件特点的简单叠加,而是事件系统所具有的必然性。
(3)单因素敏感性分析可以从多方案中快速找到最优方案。可以更好的寻求突破点加快分析进程。
(4)归一化方法操作方便,程序固定,操作要求不高。
5.2 模型缺点
(1)统计规律除非在一个稳定的群体中,连续进行同样的现况调查,否则难以调查,过程长短不确定。
(2)单因素敏感性分析在一定程度上带有自我性、臆测性,它是不全面的,容易造成低估评价风险后果,具有较大的误差。
5.3 模型改进
用随机抽样的方法抽取可加快进程缩短时间,应用多因素敏感性分析让敏感性分析更全面。
- 参考文献
[1] baike.baidu.com/item/归一化方法
[2] zhuanlan.zhihu.com/p/127962782统计规律
[3]卢佳欣.基于单因素敏感性分析的汽车三滤纸项目[J].黑龙江造纸,2018,46(04):10-14.
[4]郁玉环.利用Excel自动实现投资项目敏感性分析[J].会计之友,2014(06):117-120.
附 录
| 支撑材料文件列表 |
||||
| 序号 |
名称 |
说明 |
类型 |
备注 |
| 1 |
表单二归一化处理后的数据 |
对附录表单二数据归一化处理 |
xlsx文件 |
|
| 2 |
matlab代码 |
本文涉及的matlab代码文本 |
txt文件 |
|
| 3 |
预测值 |
问题二第二小问的预测的其风化前的化学成分含量 |
xlsx文件 |
|
| 4 |
亚类划分 |
问题二亚类划分及其划分结果 |
xlsx文件 |
|
| 5 |
所属类型 |
问题三对附件表单3中文物所属类型 |
xlsx文件 |
|
| 6 |
敏感度分析1 |
问题二敏感度分析 |
xlsx文件 |
|
| 7 |
敏感度分析2 |
问题三敏感度分析 |
xlsx文件 |
|
问题1第2问
| 文物采样点 |
类型 |
表面风化 |
二氧化硅(SiO2) |
氧化钠(Na2O) |
氧化钾(K2O) |
|||||
| 01 |
高钾 |
无风化 |
0.685821362 |
0 |
0.10066625 |
|||||
| 02 |
铅钡 |
风化 |
0.199401131 |
0 |
0 |
|||||
| 03部位1 |
高钾 |
无风化 |
0.638068594 |
0 |
0.113459745 |
|||||
| 03部位2 |
高钾 |
无风化 |
0.817069409 |
0 |
0.096863753 |
|||||
| 04 |
高钾 |
无风化 |
0.710275587 |
0 |
0.102346071 |
|||||
| 05 |
高钾 |
无风化 |
0.60128388 |
0.029142042 |
0.12767475 |
|||||
| 06部位1 |
高钾 |
无风化 |
0.662331064 |
0.021339295 |
0.147545981 |
|||||
| 06部位2 |
高钾 |
无风化 |
0.778319123 |
0 |
0 |
|||||
| 07 |
高钾 |
风化 |
0.938366562 |
0 |
0 |
|||||
| 08 |
铅钡 |
风化 |
0.232824427 |
0 |
0 |
|||||
| 08严重风化点 |
铅钡 |
风化 |
0.316023417 |
0 |
0 |
|||||
| 09 |
高钾 |
风化 |
0.94697198 |
0 |
0.010143618 |
|||||
| 10 |
高钾 |
风化 |
0.929087262 |
0 |
0 |
|||||
| 11 |
铅钡 |
风化 |
0.274899968 |
0 |
0 |
|||||
| 12 |
高钾 |
风化 |
0.952390498 |
0 |
0.005913601 |
|||||
| 13 |
高钾 |
无风化 |
0.605119385 |
0 |
0.077701335 |
|||||
| 14 |
高钾 |
无风化 |
0.624089806 |
0 |
0.125101133 |
|||||
| 16 |
高钾 |
无风化 |
0.683885968 |
0 |
0.07450465 |
|||||
| 18 |
高钾 |
无风化 |
0.631010101 |
0.034141414 |
0.124040404 |
|||||
| 19 |
铅钡 |
风化 |
0.196990716 |
0 |
0.004695337 |
|||||
| 20 |
铅钡 |
无风化 |
0.422576632 |
0 |
0.008030766 |
|||||
| 21 |
高钾 |
无风化 |
0.8705 |
0 |
0.0519 |
|||||
| 22 |
高钾 |
风化 |
0.96954213 |
0 |
0.009217513 |
|||||
| 23未风化点 |
铅钡 |
无风化 |
0.685063923 |
0 |
0 |
|||||
| 24 |
铅钡 |
无风化 |
0.508507989 |
0.028117867 |
0 |
|||||
| 25未风化点 |
铅钡 |
无风化 |
0.557409326 |
0.082072539 |
0 |
|||||
| 26 |
铅钡 |
风化 |
0.273596939 |
0.012967687 |
0 |
|||||
| 26严重风化点 |
铅钡 |
风化 |
0.131073088 |
0 |
0 |
|||||
| 27 |
高钾 |
风化 |
0.9235 |
0 |
0.0074 |
|||||
| 28未风化点 |
铅钡 |
无风化 |
0.464410976 |
0 |
0 |
|||||
| 29未风化点 |
铅钡 |
无风化 |
0.521430043 |
0.023799712 |
0 |
|||||
| 30部位1 |
铅钡 |
无风化 |
0.529919803 |
0.047912811 |
0.002981699 |
|||||
| 30部位2 |
铅钡 |
无风化 |
0.524310521 |
0.058018386 |
0.003575077 |
|||||
| 31 |
铅钡 |
无风化 |
0.350587034 |
0 |
0.0143951 |
|||||
| 32 |
铅钡 |
无风化 |
0.523007856 |
0.058565453 |
0.001530456 |
|||||
| 33 |
铅钡 |
无风化 |
0.55588355 |
0 |
0.003053746 |
|||||
| 34 |
铅钡 |
风化 |
0.25815588 |
0 |
0 |
|||||
| 35 |
铅钡 |
无风化 |
0.647938931 |
0.030941476 |
0.001119593 |
|||||
| 36 |
铅钡 |
风化 |
0.352133347 |
0 |
0.002201489 |
|||||
| 37 |
铅钡 |
无风化 |
0.623144194 |
0.027049014 |
0.001118568 |
|||||
| 38 |
铅钡 |
风化 |
0.301465969 |
0 |
0 |
|||||
| 39 |
铅钡 |
风化 |
0.224475018 |
0 |
0 |
|||||
| 40 |
铅钡 |
风化 |
0.17653735 |
0 |
0 |
|||||
| 41 |
铅钡 |
风化 |
0.366899098 |
0 |
0.002563577 |
|||||
| 42未风化点1 |
铅钡 |
无风化 |
0.669612923 |
0 |
0 |
|||||
| 42未风化点2 |
铅钡 |
无风化 |
0.559768833 |
0 |
0.002534726 |
|||||
| 43部位1 |
铅钡 |
风化 |
0.405305746 |
0.022738912 |
0.001433985 |
|||||
| 43部位2 |
铅钡 |
风化 |
0.046925896 |
0 |
0 |
|||||
| 44未风化点 |
铅钡 |
无风化 |
0.706568011 |
0 |
0.002128522 |
|||||
| 45 |
铅钡 |
无风化 |
0.689836863 |
0 |
0.002634512 |
|||||
| 46 |
铅钡 |
无风化 |
0.374164134 |
0 |
0 |
|||||
| 47 |
铅钡 |
无风化 |
0.323017799 |
0 |
0 |
|||||
| 48 |
铅钡 |
风化 |
0.169524196 |
0 |
0 |
|||||
| 49 |
铅钡 |
风化 |
0.334077305 |
0.014000203 |
0 |
|||||
| 49未风化点 |
铅钡 |
无风化 |
0.612792575 |
0.030871671 |
0.002017756 |
|||||
| 50 |
铅钡 |
风化 |
0.300121507 |
0 |
0 |
|||||
| 50未风化点 |
铅钡 |
无风化 |
0.633760513 |
0.009211053 |
0.003003604 |
|||||
| 51部位1 |
铅钡 |
风化 |
0.265795869 |
0 |
0 |
|||||
| 51部位2 |
铅钡 |
风化 |
0.307715674 |
0 |
0.003442689 |
|||||
| 52 |
铅钡 |
风化 |
0.537763437 |
0.008066956 |
0.003226782 |
|||||
| 53未风化点 |
铅钡 |
无风化 |
0.755402161 |
0 |
0.0015006 |
|||||
| 54 |
铅钡 |
风化 |
0.223537674 |
0 |
0.003210595 |
|||||
| 54严重风化点 |
铅钡 |
风化 |
0.198256862 |
0 |
0 |
|||||
| 55 |
铅钡 |
无风化 |
0.601320264 |
0 |
0.00230046 |
|||||
| 56 |
铅钡 |
风化 |
0.201763174 |
0 |
0 |
|||||
| 57 |
铅钡 |
风化 |
0.363199519 |
0 |
0.010511563 |
|||||
| 58 |
铅钡 |
风化 |
0.037240965 |
0 |
0.004004405 |
|||||
| 氧化钙(CaO) |
氧化镁(MgO) |
氧化铝(Al2O3) |
氧化铁(Fe2O3) |
氧化铜(CuO) |
氧化铅(PbO) |
|||||
| 0.074120341 |
0.01623985 |
0.067041432 |
0.02144493 |
0.022694149 |
0 |
|||||
| 0.03537762 |
0.005212377 |
0.020738605 |
0.003659754 |
0.012531884 |
0.487967173 |
|||||
| 0.076157911 |
0.018340068 |
0.077712154 |
0.027147446 |
0.033882499 |
0 |
|||||
| 0 |
0.015732648 |
0.031362468 |
0 |
0 |
0 |
|||||
| 0.064747464 |
0.008913021 |
0.040262268 |
0.017826042 |
0.039647577 |
0 |
|||||
| 0.088648869 |
0 |
0.062767475 |
0.029345832 |
0.048196454 |
0 |
|||||
| 0.084036175 |
0.005284016 |
0.062798496 |
0.004267859 |
0.010872879 |
0.001117773 |
|||||
| 0.047807552 |
0.012383272 |
0.062829882 |
0.024056029 |
0.033292732 |
0.010150223 |
|||||
| 0.009513207 |
0.005465034 |
0.025402287 |
0.002024087 |
0.015585467 |
0 |
|||||
| 0.055943293 |
0.015812432 |
0.027371865 |
0.004580153 |
0.008178844 |
0.559869138 |
|||||
| 0.013117953 |
0 |
0.020056375 |
0 |
0.008564614 |
0.447202949 |
|||||
| 0.007231094 |
0 |
0.014663051 |
0.002912524 |
0.016571256 |
0 |
|||||
| 0.010732197 |
0 |
0.019859579 |
0.001705115 |
0.032497492 |
0 |
|||||
| 0.014166757 |
0 |
0.023575214 |
0 |
0.012544609 |
0.487725749 |
|||||
| 0.006214293 |
0 |
0.01323043 |
0.003207377 |
0.015535732 |
0 |
|||||
| 0.054734925 |
0.017503035 |
0.101679482 |
0.061108863 |
0.022055848 |
0.003541076 |
|||||
| 0.059364887 |
0.011225728 |
0.055622977 |
0.02184466 |
0.051476537 |
0.014259709 |
|||||
| 0 |
0.020016175 |
0.112717347 |
0.024160938 |
0.02537404 |
0.002021836 |
|||||
| 0.083131313 |
0.006666667 |
0.093232323 |
0.005050505 |
0.004747475 |
0.016363636 |
|||||
| 0.05292925 |
0.02913243 |
0.035535162 |
0.019101483 |
0.002027532 |
0.470814214 |
|||||
| 0 |
0 |
0.06164461 |
0.017079516 |
0.054066282 |
0.10519172 |
|||||
| 0.0201 |
0 |
0.0406 |
0 |
0.0078 |
0.0025 |
|||||
| 0.002103998 |
0 |
0.008115419 |
0.002604949 |
0.00841599 |
0 |
|||||
| 0.003949693 |
0 |
0.014967259 |
0.001766968 |
0.001663029 |
0.229186155 |
|||||
| 0.011724424 |
0 |
0.015044615 |
0 |
0.008923013 |
0.34156464 |
|||||
| 0.005181347 |
0.007357513 |
0.014715026 |
0 |
0.030984456 |
0.175958549 |
|||||
| 0.024128401 |
0.005846088 |
0.012329932 |
0.002444728 |
0.007440476 |
0.504039116 |
|||||
| 0.055344318 |
0.009400084 |
0.023764259 |
0.008027038 |
0.056506126 |
0.632129278 |
|||||
| 0.0166 |
0.0064 |
0.035 |
0.0035 |
0.0055 |
0 |
|||||
| 0.032184857 |
0.005570456 |
0.042913142 |
0 |
0.007220961 |
0.315762327 |
|||||
| 0.00649083 |
0 |
0.01957552 |
0.015969503 |
0.011539254 |
0.328662683 |
|||||
| 0.008945096 |
0.006271849 |
0.03146206 |
0 |
0.006683117 |
0.261155665 |
|||||
| 0 |
0.011848825 |
0.057814096 |
0 |
0.027783453 |
0.205515832 |
|||||
| 0.045839714 |
0.010005105 |
0.044410413 |
0.021643696 |
0 |
0.400408372 |
|||||
| 0.008060402 |
0.011121314 |
0.036016733 |
0 |
0.027242118 |
0.223242526 |
|||||
| 0.021172638 |
0.012214984 |
0.066164495 |
0.012927524 |
0.004580619 |
0.234324104 |
|||||
| 0.037553761 |
0.012482954 |
0.055071856 |
0.012482954 |
0.014371132 |
0.422112661 |
|||||
| 0.007938931 |
0.011603053 |
0.061679389 |
0 |
0.005496183 |
0.139033079 |
|||||
| 0.03679631 |
0.007443128 |
0.028200021 |
0 |
0.051682566 |
0.266170458 |
|||||
| 0.008541794 |
0.007524914 |
0.050844011 |
0 |
0.005389465 |
0.162599146 |
|||||
| 0.047958115 |
0.01539267 |
0.056335079 |
0.028691099 |
0.007329843 |
0.357905759 |
|||||
| 0.066204614 |
0.009827247 |
0.035274646 |
0.014378815 |
0.015620151 |
0.462915072 |
|||||
| 0 |
0.011452745 |
0.037659926 |
0 |
0.013825836 |
0.603177879 |
|||||
| 0.007998359 |
0 |
0.016611977 |
0.004819524 |
0.015484003 |
0.477337982 |
|||||
| 0.016255207 |
0.009041959 |
0.031596058 |
0.046632124 |
0.004470182 |
0.168139795 |
|||||
| 0 |
0.016931968 |
0.048565345 |
0 |
0.007806955 |
0.2560073 |
|||||
| 0.003789819 |
0 |
0.016388405 |
0.003277681 |
0.006965072 |
0.426200963 |
|||||
| 0.032471498 |
0 |
0.01129886 |
0 |
0.031962541 |
0.330313518 |
|||||
| 0.004662477 |
0 |
0.023920535 |
0.01013582 |
0.00111494 |
0.200283803 |
|||||
| 0.01357787 |
0.010132739 |
0.047623873 |
0.004154423 |
0.003343804 |
0.173675144 |
|||||
| 0.04295846 |
0.005167173 |
0.039108409 |
0.027760892 |
0 |
0.382370821 |
|||||
| 0.004753236 |
0 |
0.016080097 |
0 |
0.085558252 |
0.294700647 |
|||||
| 0.018971289 |
0 |
0.004565284 |
0.001927564 |
0 |
0.712285685 |
|||||
| 0.006898651 |
0 |
0.026072842 |
0.002942072 |
0.007405904 |
0.500253627 |
|||||
| 0.021589992 |
0 |
0.128026634 |
0.007768362 |
0.004338176 |
0.137308313 |
|||||
| 0.029667882 |
0.005974079 |
0.036148238 |
0.013466991 |
0.035540705 |
0.433576347 |
|||||
| 0.029835803 |
0.014917901 |
0.143572287 |
0.008109732 |
0.007408891 |
0.123247897 |
|||||
| 0.011239368 |
0 |
0.005062778 |
0 |
0.00891049 |
0.617962738 |
|||||
| 0.035338194 |
0.00799919 |
0.03564196 |
0.008707979 |
0.031692993 |
0.398440664 |
|||||
| 0.028436019 |
0.01552889 |
0.137642432 |
0.010386206 |
0 |
0.158414843 |
|||||
| 0.006402561 |
0.010004002 |
0.023509404 |
0 |
0.004701881 |
0.161664666 |
|||||
| 0.032005619 |
0.01284238 |
0.041637403 |
0 |
0.008327481 |
0.55643624 |
|||||
| 0.014425967 |
0 |
0.007012623 |
0 |
0.105890603 |
0.295832498 |
|||||
| 0.00890178 |
0 |
0.027205441 |
0 |
0.030106021 |
0.172434487 |
|||||
| 0.014826688 |
0 |
0.013424163 |
0 |
0.104287718 |
0.287317171 |
|||||
| 0.023425768 |
0.011812994 |
0.057363099 |
0.018620483 |
0.002602863 |
0.474822305 |
|||||
| 0.030133146 |
0 |
0.011812994 |
0 |
0.036039644 |
0.299529482 |
|||||
| 氧化钡(BaO) |
五氧化二磷(P2O5) |
氧化锶(SrO) |
氧化锡(SnO2) |
二氧化硫(SO2) |
||||||
| 0 |
0.008224027 |
0 |
0 |
0.003747658 |
||||||
| 0.157480315 |
0.070311634 |
0.007319508 |
0 |
0 |
||||||
| 0 |
0.009739923 |
0.000621697 |
0 |
0.004869962 |
||||||
| 0 |
0.013984576 |
0.000719794 |
0.024267352 |
0 |
||||||
| 0 |
0.011986477 |
0 |
0 |
0.003995492 |
||||||
| 0 |
0.012940697 |
0 |
0 |
0 |
||||||
| 0 |
0 |
0.000406463 |
0 |
0 |
||||||
| 0.01999594 |
0.011165246 |
0 |
0 |
0 |
||||||
| 0 |
0.003643356 |
0 |
0 |
0 |
||||||
| 0 |
0.095419847 |
0 |
0 |
0 |
||||||
| 0.167497832 |
0.02753686 |
0 |
0 |
0 |
||||||
| 0 |
0.001506478 |
0 |
0 |
0 |
||||||
| 0 |
0.006118355 |
0 |
0 |
0 |
||||||
| 0.187087704 |
0 |
0 |
0 |
0 |
||||||
| 0 |
0.003508069 |
0 |
0 |
0 |
||||||
| 0.009813841 |
0.045528126 |
0.001214083 |
0 |
0 |
||||||
| 0.028923948 |
0.007079288 |
0.001011327 |
0 |
0 |
||||||
| 0.013950667 |
0.042256369 |
0.00111201 |
0 |
0 |
||||||
| 0 |
0.001616162 |
0 |
0 |
0 |
||||||
| 0.104151104 |
0.079607299 |
0.005015473 |
0 |
0 |
||||||
| 0.266372582 |
0.065037892 |
0 |
0 |
0 |
||||||
| 0 |
0.0066 |
0 |
0 |
0 |
||||||
| 0 |
0 |
0 |
0 |
0 |
||||||
| 0.059037522 |
0.004365451 |
0 |
0 |
0 |
||||||
| 0.082485993 |
0.003631459 |
0 |
0 |
0 |
||||||
| 0.122901554 |
0 |
0.003419689 |
0 |
0 |
||||||
| 0.091836735 |
0.060693027 |
0.004676871 |
0 |
0 |
||||||
| 0.076996198 |
0 |
0.006759611 |
0 |
0 |
||||||
| 0 |
0.0021 |
0 |
0 |
0 |
||||||
| 0.0641634 |
0.065401279 |
0.002372602 |
0 |
0 |
||||||
| 0.068514321 |
0.001957552 |
0.002060581 |
0 |
0 |
||||||
| 0.094900267 |
0.001028172 |
0.008739461 |
0 |
0 |
||||||
| 0.11113381 |
0 |
0 |
0 |
0 |
||||||
| 0.105053599 |
0 |
0.003573252 |
0.004083716 |
0 |
||||||
| 0.106825834 |
0.000816243 |
0.003571064 |
0 |
0 |
||||||
| 0.042650651 |
0.043973941 |
0.003053746 |
0 |
0 |
||||||
| 0.093779503 |
0.084968006 |
0.004091052 |
0.004930242 |
0 |
||||||
| 0.091501272 |
0 |
0.002748092 |
0 |
0 |
||||||
| 0.153160709 |
0.098333159 |
0.003878813 |
0 |
0 |
||||||
| 0.111450071 |
0 |
0.002338824 |
0 |
0 |
||||||
| 0.063874346 |
0.116230366 |
0.004816754 |
0 |
0 |
||||||
| 0.033722975 |
0.132719561 |
0.004861901 |
0 |
0 |
||||||
| 0 |
0.145790343 |
0.011555922 |
0 |
0 |
||||||
| 0.102543068 |
0.003486464 |
0.002255947 |
0 |
0 |
||||||
| 0.034745504 |
0.016458397 |
0.003047851 |
0 |
0 |
||||||
| 0.101997364 |
0.002027781 |
0.004359728 |
0 |
0 |
||||||
| 0.110929018 |
0.000716993 |
0.002253406 |
0 |
0 |
||||||
| 0.311685668 |
0.076954397 |
0.005394951 |
0 |
0.152992671 |
||||||
| 0.049462802 |
0.001723089 |
0 |
0 |
0 |
||||||
| 0.040936265 |
0.010538048 |
0.001215929 |
0.00233053 |
0 |
||||||
| 0.104863222 |
0.014285714 |
0.004863222 |
0.004457953 |
0 |
||||||
| 0.265271036 |
0.001415858 |
0.009203074 |
0 |
0 |
||||||
| 0.067870549 |
0.017956782 |
0.006898651 |
0 |
0 |
||||||
| 0.09932028 |
0.004869636 |
0.004159481 |
0 |
0 |
||||||
| 0.052663438 |
0 |
0.002623083 |
0 |
0 |
||||||
| 0.054171729 |
0.089408667 |
0.001923856 |
0 |
0 |
||||||
| 0.020324389 |
0.004104926 |
0.002503004 |
0 |
0 |
||||||
| 0.073106521 |
0.011745646 |
0.00617659 |
0 |
0 |
||||||
| 0.077561766 |
0.091028757 |
0.002430134 |
0 |
0 |
||||||
| 0.073711808 |
0.011092064 |
0.002520924 |
0.01320964 |
0 |
||||||
| 0.035514206 |
0.00130052 |
0 |
0 |
0 |
||||||
| 0.070633089 |
0.042540383 |
0.008829136 |
0 |
0 |
||||||
| 0.323081547 |
0.031356442 |
0.004508115 |
0 |
0.019635344 |
||||||
| 0.103420684 |
0.014602921 |
0.00310062 |
0 |
0.036607321 |
||||||
| 0.312863154 |
0.035964737 |
0.003706672 |
0 |
0.025846524 |
||||||
| 0 |
0.035739313 |
0.001902092 |
0 |
0 |
||||||
| 0.354890379 |
0.060466513 |
0.006206828 |
0 |
0.159675643 |
||||||
第1题第3小问预测值
| 文物采样点 |
类型 |
二氧化硅(SiO2) |
氧化钠(Na2O) |
氧化钾(K2O) |
氧化钙(CaO) |
氧化镁(MgO) |
氧化铝(Al2O3) |
氧化铁(Fe2O3) |
||||
| 02 |
铅钡 |
65.13 |
1.02 |
1.19 |
1.06 |
7.23 |
2.45 |
|||||
| 07 |
高钾 |
62.42 |
6.19 |
6.23 |
2.07 |
|||||||
| 08 |
铅钡 |
48.99 |
0.33 |
2.84 |
||||||||
| 08严重风化点 |
铅钡 |
33.46 |
2.04 |
2.61 |
||||||||
| 09 |
高钾 |
64.81 |
9.29 |
5.74 |
5.57 |
2.22 |
||||||
| 10 |
高钾 |
66.56 |
9.62 |
5.33 |
5.06 |
2.16 |
||||||
| 11 |
铅钡 |
62.44 |
0.18 |
2.36 |
0.59 |
4.19 |
||||||
| 12 |
高钾 |
64.08 |
9.71 |
5.84 |
5.71 |
2.19 |
||||||
| 19 |
铅钡 |
58.49 |
1.78 |
0.47 |
5.07 |
1.92 |
||||||
| 22 |
高钾 |
62.14 |
9.44 |
6.78 |
1.26 |
7.75 |
2.25 |
|||||
| 26 |
铅钡 |
48.64 |
0.29 |
2.2 |
||||||||
| 26严重风化点 |
铅钡 |
32.57 |
0.37 |
1.86 |
2.68 |
|||||||
| 27 |
高钾 |
62.51 |
6.06 |
1.16 |
6.76 |
2.1 |
||||||
| 34 |
铅钡 |
64.63 |
0.22 |
3.12 |
1.06 |
|||||||
| 36 |
铅钡 |
68.42 |
4.61 |
0.11 |
3.1 |
0.91 |
||||||
| 38 |
铅钡 |
61.78 |
3.77 |
4.07 |
0.88 |
|||||||
| 39 |
铅钡 |
55.1 |
2 |
|||||||||
| 40 |
铅钡 |
45.56 |
0.72 |
1.95 |
0.78 |
|||||||
| 41 |
铅钡 |
47.31 |
0.41 |
3.81 |
2.61 |
4.83 |
2.38 |
|||||
| 43部位1 |
铅钡 |
41.26 |
4.09 |
0.77 |
3.75 |
1.35 |
||||||
| 43部位2 |
铅钡 |
50.55 |
5.25 |
0.83 |
4.91 |
1.98 |
||||||
| 48 |
铅钡 |
82.18 |
3.19 |
0.29 |
1.67 |
1.42 |
15.15 |
1.62 |
||||
| 49 |
铅钡 |
57.64 |
3.43 |
1.35 |
6.88 |
3.33 |
||||||
| 50 |
铅钡 |
46.83 |
2.04 |
0.35 |
3.37 |
0.92 |
||||||
| 51部位1 |
铅钡 |
53.46 |
2.43 |
1.07 |
6.75 |
1.78 |
||||||
| 51部位2 |
铅钡 |
50.2 |
3.98 |
1.33 |
4.01 |
1.01 |
||||||
| 52 |
铅钡 |
54.59 |
3.61 |
1.12 |
0.43 |
2.66 |
0.82 |
|||||
| 54 |
铅钡 |
51.13 |
0.29 |
2.04 |
1.16 |
5.65 |
||||||
| 54严重风化点 |
铅钡 |
45.96 |
0.99 |
5.15 |
||||||||
| 56 |
铅钡 |
58 |
0.06 |
3.35 |
||||||||
| 57 |
铅钡 |
54.27 |
0.16 |
3.68 |
||||||||
| 58 |
铅钡 |
59.24 |
0.31 |
2.34 |
0.67 |
5.02 |
1.45 |
|||||
| 文物采样点 |
类型 |
氧化铜(CuO) |
氧化铅(PbO) |
氧化钡(BaO) |
五氧化二磷(P2O5) |
氧化锶(SrO) |
氧化锡(SnO2) |
二氧化硫(SO2) |
||||
| 02 |
铅钡 |
28.64 |
0.1 |
|||||||||
| 07 |
高钾 |
4.18 |
1.7 |
|||||||||
| 08 |
铅钡 |
9.68 |
9.89 |
27.67 |
0.28 |
|||||||
| 08严重风化点 |
铅钡 |
2.41 |
13.66 |
27.06 |
3.69 |
0.44 |
10.5 |
|||||
| 09 |
高钾 |
2.49 |
1.44 |
|||||||||
| 10 |
高钾 |
1.78 |
||||||||||
| 11 |
铅钡 |
4.2 |
6.6 |
11.05 |
5.51 |
0.28 |
||||||
| 12 |
高钾 |
2.59 |
1.24 |
|||||||||
| 19 |
铅钡 |
2.78 |
24.03 |
1.79 |
4.96 |
0.1 |
||||||
| 22 |
高钾 |
1.49 |
1.3 |
|||||||||
| 26 |
铅钡 |
9.84 |
10.74 |
28.69 |
0.36 |
|||||||
| 26严重风化点 |
铅钡 |
2.87 |
11.13 |
31.89 |
2.17 |
0.53 |
11.42 |
|||||
| 27 |
高钾 |
2.48 |
1.45 |
|||||||||
| 34 |
铅钡 |
0.78 |
27.76 |
6.44 |
0.13 |
|||||||
| 36 |
铅钡 |
22.82 |
7.27 |
0.13 |
||||||||
| 38 |
铅钡 |
0 |
30.52 |
6.23 |
0.32 |
|||||||
| 39 |
铅钡 |
0.15 |
42.24 |
3.66 |
0.52 |
|||||||
| 40 |
铅钡 |
51.42 |
3.13 |
0.59 |
||||||||
| 41 |
铅钡 |
25.33 |
6.2 |
3.59 |
0.38 |
|||||||
| 43部位1 |
铅钡 |
4.62 |
41.06 |
3.73 |
0.55 |
|||||||
| 43部位2 |
铅钡 |
0.78 |
25.96 |
8.96 |
0.38 |
|||||||
| 48 |
铅钡 |
3.75 |
0.16 |
0.84 |
||||||||
| 49 |
铅钡 |
15.39 |
2.54 |
7.23 |
0.37 |
|||||||
| 50 |
铅钡 |
0.4 |
25.21 |
10.64 |
2.47 |
0.57 |
||||||
| 51部位1 |
铅钡 |
0.64 |
21.45 |
5.38 |
4.23 |
0.3 |
||||||
| 51部位2 |
铅钡 |
0.02 |
32.55 |
4.88 |
||||||||
| 52 |
铅钡 |
28.63 |
5.08 |
1.84 |
0.35 |
|||||||
| 54 |
铅钡 |
0.1 |
36.67 |
3.48 |
0.37 |
0.79 |
||||||
| 54严重风化点 |
铅钡 |
0.61 |
39.67 |
10.26 |
1.03 |
|||||||
| 56 |
铅钡 |
0.06 |
22.46 |
11.89 |
||||||||
| 57 |
铅钡 |
0.43 |
26.31 |
13.74 |
||||||||
| 58 |
铅钡 |
2.4 |
20.56 |
4.1 |
0.15 |
|||||||
第一问第3小问的期望值差值
问题2第2小问
蓝绿色敏感性分析
| 类型 |
二氧化硅(SiO2) |
氧化钠(Na2O) |
氧化钾(K2O) |
氧化钙(CaO) |
氧化镁(MgO) |
氧化铝(Al2O3) |
氧化铁(Fe2O3) |
| 高钾 |
-25.10 |
0.71 |
8.97 |
4.57 |
0.90 |
4.80 |
1.70 |
| 铅钡 |
30.08 |
1.50 |
0.09 |
-1.46 |
-0.03 |
1.49 |
0.15 |
| 类型 |
氧化铜(CuO) |
氧化铅(PbO) |
氧化钡(BaO) |
五氧化二磷(P2O5) |
氧化锶(SrO) |
氧化锡(SnO2) |
二氧化硫(SO2) |
| 高钾 |
0.93 |
0.42 |
0.61 |
1.14 |
0.04 |
0.20 |
0.11 |
| 铅钡 |
-0.85 |
-22.31 |
-2.87 |
-4.38 |
-0.16 |
-0.02 |
-1.22 |
| 氧化铁(Fe2O3) |
0.0000 |
0.0000 |
0.0257 |
0.0027 |
0.0178 |
|
| 12.04% |
1.0000 |
1.0000 |
1.0257 |
1.0027 |
1.0178 |
|
| 1 |
12.04% |
12.04% |
12.01% |
12.04% |
12.02% |
浅绿色敏感性分析
| 氧化铅(PbO) |
0.1760 |
0.4675 |
0.0046 |
0.0000 |
0.0000 |
|
| 12.04% |
1.1760 |
1.4675 |
1.0046 |
1.0000 |
1.0000 |
|
| 1 |
11.88% |
11.61% |
12.03% |
12.04% |
12.04% |
| 氧化铁(Fe2O3) |
0.0000 |
0.0191 |
0.0060 |
|
| 33.02% |
1.0000 |
1.0191 |
1.0060 |
|
| 1 |
33.02% |
33.02% |
33.02% |
绿色敏感性分析
| 氧化铅(PbO) |
0.0000 |
0.4708 |
0.2147 |
|
| 33.02% |
1.0000 |
1.4708 |
1.2147 |
|
| 1 |
0.330248158 |
0.330248158 |
0.330248158 |
| 氧化铁(Fe2O3) |
0.0000 |
|
| 17.08% |
1.0000 |
|
| 1 |
17.08% |
| 氧化铅(PbO) |
0 |
0.3416 |
|
| 17.08% |
1.0000 |
1.3416 |
|
| 1 |
0.17078232 |
0.17078232 |
浅蓝敏感性分析
| 氧化铁(Fe2O3) |
0.0186 |
0.0037 |
0.0052 |
0.0160 |
0.0168 |
|
| 28.47% |
1.0186 |
1.0037 |
1.0052 |
1.0160 |
1.0168 |
|
| 1 |
28.47% |
28.47% |
28.47% |
28.47% |
28.47% |
| 氧化铅(PbO) |
0.4748 |
0.1787 |
0.5009 |
0.3287 |
0.0006 |
|
| 28.47% |
1.4748 |
1.1787 |
1.5009 |
1.3287 |
1.0006 |
|
| 1 |
28.47% |
28.47% |
28.47% |
28.47% |
28.47% |
深绿敏感性分析
| 氧化铁(Fe2O3) |
0.0028 |
0.0000 |
0.0051 |
|
| 22.70% |
1.0028 |
1.0000 |
1.0051 |
|
| 1 |
22.70% |
22.70% |
22.70% |
|
| 氧化铅(PbO) |
0.5054 |
0.1670 |
0.0164 |
|
| 22.70% |
1.5054 |
1.1670 |
1.0164 |
|
| 1.00% |
22.70% |
22.70% |
22.70% |
深蓝敏感性分析
| 氧化铁(Fe2O3) |
0.0247 |
0.0000 |
|
| 18.33% |
1.0247 |
1.0000 |
|
| 1 |
18.33% |
18.33% |
| 氧化铅(PbO) |
0.3914 |
0.0000 |
|
| 0.1833 |
1.3914 |
1.0000 |
|
| 1 |
18.33% |
18.33% |
第3问第1小问
| 文物编号 |
表面风化 |
类型 |
二氧化硅(SiO2) |
氧化钠(Na2O) |
氧化钾(K2O) |
氧化钙(CaO) |
氧化镁(MgO) |
| A1 |
无风化 |
铅钡 |
0.788600724 |
0.061117813 |
0.018697226 |
||
| A2 |
风化 |
铅钡 |
0.392085584 |
0.079248027 |
|||
| A3 |
无风化 |
铅钡 |
0.322792483 |
0.01374015 |
0.072640938 |
0.008183471 |
|
| A4 |
无风化 |
铅钡 |
0.369479167 |
0.008229167 |
0.030104167 |
0.0109375 |
|
| A5 |
风化 |
铅钡 |
0.645352339 |
0.012045774 |
0.003714114 |
0.016462558 |
0.023489259 |
| A6 |
风化 |
高钾 |
0.940161453 |
0.013622603 |
0.006458123 |
0.002119072 |
|
| A7 |
风化 |
高钾 |
0.911581694 |
0.009835407 |
0.011240466 |
||
| A8 |
无风化 |
铅钡 |
0.51130226 |
0.00230046 |
0.00890178 |
||
| 文物编号 |
表面风化 |
类型 |
氧化铝(Al2O3) |
氧化铁(Fe2O3) |
氧化铜(CuO) |
氧化铅(PbO) |
氧化钡(BaO) |
| A1 |
无风化 |
铅钡 |
0.072677925 |
0.021612384 |
0.021210294 |
||
| A2 |
风化 |
铅钡 |
0.024200249 |
0.356252597 |
|||
| A3 |
无风化 |
铅钡 |
0.02960194 |
0.071327541 |
0.002121641 |
0.399878763 |
0.04738331 |
| A4 |
无风化 |
铅钡 |
0.073645833 |
0.0671875 |
0.01 |
0.252916667 |
0.0865625 |
| A5 |
风化 |
铅钡 |
0.127986348 |
0.008130897 |
0.009435856 |
0.122766513 |
0.021682393 |
| A6 |
风化 |
高钾 |
0.015338042 |
0.002724521 |
0.017457114 |
||
| A7 |
风化 |
高钾 |
0.050782818 |
0.002408671 |
0.011742272 |
||
| A8 |
无风化 |
铅钡 |
0.021204241 |
0.090118024 |
0.212442488 |
0.113422685 |
|
| 文物编号 |
表面风化 |
类型 |
五氧化二磷(P2O5) |
氧化锶(SrO) |
氧化锡(SnO2) |
二氧化硫(SO2) |
|
| A1 |
无风化 |
铅钡 |
0.010655408 |
0.000301568 |
0.005126659 |
||
| A2 |
风化 |
铅钡 |
0.148213544 |
||||
| A3 |
无风化 |
铅钡 |
0.027076177 |
0.005253587 |
|||
| A4 |
无风化 |
铅钡 |
0.088020833 |
0.002916667 |
|||
| A5 |
风化 |
铅钡 |
0.001907248 |
0.00210801 |
0.004918691 |
||
| A6 |
风化 |
高钾 |
0.002119072 |
||||
| A7 |
风化 |
高钾 |
0.001304697 |
0.001103974 |
|||
| A8 |
无风化 |
铅钡 |
0.014602921 |
0.00310062 |
0.022604521 |
%问题一第二小问matlab代码
clear
clc
%高钾玻璃
gjfh=[92.72 0 0 0.94 0.54 2.51 0.2 1.54 0 0 0.36 0 0 0
94.29 0 1.01 0.72 0 1.46 0.29 1.65 0 0 0.15 0 0 0
92.63 0 0 1.07 0 1.98 0.17 3.24 0 0 0.61 0 0 0
95.02 0 0.59 0.62 0 1.32 0.32 1.55 0 0 0.35 0 0 0
96.77 0 0.92 0.21 0 0.81 0.26 0.84 0 0 0 0 0 0
92.35 0 0.74 1.66 0.64 3.5 0.35 0.55 0 0 0.21 0 0 0];
gjfhcfzh=zeros();gjfhcfzb=zeros();gjfhcfzbqwz=zeros();
[m,n]=size(gjfh);
for i=1:m
su=0;
for j=1:n
su=su+gjfh(i,j);
end
gjfhcfzh(i,1)=su;
end
for i=1:m
for j=1:n
gjfhcfzb(i,j)=gjfh(i,j)./gjfhcfzh(i,1);
end
end
for j=1:n
su=0;k=0;
for i=1:m
if gjfh(i,j)>0
k=k+1;
su=su+gjfhcfzb(i,j);
end
end
if su==0
k=1;
end
gjfhcfzbqwz(j)=su./k;
end
gjwfh=[65.88 0 9.67 7.12 1.56 6.44 2.06 2.18 0 0 0.79 0 0 0.36
61.58 0 10.95 7.35 1.77 7.5 2.62 3.27 0 0 0.94 0.06 0 0.47
79.46 0 9.42 0 1.53 3.05 0 0 0 0 1.36 0.07 2.36 0
69.33 0 9.99 6.32 0.87 3.93 1.74 3.87 0 0 1.17 0 0 0.39
59.01 2.86 12.53 8.7 0 6.16 2.88 4.73 0 0 1.27 0 0 0
65.18 2.1 14.52 8.27 0.52 6.18 0.42 1.07 0.11 0 0 0.04 0 0
76.68 0 0 4.71 1.22 6.19 2.37 3.28 1 1.97 1.1 0 0 0
59.81 0 7.68 5.41 1.73 10.05 6.04 2.18 0.35 0.97 4.5 0.12 0 0
61.71 0 12.37 5.87 1.11 5.5 2.16 5.09 1.41 2.86 0.7 0.1 0 0
67.65 0 7.37 0 1.98 11.15 2.39 2.51 0.2 1.38 4.18 0.11 0 0
62.47 3.38 12.28 8.23 0.66 9.23 0.5 0.47 1.62 0 0.16 0 0 0
87.05 0 5.19 2.01 0 4.06 0 0.78 0.25 0 0.66 0 0 0];
gjwfhcfzh=zeros();gjwfhcfzb=zeros();gjwfhcfzbqwz=zeros();
[m,n]=size(gjwfh);
for i=1:m
su=0;
for j=1:n
su=su+gjwfh(i,j);
end
gjwfhcfzh(i,1)=su;
end
for i=1:m
for j=1:n
gjwfhcfzb(i,j)=gjwfh(i,j)./gjwfhcfzh(i,1);
end
end
for j=1:n
su=0;k=0;
for i=1:m
if gjwfh(i,j)>0
k=k+1;
su=su+gjwfhcfzb(i,j);
end
end
if su==0
k=1;
end
gjwfhcfzbqwz(j)=su./k;
end
%铅钡玻璃
qbfh=[17.98 0 0 3.19 0.47 1.87 0.33 1.13 44 14.2 6.34 0.66 0 0
21.35 0 0 5.13 1.45 2.51 0.42 0.75 51.34 0 8.75 0 0 0
29.15 0 0 1.21 0 1.85 0 0.79 41.25 15.45 2.54 0 0 0
25.42 0 0 1.31 0 2.18 0 1.16 45.1 17.3 0 0 0 0
18.46 0 0.44 4.96 2.73 3.33 1.79 0.19 44.12 9.76 7.46 0.47 0 0
25.74 1.22 0 2.27 0.55 1.16 0.23 0.7 47.42 8.64 5.71 0.44 0 0
12.41 0 0 5.24 0.89 2.25 0.76 5.35 59.85 7.29 0 0.64 0 0
24.61 0 0 3.58 1.19 5.25 1.19 1.37 40.24 8.94 8.1 0.39 0.47 0
33.59 0 0.21 3.51 0.71 2.69 0 4.93 25.39 14.61 9.38 0.37 0 0
28.79 0 0 4.58 1.47 5.38 2.74 0.7 34.18 6.1 11.1 0.46 0 0
21.7 0 0 6.4 0.95 3.41 1.39 1.51 44.75 3.26 12.83 0.47 0 0
17.11 0 0 0 1.11 3.65 0 1.34 58.46 0 14.13 1.12 0 0
35.78 0 0.25 0.78 0 1.62 0.47 1.51 46.55 10 0.34 0.22 0 0
39.57 2.22 0.14 0.37 0 1.6 0.32 0.68 41.61 10.83 0.07 0.22 0 0
4.61 0 0 3.19 0 1.11 0 3.14 32.45 30.62 7.56 0.53 0 15.03
16.71 0 0 1.87 0 0.45 0.19 0 70.21 6.69 1.77 0.68 0 0
32.93 1.38 0 0.68 0 2.57 0.29 0.73 49.31 9.79 0.48 0.41 0 0
29.64 0 0 2.93 0.59 3.57 1.33 3.51 42.82 5.35 8.83 0.19 0 0
26.25 0 0 1.11 0 0.5 0 0.88 61.03 7.22 1.16 0.61 0 0
30.39 0 0.34 3.49 0.79 3.52 0.86 3.13 39.35 7.66 8.99 0.24 0 0
53.33 0.8 0.32 2.82 1.54 13.65 1.03 0 15.71 7.31 1.1 0.25 1.31 0
22.28 0 0.32 3.19 1.28 4.15 0 0.83 55.46 7.04 4.24 0.88 0 0
19.79 0 0 1.44 0 0.7 0 10.57 29.53 32.25 3.13 0.45 0 1.96
20.14 0 0 1.48 0 1.34 0 10.41 28.68 31.23 3.59 0.37 0 2.58
36.28 0 1.05 2.34 1.18 5.73 1.86 0.26 47.43 0 3.57 0.19 0 0
3.72 0 0.4 3.01 0 1.18 0 3.6 29.92 35.45 6.04 0.62 0 15.95];
qbfhcfzh=zeros();qbfhcfzb=zeros();qbfhcfzbqwz=zeros();
[m,n]=size(qbfh);
for i=1:m
su=0;
for j=1:n
su=su+qbfh(i,j);
end
qbfhcfzh(i,1)=su;
end
for i=1:m
for j=1:n
qbfhcfzb(i,j)=qbfh(i,j)./qbfhcfzh(i,1);
end
end
for j=1:n
su=0;k=0;
for i=1:m
if qbfh(i,j)>0
k=k+1;
su=su+qbfhcfzb(i,j);
end
end
if su==0
k=1;
end
qbfhcfzbqwz(j)=su./k;
end
qbwfh=[37.36 0 0.71 0 0 5.45 1.51 4.78 9.3 23.55 5.75 0 0 0
65.91 0 0 0.38 0 1.44 0.17 0.16 22.05 5.68 0.42 0 0 0
49.01 2.71 0 1.13 0 1.45 0 0.86 32.92 7.95 0.35 0 0 0
53.79 7.92 0 0.5 0.71 1.42 0 2.99 16.98 11.86 0 0.33 0 0
45.02 0 0 3.12 0.54 4.16 0 0.7 30.61 6.22 6.34 0.23 0 0
50.61 2.31 0 0.63 0 1.9 1.55 1.12 31.9 6.65 0.19 0.2 0 0
51.54 4.66 0.29 0.87 0.61 3.06 0 0.65 25.4 9.23 0.1 0.85 0 0
51.33 5.68 0.35 0 1.16 5.66 0 2.72 20.12 10.88 0 0 0 0
34.34 0 1.41 4.49 0.98 4.35 2.12 0 39.22 10.29 0 0.35 0.4 0
51.26 5.74 0.15 0.79 1.09 3.53 0 2.67 21.88 10.47 0.08 0.35 0 0
54.61 0 0.3 2.08 1.2 6.5 1.27 0.45 23.02 4.19 4.32 0.3 0 0
63.66 3.04 0.11 0.78 1.14 6.06 0 0.54 13.66 8.99 0 0.27 0 0
61.28 2.66 0.11 0.84 0.74 5 0 0.53 15.99 10.96 0 0.23 0 0
65.91 0 0 1.6 0.89 3.11 4.59 0.44 16.55 3.42 1.62 0.3 0 0
55.21 0 0.25 0 1.67 4.79 0 0.77 25.25 10.06 0.2 0.43 0 0
69.71 0 0.21 0.46 0 2.36 1 0.11 19.76 4.88 0.17 0 0 0
68.08 0 0.26 1.34 1 4.7 0.41 0.33 17.14 4.04 1.04 0.12 0.23 0
36.93 0 0 4.24 0.51 3.86 2.74 0 37.74 10.35 1.41 0.48 0.44 0
31.94 0 0 0.47 0 1.59 0 8.46 29.14 26.23 0.14 0.91 0 0
60.74 3.06 0.2 2.14 0 12.69 0.77 0.43 13.61 5.22 0 0.26 0 0
63.3 0.92 0.3 2.98 1.49 14.34 0.81 0.74 12.31 2.03 0.41 0.25 0 0
75.51 0 0.15 0.64 1 2.35 0 0.47 16.16 3.55 0.13 0 0 0
60.12 0 0.23 0.89 0 2.72 0 3.01 17.24 10.34 1.46 0.31 0 3.66];
qbwfhcfzh=zeros();qbwfhcfzb=zeros();qbwfhcfzbqwz=zeros();
[m,n]=size(qbwfh);
for i=1:m
su=0;
for j=1:n
su=su+qbwfh(i,j);
end
qbwfhcfzh(i,1)=su;
end
for i=1:m
for j=1:n
qbwfhcfzb(i,j)=qbwfh(i,j)./qbwfhcfzh(i,1);
end
end
for j=1:n
su=0;k=0;
for i=1:m
if qbwfh(i,j)>0
k=k+1;
su=su+qbwfhcfzb(i,j);
end
end
if su==0
k=1;
end
qbwfhcfzbqwz(j)=su./k;
end
fhfx=zeros();
for i=1:14
if gjfhcfzbqwz(i)>=qbfhcfzbqwz(i)
fhfx(i)=0;
else
fhfx(i)=1;
end
end
wfhfx=zeros();
for i=1:14
if gjwfhcfzbqwz(i)>=qbwfhcfzbqwz(i)
wfhfx(i)=0;
else
wfhfx(i)=1;
end
end
gjfhljh=sum(gjfhcfzbqwz,'all');
gjwfhljh=sum(gjwfhcfzbqwz,'all');
qbfhljh=sum(qbfhcfzbqwz,'all');
qbwfhljh=sum(qbwfhcfzbqwz,'all');
fhgj=zeros();
for i=1:14
fhgj(i)=gjfhcfzbqwz(i)./gjfhljh;
end
wfhgj=zeros();
for i=1:14
wfhgj(i)=gjwfhcfzbqwz(i)./gjwfhljh;
end
fhqb=zeros();
for i=1:14
fhqb(i)=qbfhcfzbqwz(i)./qbfhljh;
end
wfhqb=zeros();
for i=1:14
wfhqb(i)=qbwfhcfzbqwz(i)./qbwfhljh;
end
gjcz=zeros();
for i=1:14
gjcz(i)=wfhgj(i)-fhgj(i);
end
qbcz=zeros();
for i=1:14
qbcz(i)=wfhqb(i)-fhqb(i);
end
%问题二第一小问matlab代码
clear
clc
%高钾玻璃
gjfh=[92.72 0 0 0.94 0.54 2.51 0.2 1.54 0 0 0.36 0 0 0
94.29 0 1.01 0.72 0 1.46 0.29 1.65 0 0 0.15 0 0 0
92.63 0 0 1.07 0 1.98 0.17 3.24 0 0 0.61 0 0 0
95.02 0 0.59 0.62 0 1.32 0.32 1.55 0 0 0.35 0 0 0
96.77 0 0.92 0.21 0 0.81 0.26 0.84 0 0 0 0 0 0
92.35 0 0.74 1.66 0.64 3.5 0.35 0.55 0 0 0.21 0 0 0];
gjfhcfzh=zeros();gjfhcfzb=zeros();gjfhcfzbqwz=zeros();
[m,n]=size(gjfh);
for i=1:m
sum=0;
for j=1:n
sum=sum+gjfh(i,j);
end
gjfhcfzh(i,1)=sum;
end
for i=1:m
for j=1:n
gjfhcfzb(i,j)=gjfh(i,j)./gjfhcfzh(i,1);
end
end
for j=1:n
sum=0;k=0;
for i=1:m
if gjfh(i,j)>0
k=k+1;
sum=sum+gjfhcfzb(i,j);
end
end
gjfhcfzbqwz(j)=sum./k;
end
gjwfh=[65.88 0 9.67 7.12 1.56 6.44 2.06 2.18 0 0 0.79 0 0 0.36
61.58 0 10.95 7.35 1.77 7.5 2.62 3.27 0 0 0.94 0.06 0 0.47
79.46 0 9.42 0 1.53 3.05 0 0 0 0 1.36 0.07 2.36 0
69.33 0 9.99 6.32 0.87 3.93 1.74 3.87 0 0 1.17 0 0 0.39
59.01 2.86 12.53 8.7 0 6.16 2.88 4.73 0 0 1.27 0 0 0
65.18 2.1 14.52 8.27 0.52 6.18 0.42 1.07 0.11 0 0 0.04 0 0
76.68 0 0 4.71 1.22 6.19 2.37 3.28 1 1.97 1.1 0 0 0
59.81 0 7.68 5.41 1.73 10.05 6.04 2.18 0.35 0.97 4.5 0.12 0 0
61.71 0 12.37 5.87 1.11 5.5 2.16 5.09 1.41 2.86 0.7 0.1 0 0
67.65 0 7.37 0 1.98 11.15 2.39 2.51 0.2 1.38 4.18 0.11 0 0
62.47 3.38 12.28 8.23 0.66 9.23 0.5 0.47 1.62 0 0.16 0 0 0
87.05 0 5.19 2.01 0 4.06 0 0.78 0.25 0 0.66 0 0 0];
gjwfhcfzh=zeros();gjwfhcfzb=zeros();gjwfhcfzbqwz=zeros();
[m,n]=size(gjwfh);
for i=1:m
sum=0;
for j=1:n
sum=sum+gjwfh(i,j);
end
gjwfhcfzh(i,1)=sum;
end
for i=1:m
for j=1:n
gjwfhcfzb(i,j)=gjwfh(i,j)./gjwfhcfzh(i,1);
end
end
for j=1:n
sum=0;k=0;
for i=1:m
if gjwfh(i,j)>0
k=k+1;
sum=sum+gjwfhcfzb(i,j);
end
end
gjwfhcfzbqwz(j)=sum./k;
end
%铅钡玻璃
qbfh=[17.98 0 0 3.19 0.47 1.87 0.33 1.13 44 14.2 6.34 0.66 0 0
21.35 0 0 5.13 1.45 2.51 0.42 0.75 51.34 0 8.75 0 0 0
29.15 0 0 1.21 0 1.85 0 0.79 41.25 15.45 2.54 0 0 0
25.42 0 0 1.31 0 2.18 0 1.16 45.1 17.3 0 0 0 0
18.46 0 0.44 4.96 2.73 3.33 1.79 0.19 44.12 9.76 7.46 0.47 0 0
25.74 1.22 0 2.27 0.55 1.16 0.23 0.7 47.42 8.64 5.71 0.44 0 0
12.41 0 0 5.24 0.89 2.25 0.76 5.35 59.85 7.29 0 0.64 0 0
24.61 0 0 3.58 1.19 5.25 1.19 1.37 40.24 8.94 8.1 0.39 0.47 0
33.59 0 0.21 3.51 0.71 2.69 0 4.93 25.39 14.61 9.38 0.37 0 0
28.79 0 0 4.58 1.47 5.38 2.74 0.7 34.18 6.1 11.1 0.46 0 0
21.7 0 0 6.4 0.95 3.41 1.39 1.51 44.75 3.26 12.83 0.47 0 0
17.11 0 0 0 1.11 3.65 0 1.34 58.46 0 14.13 1.12 0 0
35.78 0 0.25 0.78 0 1.62 0.47 1.51 46.55 10 0.34 0.22 0 0
39.57 2.22 0.14 0.37 0 1.6 0.32 0.68 41.61 10.83 0.07 0.22 0 0
4.61 0 0 3.19 0 1.11 0 3.14 32.45 30.62 7.56 0.53 0 15.03
16.71 0 0 1.87 0 0.45 0.19 0 70.21 6.69 1.77 0.68 0 0
32.93 1.38 0 0.68 0 2.57 0.29 0.73 49.31 9.79 0.48 0.41 0 0
29.64 0 0 2.93 0.59 3.57 1.33 3.51 42.82 5.35 8.83 0.19 0 0
26.25 0 0 1.11 0 0.5 0 0.88 61.03 7.22 1.16 0.61 0 0
30.39 0 0.34 3.49 0.79 3.52 0.86 3.13 39.35 7.66 8.99 0.24 0 0
53.33 0.8 0.32 2.82 1.54 13.65 1.03 0 15.71 7.31 1.1 0.25 1.31 0
22.28 0 0.32 3.19 1.28 4.15 0 0.83 55.46 7.04 4.24 0.88 0 0
19.79 0 0 1.44 0 0.7 0 10.57 29.53 32.25 3.13 0.45 0 1.96
20.14 0 0 1.48 0 1.34 0 10.41 28.68 31.23 3.59 0.37 0 2.58
36.28 0 1.05 2.34 1.18 5.73 1.86 0.26 47.43 0 3.57 0.19 0 0
3.72 0 0.4 3.01 0 1.18 0 3.6 29.92 35.45 6.04 0.62 0 15.95];
qbfhcfzh=zeros();qbfhcfzb=zeros();qbfhcfzbqwz=zeros();
[m,n]=size(qbfh);
for i=1:m
sum=0;
for j=1:n
sum=sum+qbfh(i,j);
end
qbfhcfzh(i,1)=sum;
end
for i=1:m
for j=1:n
qbfhcfzb(i,j)=qbfh(i,j)./qbfhcfzh(i,1);
end
end
for j=1:n
sum=0;k=0;
for i=1:m
if qbfh(i,j)>0
k=k+1;
sum=sum+qbfhcfzb(i,j);
end
end
qbfhcfzbqwz(j)=sum./k;
end
qbwfh=[37.36 0 0.71 0 0 5.45 1.51 4.78 9.3 23.55 5.75 0 0 0
65.91 0 0 0.38 0 1.44 0.17 0.16 22.05 5.68 0.42 0 0 0
49.01 2.71 0 1.13 0 1.45 0 0.86 32.92 7.95 0.35 0 0 0
53.79 7.92 0 0.5 0.71 1.42 0 2.99 16.98 11.86 0 0.33 0 0
45.02 0 0 3.12 0.54 4.16 0 0.7 30.61 6.22 6.34 0.23 0 0
50.61 2.31 0 0.63 0 1.9 1.55 1.12 31.9 6.65 0.19 0.2 0 0
51.54 4.66 0.29 0.87 0.61 3.06 0 0.65 25.4 9.23 0.1 0.85 0 0
51.33 5.68 0.35 0 1.16 5.66 0 2.72 20.12 10.88 0 0 0 0
34.34 0 1.41 4.49 0.98 4.35 2.12 0 39.22 10.29 0 0.35 0.4 0
51.26 5.74 0.15 0.79 1.09 3.53 0 2.67 21.88 10.47 0.08 0.35 0 0
54.61 0 0.3 2.08 1.2 6.5 1.27 0.45 23.02 4.19 4.32 0.3 0 0
63.66 3.04 0.11 0.78 1.14 6.06 0 0.54 13.66 8.99 0 0.27 0 0
61.28 2.66 0.11 0.84 0.74 5 0 0.53 15.99 10.96 0 0.23 0 0
65.91 0 0 1.6 0.89 3.11 4.59 0.44 16.55 3.42 1.62 0.3 0 0
55.21 0 0.25 0 1.67 4.79 0 0.77 25.25 10.06 0.2 0.43 0 0
69.71 0 0.21 0.46 0 2.36 1 0.11 19.76 4.88 0.17 0 0 0
68.08 0 0.26 1.34 1 4.7 0.41 0.33 17.14 4.04 1.04 0.12 0.23 0
36.93 0 0 4.24 0.51 3.86 2.74 0 37.74 10.35 1.41 0.48 0.44 0
31.94 0 0 0.47 0 1.59 0 8.46 29.14 26.23 0.14 0.91 0 0
60.74 3.06 0.2 2.14 0 12.69 0.77 0.43 13.61 5.22 0 0.26 0 0
63.3 0.92 0.3 2.98 1.49 14.34 0.81 0.74 12.31 2.03 0.41 0.25 0 0
75.51 0 0.15 0.64 1 2.35 0 0.47 16.16 3.55 0.13 0 0 0
60.12 0 0.23 0.89 0 2.72 0 3.01 17.24 10.34 1.46 0.31 0 3.66];
qbwfhcfzh=zeros();qbwfhcfzb=zeros();qbwfhcfzbqwz=zeros();
[m,n]=size(qbwfh);
for i=1:m
sum=0;
for j=1:n
sum=sum+qbwfh(i,j);
end
qbwfhcfzh(i,1)=sum;
end
for i=1:m
for j=1:n
qbwfhcfzb(i,j)=qbwfh(i,j)./qbwfhcfzh(i,1);
end
end
for j=1:n
sum=0;k=0;
for i=1:m
if qbwfh(i,j)>0
k=k+1;
sum=sum+qbwfhcfzb(i,j);
end
end
qbwfhcfzbqwz(j)=sum./k;
end
fhfx=zeros();
for i=1:14
if gjfhcfzbqwz(i)>=qbfhcfzbqwz(i)
fhfx(i)=0;
else
fhfx(i)=1;
end
end
wfhfx=zeros();
for i=1:14
if gjwfhcfzbqwz(i)>=qbwfhcfzbqwz(i)
wfhfx(i)=0;
else
wfhfx(i)=1;
end
end
%问题二第二小问的matlab代码
%初始数据归一化处理
clear
clc
z=[69.33 0 9.99 6.32 0.87 3.93 1.74 3.87 0 0 1.17 0 0 0.39
36.28 0 1.05 2.34 1.18 5.73 1.86 0.26 47.43 0 3.57 0.19 0 0
87.05 0 5.19 2.01 0 4.06 0 0.78 0.25 0 0.66 0 0 0
61.71 0 12.37 5.87 1.11 5.5 2.16 5.09 1.41 2.86 0.7 0.1 0 0
65.88 0 9.67 7.12 1.56 6.44 2.06 2.18 0 0 0.79 0 0 0.36
61.58 0 10.95 7.35 1.77 7.5 2.62 3.27 0 0 0.94 0.06 0 0.47
67.65 0 7.37 0 1.98 11.15 2.39 2.51 0.2 1.38 4.18 0.11 0 0
59.81 0 7.68 5.41 1.73 10.05 6.04 2.18 0.35 0.97 4.5 0.12 0 0
92.63 0 0 1.07 0 1.98 0.17 3.24 0 0 0.61 0 0 0
20.14 0 0 1.48 0 1.34 0 10.41 28.68 31.23 3.59 0.37 0 2.58
4.61 0 0 3.19 0 1.11 0 3.14 32.45 30.62 7.56 0.53 0 15.03
95.02 0 0.59 0.62 0 1.32 0.32 1.55 0 0 0.35 0 0 0
96.77 0 0.92 0.21 0 0.81 0.26 0.84 0 0 0 0 0 0
33.59 0 0.21 3.51 0.71 2.69 0 4.93 25.39 14.61 9.38 0.37 0 0
94.29 0 1.01 0.72 0 1.46 0.29 1.65 0 0 0.15 0 0 0
59.01 2.86 12.53 8.7 0 6.16 2.88 4.73 0 0 1.27 0 0 0
62.47 3.38 12.28 8.23 0.66 9.23 0.5 0.47 1.62 0 0.16 0 0 0
65.18 2.1 14.52 8.27 0.52 6.18 0.42 1.07 0.11 0 0 0.04 0 0
79.46 0 9.42 0 1.53 3.05 0 0 0 0 1.36 0.07 2.36 0
29.64 0 0 2.93 0.59 3.57 1.33 3.51 42.82 5.35 8.83 0.19 0 0
53.33 0.8 0.32 2.82 1.54 13.65 1.03 0 15.71 7.31 1.1 0.25 1.31 0
76.68 0 0 4.71 1.22 6.19 2.37 3.28 1 1.97 1.1 0 0 0
92.35 0 0.74 1.66 0.64 3.5 0.35 0.55 0 0 0.21 0 0 0
28.79 0 0 4.58 1.47 5.38 2.74 0.7 34.18 6.1 11.1 0.46 0 0
31.94 0 0 0.47 0 1.59 0 8.46 29.14 26.23 0.14 0.91 0 0
50.61 2.31 0 0.63 0 1.9 1.55 1.12 31.9 6.65 0.19 0.2 0 0
19.79 0 0 1.44 0 0.7 0 10.57 29.53 32.25 3.13 0.45 0 1.96
3.72 0 0.4 3.01 0 1.18 0 3.6 29.92 35.45 6.04 0.62 0 15.95
92.72 0 0 0.94 0.54 2.51 0.2 1.54 0 0 0.36 0 0 0
17.98 0 0 3.19 0.47 1.87 0.33 1.13 44 14.2 6.34 0.66 0 0
37.36 0 0.71 0 0 5.45 1.51 4.78 9.3 23.55 5.75 0 0 0
53.79 7.92 0 0.5 0.71 1.42 0 2.99 16.98 11.86 0 0.33 0 0
68.08 0 0.26 1.34 1 4.7 0.41 0.33 17.14 4.04 1.04 0.12 0.23 0
65.91 0 0 1.6 0.89 3.11 4.59 0.44 16.55 3.42 1.62 0.3 0 0
69.71 0 0.21 0.46 0 2.36 1 0.11 19.76 4.88 0.17 0 0 0
75.51 0 0.15 0.64 1 2.35 0 0.47 16.16 3.55 0.13 0 0 0
35.78 0 0.25 0.78 0 1.62 0.47 1.51 46.55 10 0.34 0.22 0 0
65.91 0 0 0.38 0 1.44 0.17 0.16 22.05 5.68 0.42 0 0 0
39.57 2.22 0.14 0.37 0 1.6 0.32 0.68 41.61 10.83 0.07 0.22 0 0
60.12 0 0.23 0.89 0 2.72 0 3.01 17.24 10.34 1.46 0.31 0 3.66
32.93 1.38 0 0.68 0 2.57 0.29 0.73 49.31 9.79 0.48 0.41 0 0
26.25 0 0 1.11 0 0.5 0 0.88 61.03 7.22 1.16 0.61 0 0
16.71 0 0 1.87 0 0.45 0.19 0 70.21 6.69 1.77 0.68 0 0
18.46 0 0.44 4.96 2.73 3.33 1.79 0.19 44.12 9.76 7.46 0.47 0 0
63.3 0.92 0.3 2.98 1.49 14.34 0.81 0.74 12.31 2.03 0.41 0.25 0 0
34.34 0 1.41 4.49 0.98 4.35 2.12 0 39.22 10.29 0 0.35 0.4 0
12.41 0 0 5.24 0.89 2.25 0.76 5.35 59.85 7.29 0 0.64 0 0
21.7 0 0 6.4 0.95 3.41 1.39 1.51 44.75 3.26 12.83 0.47 0 0
36.93 0 0 4.24 0.51 3.86 2.74 0 37.74 10.35 1.41 0.48 0.44 0
51.26 5.74 0.15 0.79 1.09 3.53 0 2.67 21.88 10.47 0.08 0.35 0 0
51.33 5.68 0.35 0 1.16 5.66 0 2.72 20.12 10.88 0 0 0 0
60.74 3.06 0.2 2.14 0 12.69 0.77 0.43 13.61 5.22 0 0.26 0 0
61.28 2.66 0.11 0.84 0.74 5 0 0.53 15.99 10.96 0 0.23 0 0
55.21 0 0.25 0 1.67 4.79 0 0.77 25.25 10.06 0.2 0.43 0 0
51.54 4.66 0.29 0.87 0.61 3.06 0 0.65 25.4 9.23 0.1 0.85 0 0
54.61 0 0.3 2.08 1.2 6.5 1.27 0.45 23.02 4.19 4.32 0.3 0 0
45.02 0 0 3.12 0.54 4.16 0 0.7 30.61 6.22 6.34 0.23 0 0
24.61 0 0 3.58 1.19 5.25 1.19 1.37 40.24 8.94 8.1 0.39 0.47 0
21.35 0 0 5.13 1.45 2.51 0.42 0.75 51.34 0 8.75 0 0 0
25.74 1.22 0 2.27 0.55 1.16 0.23 0.7 47.42 8.64 5.71 0.44 0 0
63.66 3.04 0.11 0.78 1.14 6.06 0 0.54 13.66 8.99 0 0.27 0 0
22.28 0 0.32 3.19 1.28 4.15 0 0.83 55.46 7.04 4.24 0.88 0 0
17.11 0 0 0 1.11 3.65 0 1.34 58.46 0 14.13 1.12 0 0
49.01 2.71 0 1.13 0 1.45 0 0.86 32.92 7.95 0.35 0 0 0
29.15 0 0 1.21 0 1.85 0 0.79 41.25 15.45 2.54 0 0 0
25.42 0 0 1.31 0 2.18 0 1.16 45.1 17.3 0 0 0 0
30.39 0 0.34 3.49 0.79 3.52 0.86 3.13 39.35 7.66 8.99 0.24 0 0];
h=[97.61 99.89 100 98.88 96.06 96.51 98.92 98.84 99.7 99.82 98.24 99.77 99.81 95.39 99.57 98.14 99 98.41 97.25 98.76 99.17 98.52 100 95.5 98.88 97.06 99.82 99.89 98.81 90.17 88.41 96.5 98.69 98.43 98.66 99.96 97.52 96.21 97.63 99.98 98.57 98.76 98.57 93.71 99.88 97.95 94.68 96.67 98.7 98.01 97.9 99.12 98.34 98.63 97.26 98.24 96.94 95.33 91.7 94.08 98.25 99.67 96.92 96.38 92.24 92.47 98.76]';
gui=zeros();
for i=1:67
for j=1:14
gui(i,j)=z(i,j)./h(i);
end
end
clear
clc
yl1=[0.028691099 0.007329843 0.357905759 0
0.003659754 0.012531884 0.487967173 0];
yl2=[0.018620483 0.002602863 0.474822305 0];
yl3=[0.012927524 0.004580619 0.234324104 0
0 0.007220961 0.315762327 0];
yl4=[0 0.030984456 0.175958549 0];
yl5=[0.017079516 0.054066282 0.10519172 0
0.004154423 0.003343804 0.173675144 0.00233053
0.008109732 0.007408891 0.123247897 0
0 0.027242118 0.223242526 0
0 0.027783453 0.205515832 0
0.007768362 0.004338176 0.137308313 0
0 0.005389465 0.162599146 0
0 0.007806955 0.2560073 0
0 0.006683117 0.261155665 0
0 0.005496183 0.139033079 0];
yl6=[0.021643696 0 0.400408372 0.004083716
0.027760892 0 0.382370821 0.004457953];
yl7=[0 0.008564614 0.447202949 0
0 0.012544609 0.487725749 0];
yl8=[0 0.051682566 0.266170458 0
0.008027038 0.056506126 0.632129278 0
0.014378815 0.015620151 0.462915072 0
0.012482954 0.014371132 0.422112661 0.004930242
0.004580153 0.008178844 0.559869138 0
0.002444728 0.007440476 0.504039116 0
0 0.008327481 0.55643624 0
0 0.013825836 0.603177879 0];
yl9=[0.019101483 0.002027532 0.470814214 0];
yl10=[0.004819524 0.015484003 0.477337982 0
0.003277681 0.006965072 0.426200963 0
0.002942072 0.007405904 0.500253627 0
0 0.00891049 0.617962738 0];
yl11=[0 0.104287718 0.287317171 0
0 0.031962541 0.330313518 0
0 0.105890603 0.295832498 0
0 0.036039644 0.299529482 0];
yl12=[0 0.008923013 0.34156464 0];
yl13=[0.015969503 0.011539254 0.328662683 0];
yl14=[0.01013582 0.00111494 0.200283803 0
0.001766968 0.001663029 0.229186155 0];
yl15=[0 0.004701881 0.161664666 0
0 0.030106021 0.172434487 0];
yl16=[0 0.085558252 0.294700647 0
0.046632124 0.004470182 0.168139795 0];
yl17=[0 0.0078 0.0025 0
0.02184466 0.051476537 0.014259709 0
0.02144493 0.022694149 0 0
0.027147446 0.033882499 0 0
0.024160938 0.02537404 0.002021836 0
0.061108863 0.022055848 0.003541076 0
0.024056029 0.033292732 0.010150223 0];
yl18=[0 0 0 0.024267352];
yl19=[0.001705115 0.032497492 0 0
0.003207377 0.015535732 0 0
0.002604949 0.00841599 0 0
0.002912524 0.016571256 0 0
0.0035 0.0055 0 0
0.002024087 0.015585467 0 0];
yl20=[0.017826042 0.039647577 0 0];
yl21=[0.029345832 0.048196454 0 0
0.004267859 0.010872879 0.001117773 0];
yl22=[0.005050505 0.004747475 0.016363636 0];
fl=zeros();
for j=1:4
su=0;[m,n]=size(yl1);
for i=1:m
su=su+yl1(i,j);
end
fl(1,j)=su./m;
end
for j=1:4
su=0;[m,n]=size(yl2);
for i=1:m
su=su+yl2(i,j);
end
fl(2,j)=su./m;
end
for j=1:4
su=0;[m,n]=size(yl3);
for i=1:m
su=su+yl3(i,j);
end
fl(3,j)=su./m;
end
for j=1:4
su=0;[m,n]=size(yl4);
for i=1:m
su=su+yl4(i,j);
end
fl(4,j)=su./m;
end
for j=1:4
su=0;[m,n]=size(yl5);
for i=1:m
su=su+yl5(i,j);
end
fl(5,j)=su./m;
end
for j=1:4
su=0;[m,n]=size(yl6);
for i=1:m
su=su+yl6(i,j);
end
fl(6,j)=su./m;
end
for j=1:4
su=0;[m,n]=size(yl7);
for i=1:m
su=su+yl7(i,j);
end
fl(7,j)=su./m;
end
for j=1:4
su=0;[m,n]=size(yl8);
for i=1:m
su=su+yl8(i,j);
end
fl(8,j)=su./m;
end
for j=1:4
su=0;[m,n]=size(yl9);
for i=1:m
su=su+yl9(i,j);
end
fl(9,j)=su./m;
end
for j=1:4
su=0;[m,n]=size(yl10);
for i=1:m
su=su+yl10(i,j);
end
fl(10,j)=su./m;
end
for j=1:4
su=0;[m,n]=size(yl11);
for i=1:m
su=su+yl11(i,j);
end
fl(11,j)=su./m;
end
for j=1:4
su=0;[m,n]=size(yl12);
for i=1:m
su=su+yl12(i,j);
end
fl(12,j)=su./m;
end
for j=1:4
su=0;[m,n]=size(yl13);
for i=1:m
su=su+yl13(i,j);
end
fl(13,j)=su./m;
end
for j=1:4
su=0;[m,n]=size(yl14);
for i=1:m
su=su+yl14(i,j);
end
fl(14,j)=su./m;
end
for j=1:4
su=0;[m,n]=size(yl15);
for i=1:m
su=su+yl15(i,j);
end
fl(15,j)=su./m;
end
for j=1:4
su=0;[m,n]=size(yl16);
for i=1:m
su=su+yl16(i,j);
end
fl(16,j)=su./m;
end
for j=1:4
su=0;[m,n]=size(yl17);
for i=1:m
su=su+yl17(i,j);
end
fl(17,j)=su./m;
end
for j=1:4
su=0;[m,n]=size(yl18);
for i=1:m
su=su+yl18(i,j);
end
fl(18,j)=su./m;
end
for j=1:4
su=0;[m,n]=size(yl19);
for i=1:m
su=su+yl19(i,j);
end
fl(19,j)=su./m;
end
for j=1:4
su=0;[m,n]=size(yl20);
for i=1:m
su=su+yl20(i,j);
end
fl(20,j)=su./m;
end
for j=1:4
su=0;[m,n]=size(yl21);
for i=1:m
su=su+yl21(i,j);
end
fl(21,j)=su./m;
end
for j=1:4
su=0;[m,n]=size(yl22);
for i=1:m
su=su+yl22(i,j);
end
fl(22,j)=su./m;
end
%问题四的matlab代码
%初始数据归一化处理
clear
clc
z=[69.33 0 9.99 6.32 0.87 3.93 1.74 3.87 0 0 1.17 0 0 0.39
36.28 0 1.05 2.34 1.18 5.73 1.86 0.26 47.43 0 3.57 0.19 0 0
87.05 0 5.19 2.01 0 4.06 0 0.78 0.25 0 0.66 0 0 0
61.71 0 12.37 5.87 1.11 5.5 2.16 5.09 1.41 2.86 0.7 0.1 0 0
65.88 0 9.67 7.12 1.56 6.44 2.06 2.18 0 0 0.79 0 0 0.36
61.58 0 10.95 7.35 1.77 7.5 2.62 3.27 0 0 0.94 0.06 0 0.47
67.65 0 7.37 0 1.98 11.15 2.39 2.51 0.2 1.38 4.18 0.11 0 0
59.81 0 7.68 5.41 1.73 10.05 6.04 2.18 0.35 0.97 4.5 0.12 0 0
92.63 0 0 1.07 0 1.98 0.17 3.24 0 0 0.61 0 0 0
20.14 0 0 1.48 0 1.34 0 10.41 28.68 31.23 3.59 0.37 0 2.58
4.61 0 0 3.19 0 1.11 0 3.14 32.45 30.62 7.56 0.53 0 15.03
95.02 0 0.59 0.62 0 1.32 0.32 1.55 0 0 0.35 0 0 0
96.77 0 0.92 0.21 0 0.81 0.26 0.84 0 0 0 0 0 0
33.59 0 0.21 3.51 0.71 2.69 0 4.93 25.39 14.61 9.38 0.37 0 0
94.29 0 1.01 0.72 0 1.46 0.29 1.65 0 0 0.15 0 0 0
59.01 2.86 12.53 8.7 0 6.16 2.88 4.73 0 0 1.27 0 0 0
62.47 3.38 12.28 8.23 0.66 9.23 0.5 0.47 1.62 0 0.16 0 0 0
65.18 2.1 14.52 8.27 0.52 6.18 0.42 1.07 0.11 0 0 0.04 0 0
79.46 0 9.42 0 1.53 3.05 0 0 0 0 1.36 0.07 2.36 0
29.64 0 0 2.93 0.59 3.57 1.33 3.51 42.82 5.35 8.83 0.19 0 0
53.33 0.8 0.32 2.82 1.54 13.65 1.03 0 15.71 7.31 1.1 0.25 1.31 0
76.68 0 0 4.71 1.22 6.19 2.37 3.28 1 1.97 1.1 0 0 0
92.35 0 0.74 1.66 0.64 3.5 0.35 0.55 0 0 0.21 0 0 0
28.79 0 0 4.58 1.47 5.38 2.74 0.7 34.18 6.1 11.1 0.46 0 0
31.94 0 0 0.47 0 1.59 0 8.46 29.14 26.23 0.14 0.91 0 0
50.61 2.31 0 0.63 0 1.9 1.55 1.12 31.9 6.65 0.19 0.2 0 0
19.79 0 0 1.44 0 0.7 0 10.57 29.53 32.25 3.13 0.45 0 1.96
3.72 0 0.4 3.01 0 1.18 0 3.6 29.92 35.45 6.04 0.62 0 15.95
92.72 0 0 0.94 0.54 2.51 0.2 1.54 0 0 0.36 0 0 0
17.98 0 0 3.19 0.47 1.87 0.33 1.13 44 14.2 6.34 0.66 0 0
37.36 0 0.71 0 0 5.45 1.51 4.78 9.3 23.55 5.75 0 0 0
53.79 7.92 0 0.5 0.71 1.42 0 2.99 16.98 11.86 0 0.33 0 0
68.08 0 0.26 1.34 1 4.7 0.41 0.33 17.14 4.04 1.04 0.12 0.23 0
65.91 0 0 1.6 0.89 3.11 4.59 0.44 16.55 3.42 1.62 0.3 0 0
69.71 0 0.21 0.46 0 2.36 1 0.11 19.76 4.88 0.17 0 0 0
75.51 0 0.15 0.64 1 2.35 0 0.47 16.16 3.55 0.13 0 0 0
35.78 0 0.25 0.78 0 1.62 0.47 1.51 46.55 10 0.34 0.22 0 0
65.91 0 0 0.38 0 1.44 0.17 0.16 22.05 5.68 0.42 0 0 0
39.57 2.22 0.14 0.37 0 1.6 0.32 0.68 41.61 10.83 0.07 0.22 0 0
60.12 0 0.23 0.89 0 2.72 0 3.01 17.24 10.34 1.46 0.31 0 3.66
32.93 1.38 0 0.68 0 2.57 0.29 0.73 49.31 9.79 0.48 0.41 0 0
26.25 0 0 1.11 0 0.5 0 0.88 61.03 7.22 1.16 0.61 0 0
16.71 0 0 1.87 0 0.45 0.19 0 70.21 6.69 1.77 0.68 0 0
18.46 0 0.44 4.96 2.73 3.33 1.79 0.19 44.12 9.76 7.46 0.47 0 0
63.3 0.92 0.3 2.98 1.49 14.34 0.81 0.74 12.31 2.03 0.41 0.25 0 0
34.34 0 1.41 4.49 0.98 4.35 2.12 0 39.22 10.29 0 0.35 0.4 0
12.41 0 0 5.24 0.89 2.25 0.76 5.35 59.85 7.29 0 0.64 0 0
21.7 0 0 6.4 0.95 3.41 1.39 1.51 44.75 3.26 12.83 0.47 0 0
36.93 0 0 4.24 0.51 3.86 2.74 0 37.74 10.35 1.41 0.48 0.44 0
51.26 5.74 0.15 0.79 1.09 3.53 0 2.67 21.88 10.47 0.08 0.35 0 0
51.33 5.68 0.35 0 1.16 5.66 0 2.72 20.12 10.88 0 0 0 0
60.74 3.06 0.2 2.14 0 12.69 0.77 0.43 13.61 5.22 0 0.26 0 0
61.28 2.66 0.11 0.84 0.74 5 0 0.53 15.99 10.96 0 0.23 0 0
55.21 0 0.25 0 1.67 4.79 0 0.77 25.25 10.06 0.2 0.43 0 0
51.54 4.66 0.29 0.87 0.61 3.06 0 0.65 25.4 9.23 0.1 0.85 0 0
54.61 0 0.3 2.08 1.2 6.5 1.27 0.45 23.02 4.19 4.32 0.3 0 0
45.02 0 0 3.12 0.54 4.16 0 0.7 30.61 6.22 6.34 0.23 0 0
24.61 0 0 3.58 1.19 5.25 1.19 1.37 40.24 8.94 8.1 0.39 0.47 0
21.35 0 0 5.13 1.45 2.51 0.42 0.75 51.34 0 8.75 0 0 0
25.74 1.22 0 2.27 0.55 1.16 0.23 0.7 47.42 8.64 5.71 0.44 0 0
63.66 3.04 0.11 0.78 1.14 6.06 0 0.54 13.66 8.99 0 0.27 0 0
22.28 0 0.32 3.19 1.28 4.15 0 0.83 55.46 7.04 4.24 0.88 0 0
17.11 0 0 0 1.11 3.65 0 1.34 58.46 0 14.13 1.12 0 0
49.01 2.71 0 1.13 0 1.45 0 0.86 32.92 7.95 0.35 0 0 0
29.15 0 0 1.21 0 1.85 0 0.79 41.25 15.45 2.54 0 0 0
25.42 0 0 1.31 0 2.18 0 1.16 45.1 17.3 0 0 0 0
30.39 0 0.34 3.49 0.79 3.52 0.86 3.13 39.35 7.66 8.99 0.24 0 0];
h=[97.61 99.89 100 98.88 96.06 96.51 98.92 98.84 99.7 99.82 98.24 99.77 99.81 95.39 99.57 98.14 99 98.41 97.25 98.76 99.17 98.52 100 95.5 98.88 97.06 99.82 99.89 98.81 90.17 88.41 96.5 98.69 98.43 98.66 99.96 97.52 96.21 97.63 99.98 98.57 98.76 98.57 93.71 99.88 97.95 94.68 96.67 98.7 98.01 97.9 99.12 98.34 98.63 97.26 98.24 96.94 95.33 91.7 94.08 98.25 99.67 96.92 96.38 92.24 92.47 98.76]';
gui=zeros();
for i=1:67
for j=1:14
gui(i,j)=z(i,j)./h(i);
end
end
clear
clc
lb1=[0.018620483 0.002602863 0.474822305 0
0.028691099 0.007329843 0.357905759 0
0.003659754 0.012531884 0.487967173 0];
lb2=[0.017079516 0.054066282 0.10519172 0
0 0.030984456 0.175958549 0
0.004154423 0.003343804 0.173675144 0.00233053
0.008109732 0.007408891 0.123247897 0
0.021643696 0 0.400408372 0.004083716
0.027760892 0 0.382370821 0.004457953
0 0.027242118 0.223242526 0
0 0.027783453 0.205515832 0
0.007768362 0.004338176 0.137308313 0
0 0.005389465 0.162599146 0
0 0.007806955 0.2560073 0
0 0.006683117 0.261155665 0
0.012927524 0.004580619 0.234324104 0
0 0.007220961 0.315762327 0
0 0.005496183 0.139033079 0];
lb3=[0 0.104287718 0.287317171 0
0 0.031962541 0.330313518 0
0 0.051682566 0.266170458 0
0 0.105890603 0.295832498 0
0 0.036039644 0.299529482 0
0.004819524 0.015484003 0.477337982 0
0.003277681 0.006965072 0.426200963 0
0.002942072 0.007405904 0.500253627 0
0 0.00891049 0.617962738 0
0.019101483 0.002027532 0.470814214 0
0.008027038 0.056506126 0.632129278 0
0.014378815 0.015620151 0.462915072 0
0.012482954 0.014371132 0.422112661 0.004930242
0.004580153 0.008178844 0.559869138 0
0.002444728 0.007440476 0.504039116 0
0 0.008327481 0.55643624 0
0 0.013825836 0.603177879 0
0 0.008564614 0.447202949 0
0 0.012544609 0.487725749 0];
lb4=[0 0.085558252 0.294700647 0
0.015969503 0.011539254 0.328662683 0
0.046632124 0.004470182 0.168139795 0
0.01013582 0.00111494 0.200283803 0
0 0.004701881 0.161664666 0
0.001766968 0.001663029 0.229186155 0
0 0.030106021 0.172434487 0
0 0.008923013 0.34156464 0];
lb5=[0 0.0078 0.0025 0
0.02184466 0.051476537 0.014259709 0
0.02144493 0.022694149 0 0
0.027147446 0.033882499 0 0
0.024160938 0.02537404 0.002021836 0
0.061108863 0.022055848 0.003541076 0
0 0 0 0.024267352
0.024056029 0.033292732 0.010150223 0];
lb6=[0.001705115 0.032497492 0 0
0.003207377 0.015535732 0 0
0.002604949 0.00841599 0 0
0.002912524 0.016571256 0 0
0.0035 0.0055 0 0
0.002024087 0.015585467 0 0];
lb7=[0.017826042 0.039647577 0 0
0.029345832 0.048196454 0 0
0.005050505 0.004747475 0.016363636 0
0.004267859 0.010872879 0.001117773 0];
glb=zeros();
for j=1:4
[m,n]=size(lb1);su=0;
for i=1:m
su=su+lb1(i,j);
end
glb(1,j)=su./m;
end
for j=1:4
[m,n]=size(lb2);su=0;
for i=1:m
su=su+lb2(i,j);
end
glb(2,j)=su./m;
end
for j=1:4
[m,n]=size(lb3);su=0;
for i=1:m
su=su+lb3(i,j);
end
glb(3,j)=su./m;
end
for j=1:4
[m,n]=size(lb4);su=0;
for i=1:m
su=su+lb4(i,j);
end
glb(4,j)=su./m;
end
for j=1:4
[m,n]=size(lb5);su=0;
for i=1:m
su=su+lb5(i,j);
end
glb(5,j)=su./m;
end
for j=1:4
[m,n]=size(lb6);su=0;
for i=1:m
su=su+lb6(i,j);
end
glb(6,j)=su./m;
end
for j=1:4
[m,n]=size(lb7);su=0;
for i=1:m
su=su+lb7(i,j);
end
glb(7,j)=su./m;
end
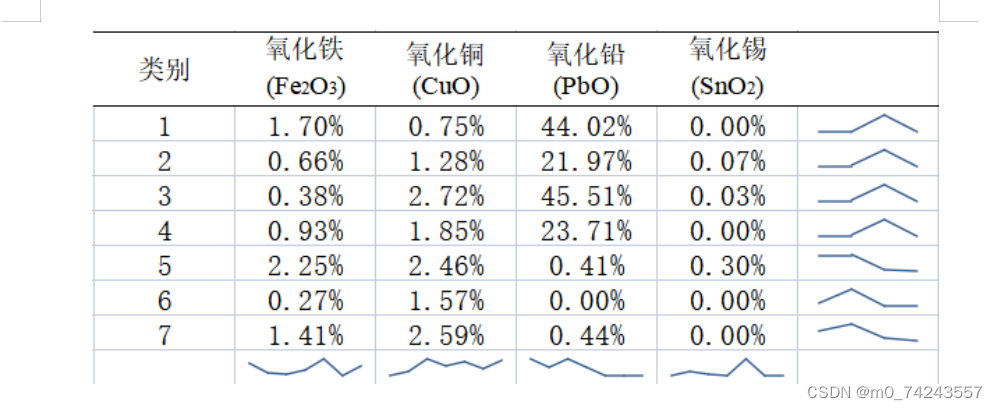 图 5关联关系折线图
图 5关联关系折线图