rWCVP:按照物种的原生分布区清洗坐标点
加载库
library(rWCVP)
library(rgbif)
library(tidyverse)
library(sf)
工作流(单个物种)
本文中,我们使用Callitris rhomboidea作为示例物种
1. 下载发现记录数据(rgbif)
第一步是从某处获取物种记录。我们将使用 rgbif 从 GBIF 获取这些。
我们在这里将物种记录限制为 1000 次,因此下载时间不会太长。我们要求只获取坐标没有被GBIF自动标记任何问题的记录。
gbif_response = occ_data(scientificName = "Callitris rhomboidea",
limit = 1000,
hasCoordinate = TRUE,
hasGeospatialIssue = FALSE)
paste(1000, "of", gbif_response$meta$count, "records downloaded.")
[1] "1000 of 3580 records downloaded."
2. 发现记录的预备
当我们从下载数据中移除非原产地发现记录前,我们需要用正确的格式获取数据。
如果发现记录非常庞大,而且某物种在非常多的地方都是本地种,那么移除非本地物种记录时以像非常耗时的计算任务。所以可以分多次进行数据清洗,比如移除海洋中的记录或在研究所中的记录。
就本文而言,我们的数据量并不大,所以并不需要预先进行清洗数据。但我们确实需要将我们的记录数据集转换为空间数据集,以便通过图层过滤分布点。
occs = gbif_response$data %>%
select(scientificName, decimalLatitude, decimalLongitude) %>%
st_as_sf(coords=c("decimalLongitude", "decimalLatitude"), crs=st_crs(4326))
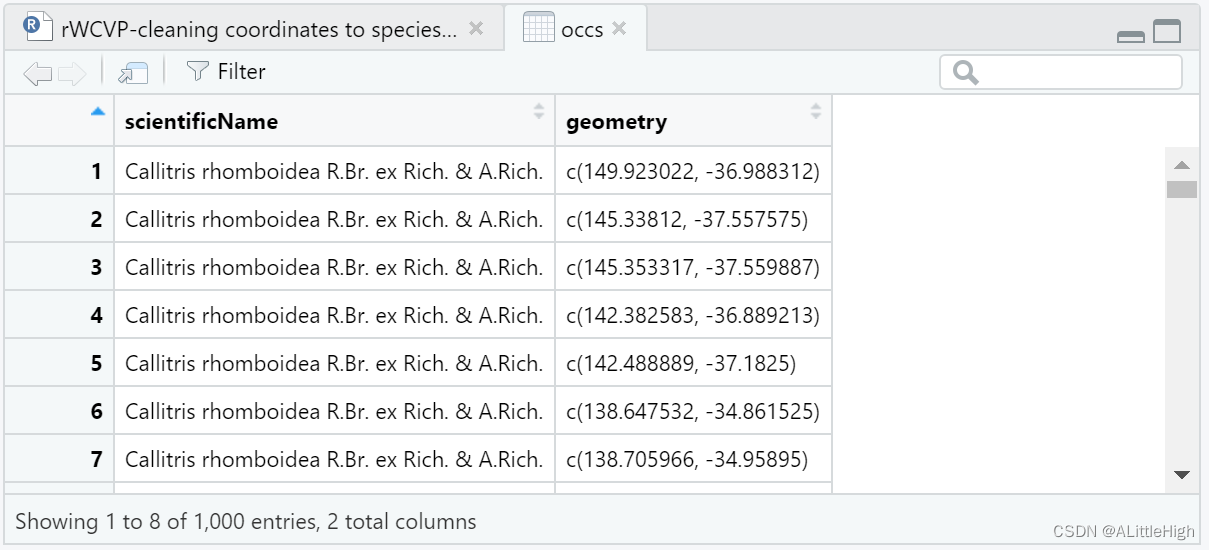
如果您使用自己的记录数据集,而不是从 GBIF 下载的数据集,您仍然需要像我们上面所做的那样将其转换为空间数据集并设置适当的坐标系。我们在这里使用了 EPSG 4326,因为它是经度和纬度坐标的默认值。
3. 获取原生区范围
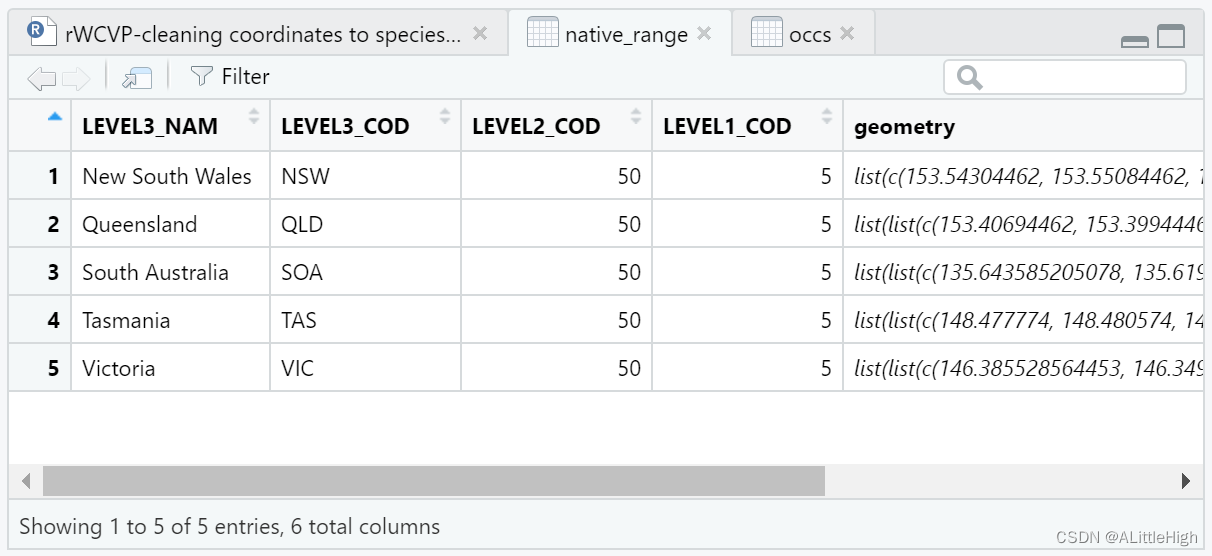
现在我们可以用rWCVP下载物种的原生分布区范围。

native_range = wcvp_distribution("Callitris rhomboidea",
taxon_rank = "species",
introduced = FALSE,
extinct = FALSE,
location_doubtful = FALSE)
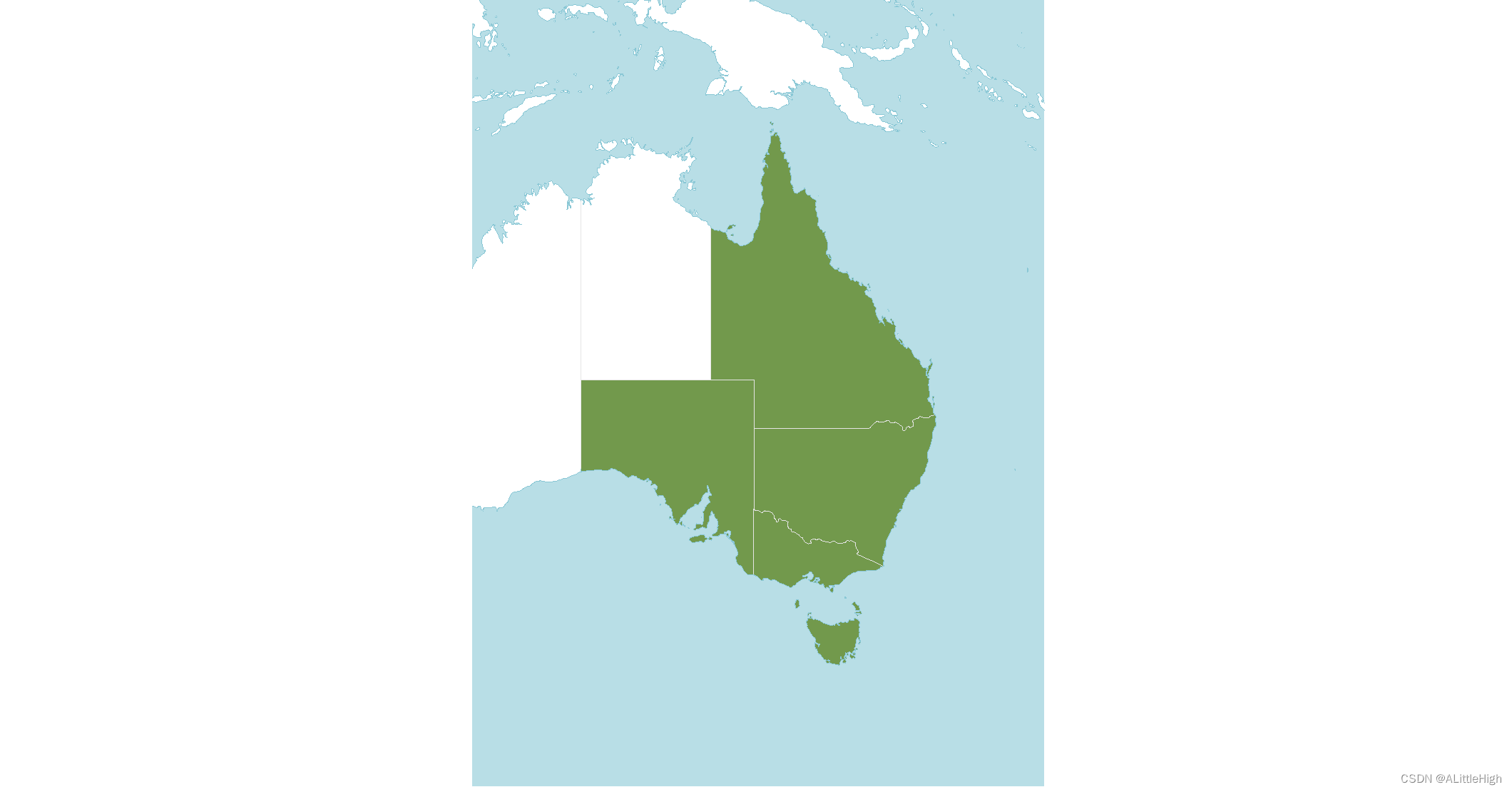
可视化观察一下分布区
(p = wcvp_distribution_map(native_range, crop_map = TRUE) +
theme(legend.position = "none"))
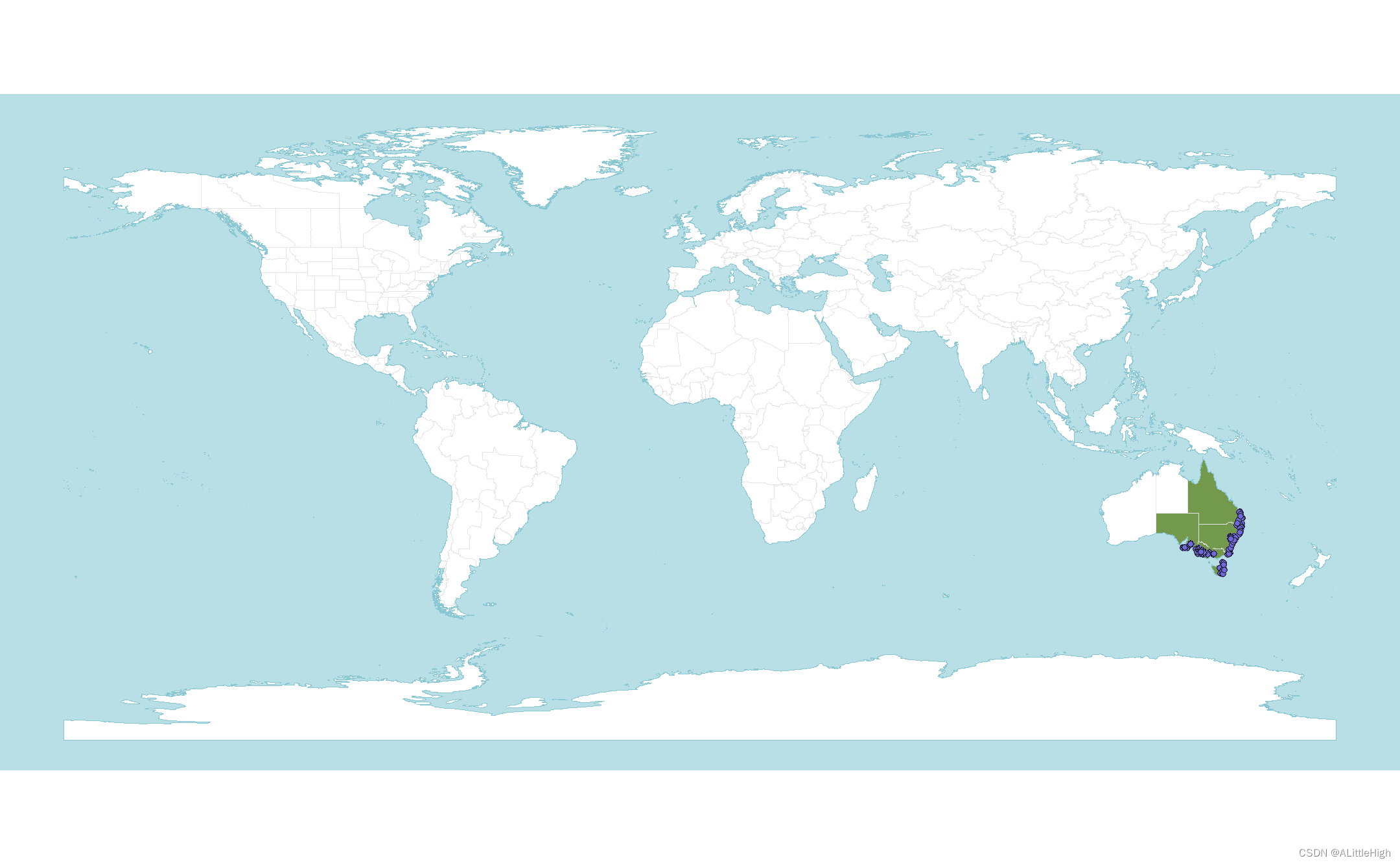
那么记录分布点在地图上的分布如何呢?
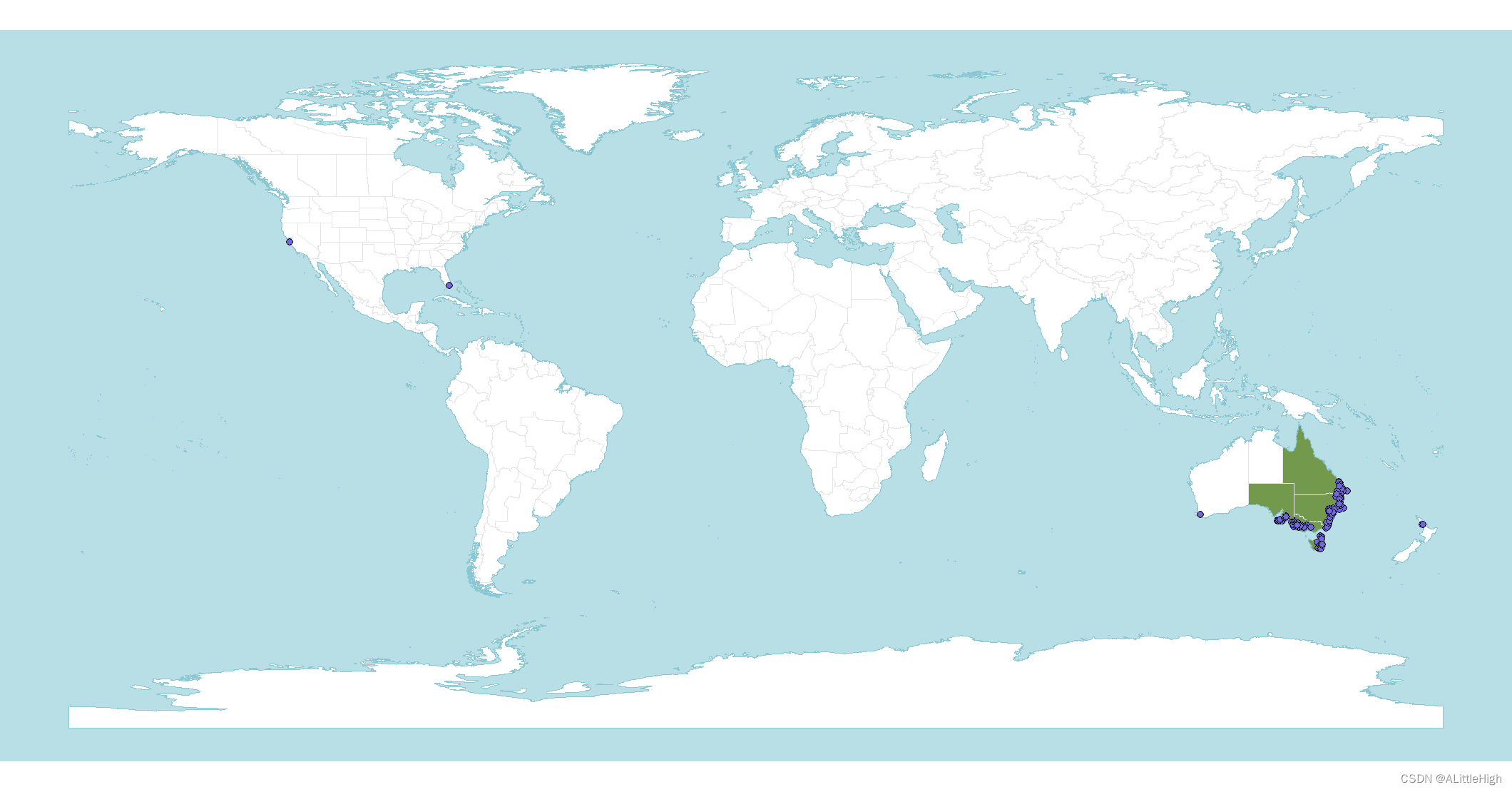
4. 清除非原生分布记录
现在, 清除掉哪些冗余的分布点
一种方法是检查哪些点落在原生范围多边形内,为每个点获取 TRUE/FALSE 值。 可以使用sf的sf_intersects完成。 因为我们获得的分布区不止一个,那么每个分布点就会获得不止一个值,所以需要使用st_union将原生区合并起来。
occs$native = st_intersects(occs, st_union(native_range), sparse = FALSE)[,1]
在地图上将有问题的记录点标注出来
p + geom_sf(data = occs,
fill=c("red", "#72994c")[factor(occs$native)],
col="black",
shape=21)
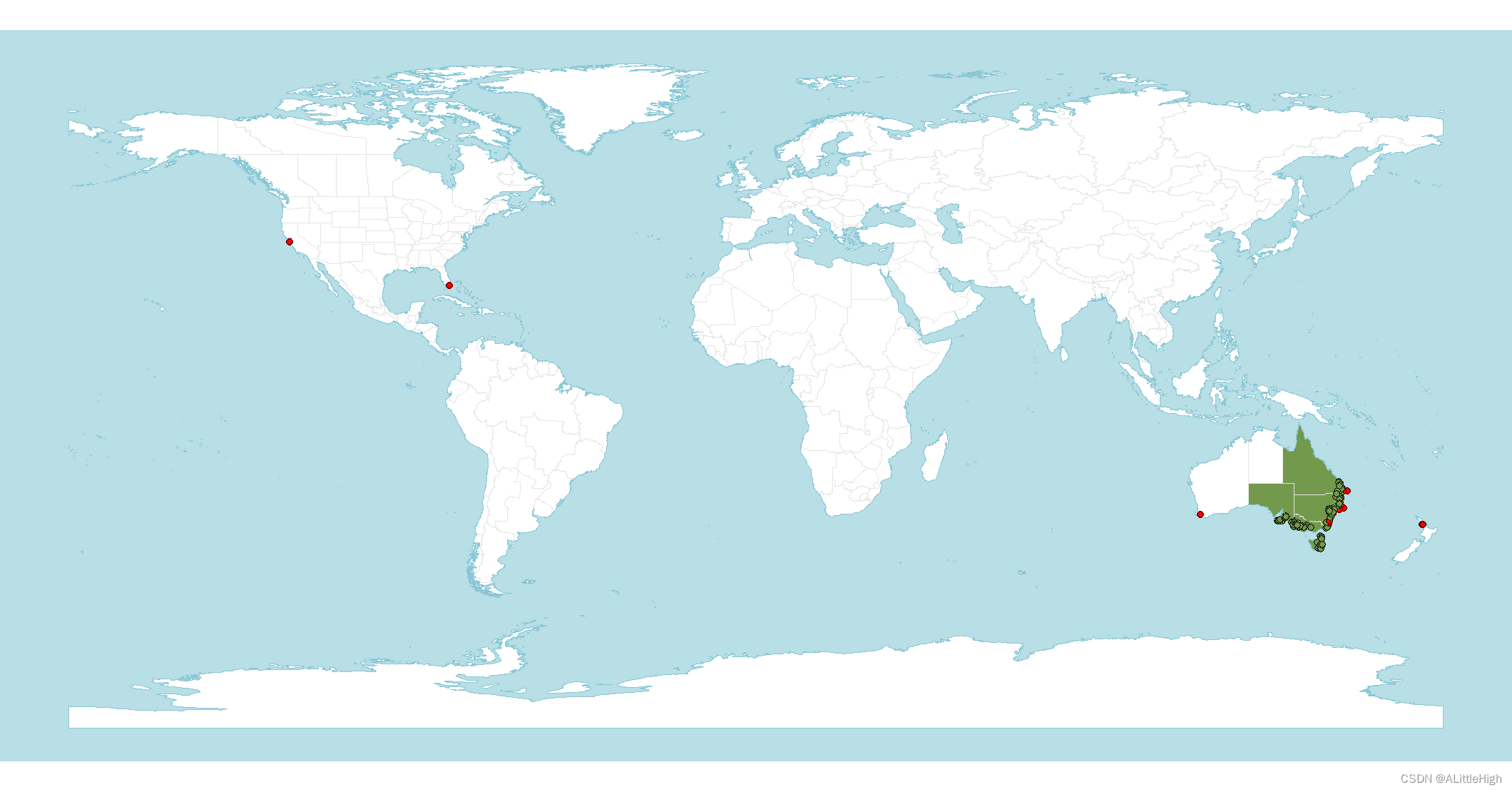
可以看到,有一些明显不在原生区内,而少数记录与原生区很近。让我们靠近些看看原生区范围周围的记录点:
lims = st_bbox(native_range)
p + geom_sf(data=occs,
fill=c("red","#72994c")[factor(occs$native)],
col="black",
shape=21) +
coord_sf(xlim = lims[c(1,3)], ylim = lims[c(2,4)])
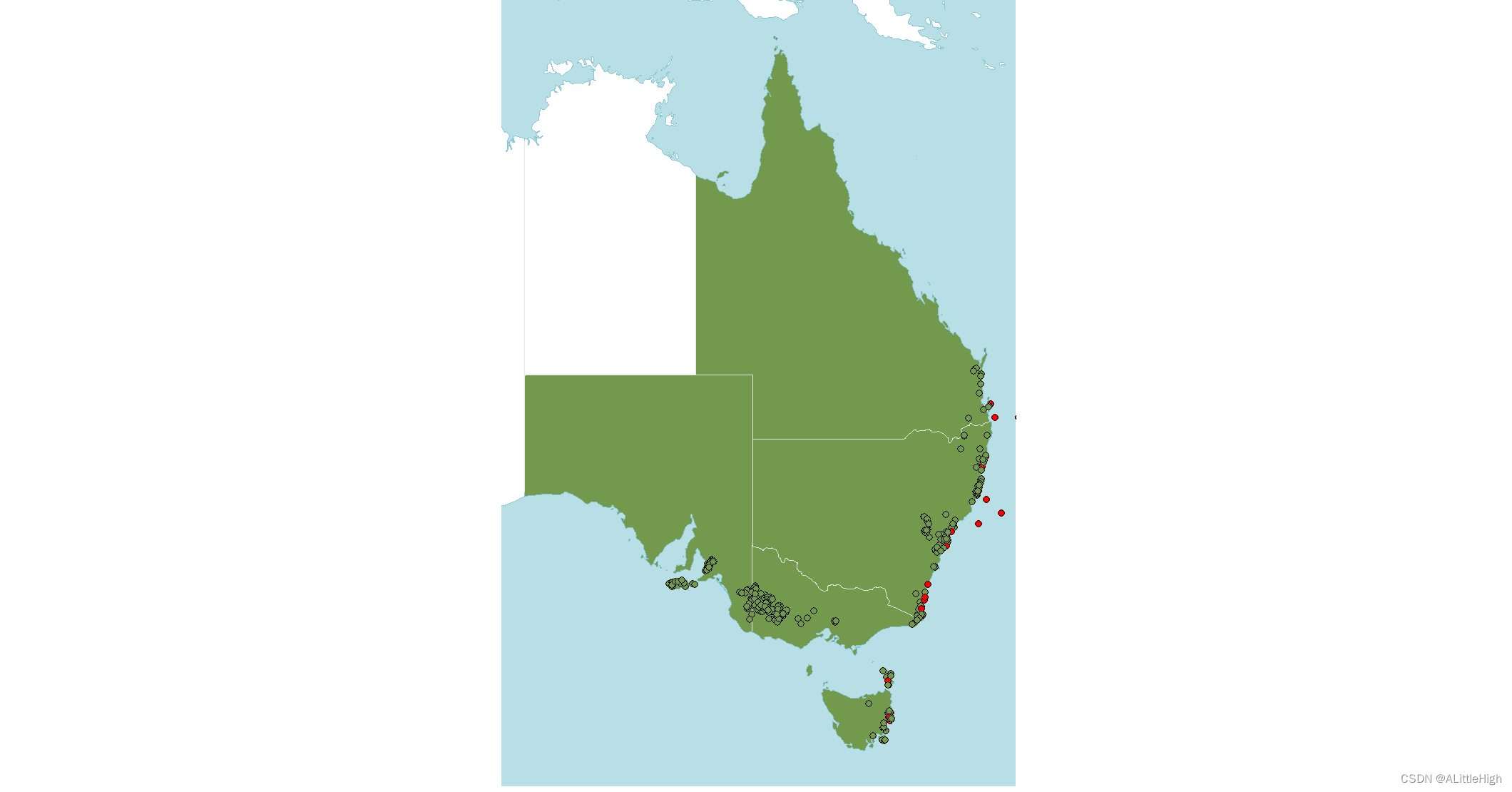
可以清楚地看到,这些点落在了海里。但是有一些点距离海岸线非常近,可能是WGSRPD图层文件的分辨率导致的。
或许我们可以添加一些条件,还让数据清洗地不那么严格。
图层文件的坐标系是基于度的,所以1km大约为0.009度。但随着纬度升高,0.009度会比1km越来越长,这就要求根据数据和分布区位置做相应的调整。
让我们观察一下有多少分布记录落在原生区外1km内。
buffered_dist = native_range %>%
st_union() %>%
st_buffer(0.009)
occs$native_buffer = st_intersects(occs, buffered_dist, sparse=FALSE)[,1]
suspect_occs = occs %>% filter(! native)
p + geom_sf(data = buffered_dist,
fill = "transparent",
col = "gold") +
geom_sf(data=suspect_occs,
fill=c("red", "gold")[factor(suspect_occs$native_buffer)],
col="black",
shape=21) +
coord_sf(xlim = lims[c(1,3)], ylim = lims[c(2,4)])
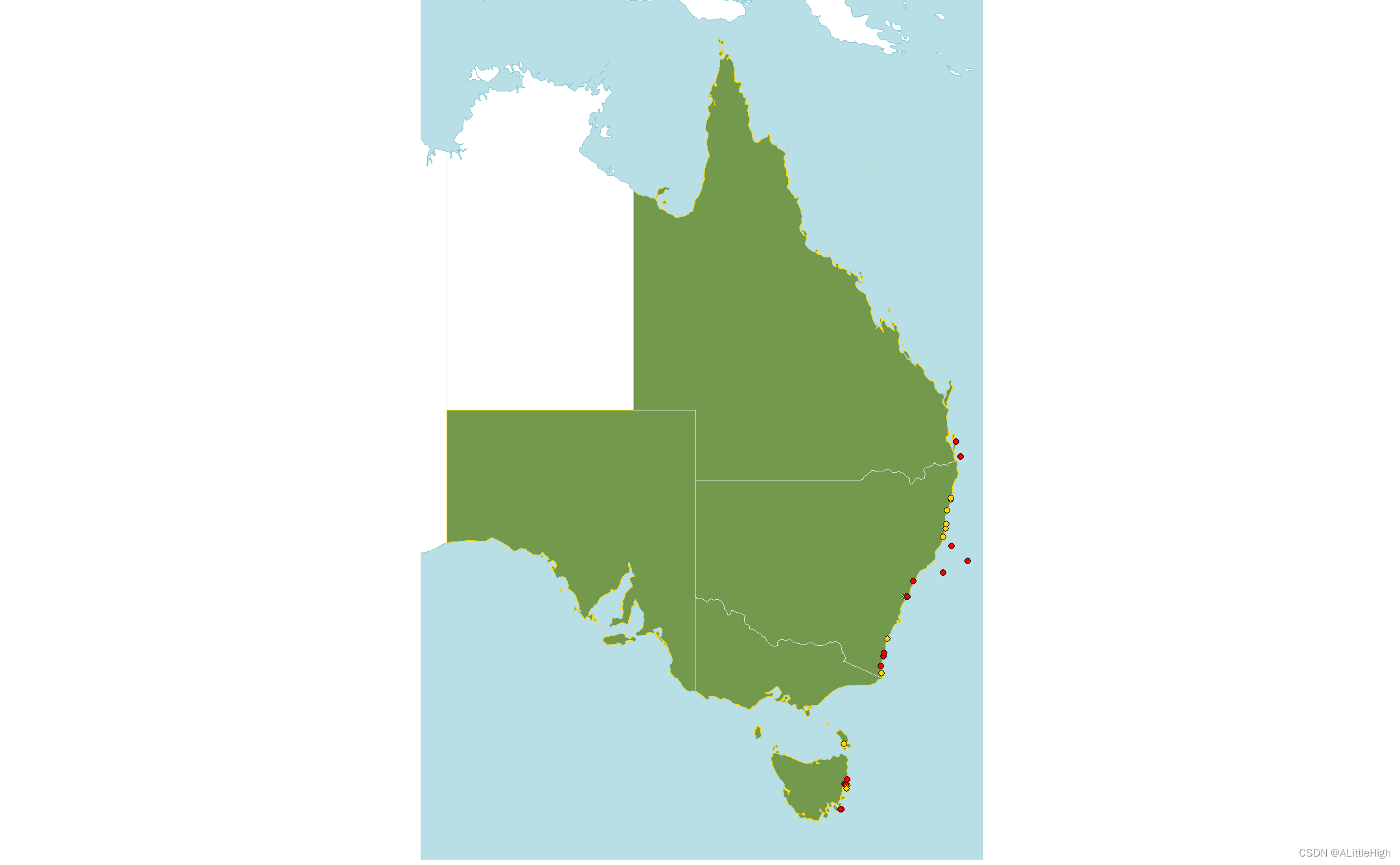
现在,我们可以丢弃在原生范围之外 >1 公里(大约)的记录。
occs_filtered = occs %>% filter(native_buffer)
p + geom_sf(dat=occs_filtered, fill="#6e6ad9", col="black", shape=21)