NC2022: Federated learning enables big data for rare cancer boundary detection
尽管机器学习(ML)在各个学科领域都显示出了潜力,但样本外泛化仍然令人担忧。目前通过共享多个站点的数据来解决这个问题,但由于各种限制,这样的集中处理很难扩展。联邦机器学习(FL)提供了一种替代范例,可以通过仅共享数值模型更新来实现准确和可泛化的ML。在这里,我们展示了迄今为止最大的FL研究,涉及来自6个大洲的71个站点的数据,为罕见疾病胶质母细胞瘤生成自动肿瘤边界检测器,并在文献中报告了最大的数据集(n = 6,314)。我们证明了针对可手术的肿瘤,我们的方法使划分边界的改进达到了33%,对于完整的肿瘤范围,改进达到了23%,超过了公开训练模型。
我们预计我们的研究将:
-
1)使更多的医疗保健研究受到大量多样化数据的启发,确保罕见疾病和代表性不足的人群得到有意义的结果;
-
2)通过发布我们的共识模型,促进对胶质母细胞瘤的进一步分析;
-
3)展示FL在这样的规模和任务复杂性下的有效性,作为多站点协作的范例转变,减轻了数据共享的需求。
-
mpMRI: 多参数磁共振成像(mpMRI)扫描
整个联邦学习过程采用分阶段的方法:
- 从“公共初始模型”(使用来自16个站点的231个病例的数据进行训练)开始
- 接着是“初步共识模型”(使用来自35个站点的2471个病例的数据进行训练)
- 最后形成“最终共识模型”(使用来自71个站点的6314个病例的数据进行训练)。
为了定量评估训练模型的性能,每个参与站点贡献的总案例数的20%被排除在模型训练过程之外,用作“本地验证数据”。为了进一步评估模型在未见数据中的泛化能力,有6个站点没有参与任何训练阶段,代表了一个未见的“样本外”数据人群,共590个案例。为了方便进一步评估而不给合作站点带来负担,这些案例的子集(n = 332)被聚合成一个“集中的样本外”数据集。训练是从预训练模型(即我们的公共初始模型)开始的,而不是从随机初始化点开始,以便更快地收敛模型性能。在这里,模型性能是使用Dice相似系数(DSC)进行定量评估的,该系数评估模型的预测与参考标准在三种肿瘤亚区(ET,TC,WT)中的空间一致性。
-
Dice相似系数:一种用于比较两个样本相似程度的度量方法,通常用于医学图像分割中。它定义为两个样本的交集的大小除以它们的并集的大小的两倍。在这个文本中,Dice相似系数被用来评估模型的性能,衡量模型的预测结果与参考标准在三个肿瘤亚区(ET、TC、WT)方面的
空间一致性。
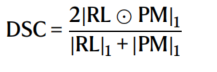
-
Wilcoxon signed-rank test是一种非参数假设检验方法,用于比较两个相关样本的中位数是否有差异。它适用于数据不服从正态分布的情况,可以用于小样本和大样本。该检验的原假设是两个相关总体的中位数相等,备择假设是两个相关总体的中位数不相等。它的基本思想是将两个样本的差值按大小排序,然后比较正负号是否相同,以此得到一个统计量。如果统计量的P值小于显著性水平,则拒绝原假设,认为两个样本的中位数有显著差异
-
我们利用了一般细致的深度学习框架(GaNDLF)的数据加载和处理管道,以便尝试各种数据增强技术。在数据加载后,我们从图像中删除了所有零轴、冠状面和矢状面,并对非零图像强度进行了Z-score标准化。
-
模型结构:3D-ResUNet,The network had 30 base filters, with a learning rate of
lr = 5 × 10−5 optimized using the Adam optimizer102. For the loss function used in training, we used the generalized DSC score -
No penalties were used in the loss function, due to our use of ‘mirrored’ DSC loss
本文介绍了肝脏肿瘤分割基准测试(LiTS)的设置和结果,该测试是与2017年IEEE国际生物医学成像研讨会(ISBI)和2017年和2018年国际医学图像计算和计算机辅助干预会议(MICCAI)一起组织的。该图像数据集包含原发和继发肿瘤,大小和外观各异,并具有各种病变与背景的水平(高密度/低密度),是与七家医院和研究机构合作创建的。共有75个提交的肝脏和肝肿瘤分割算法在131个计算机断层扫描(CT)体积上进行了训练,并在来自不同患者的70个未见过的测试图像上进行了测试。我们发现,在三个事件中,没有一个算法在肝脏和肝肿瘤的分割中表现最佳。最佳肝脏分割算法的Dice得分为0.963,而对于肿瘤分割,最佳算法的Dice得分分别为0.674(ISBI 2017)、0.702(MICCAI 2017)。
肝脏分割的技术挑战:
全自动的肝脏及其病变的分割在许多方面仍然具有挑战性。
- 首先,病变与背景对比度的变化(Moghbel等,2017)可能由以下因素引起:
- (a)对比剂的变化,
- (b)由于不同的注射时间而引起的对比增强的变化,
- 以及(c)不同的采集参数(例如分辨率、mAs和kVp曝光、重建核)。
- 其次,不同类型的病变(良性和恶性以及肿瘤亚型)的共存,其图像外观的变化,对于自动化病变分割提出了额外的挑战。
- 第三,肝脏组织背景信号在慢性肝病存在的情况下可能会有很大的变化,这是肝癌的常见前兆。观察到许多算法难以处理疾病特异性的变异性,包括病变的大小、形状和数量的差异,以及由治疗引起的对肝脏器官本身形状和外观的修改(Moghbel等,2017)。
图1中展示了两个患者的肝脏和肿瘤外观的差异,说明了在具有不同病变的未知测试案例上推广的挑战。

key contributions to fully automated liver and liver tumor segmentation
- we generate a new public multi-center dataset of 201 abdominal CT Volumes and
the reference segmentations of liver and liver tumors - present the set-up and the summary of our LiTS benchmarks in three
grand challenges - Third, we review, evaluate, rank, and analyze there sulting state-of-the-art algorithms and results.
分割方法:
Published work on liver segmentation methods can be grouped into three categories based on:
(1) prior shape and geometric knowledge,
(2) intensity distribution and spatial context
(3) deep learning.
已发表的关于肝脏分割方法的研究可以根据以下三个类别进行分组:(1) 先验形状和几何知识,(2) 强度分布和空间上下文,以及(3) 深度学习。
基于强度分布和空间上下文的方法。
概率图谱(PA)是从训练数据集中学习的具有参数的解剖图谱。Park等人提出了第一个PA,利用32个腹部CT系列进行基于互信息和薄板样条作为变形转换的配准(Park等,2003),并使用马尔可夫随机场(MRF)进行分割(Park等,2003)。- 进一步提出的基于图谱的方法在计算PA和如何将PA纳入分割任务方面不同。
- 此外,PA可以包括相邻腹部结构之间的关系,以定义包围肝脏的解剖结构(Zhou等,2006)。
- 多图谱方法通过使用B样条变换模型进行非刚性配准(Slagmolen等,2007)、动态图谱选择和标签融合(Xu等,2015)或基于k最近邻的肝和非肝体素分类改善了肝分割结果(van Rikxoort等,2007)。
- 图割方法提供了一种有效的二元分割问题初始化方法,通过自适应阈值化(Massoptier和Casciaro,2007)和超像素。
基于深度学习的方法。与上述方法不同,深度学习,特别是卷积神经网络(CNN),是一种数据驱动的方法,可以在没有手工特征工程的情况下进行端到端的优化(Litjens等人,2017)。U形CNN架构(Ronneberger等人,2015)及其变体(Milletari等人,2016;Isensee等人,2020;Li等人,2018b)被广泛用于生物医学图像分割,并已经在广泛的分割任务中证明了其效率和鲁棒性。表现最好的方法共享多阶段过程的共性,
- 从3D CNN开始进行分割,然后使用马尔可夫随机场(Markov random field)对结果概率图进行后处理(Dou等人,2016)。
许多用于肝脏分割的早期深度学习算法将神经网络与专用的后处理程序相结合:Christ等人(2016)使用3D全连接神经网络和条件随机场,Hu等人(2016)依赖于3D CNN,然后是表面模型。相反,Lu等人(2017)使用CNN进行规范化,然后进行图割分割。
- 图像分割+条件随机场
3D全连接神经网络是一种用于图像分割的深度学习算法,它可以对输入的3D图像数据进行端到端的优化,而无需手工设计特征工程。条件随机场是一种概率图模型,可以将先验知识引入到图像分割中,通过对分割结果进行平滑化和优化来提高分割的准确性。在Christ等人的方法中,他们使用3D全连接神经网络对肝脏图像进行分割,然后将条件随机场用于后处理,以提高分割的准确性。该方法可以使分割结果更加平滑和自然。
肝脏分割:
与肝脏相比,肝癌的病变形态、大小和对比度范围更加全面。肝肿瘤可以出现在几乎任何位置,常常边界模糊。对比剂吸收的差异可能会引入额外的变异性。因此,肝肿瘤分割被认为是更具挑战性的任务。已发表的肝肿瘤分割方法可分为:
(1)阈值和空间正则化、(2)局部特征和学习算法以及(3)深度学习。
- 方法之一是阈值化和空间正则化。
-
基于肿瘤区域的灰度级值与肝脏和背景区域像素/体素的差异,阈值化是一种简单而有效的工具,可以自动将肿瘤与肝脏和背景分离,最初由Soler等人(2001)首次展示。
-
此后,阈值可以通过直方图分析(Ciecholewski和Ogiela,2007)、类间最大方差(Nugroho等人,2008)和迭代算法(Abdel-massieh等人,2010)来提高肿瘤分割结果。
-
空间规则技术依赖于(先前的)图像或形态学信息,例如肿瘤大小、形状、表面或空间信息。利用这些知识,可以引入正则化或惩罚的约束。
-
自适应阈值化方法可以与基于模型的形态学处理相结合,用于异质性病变分割(Moltz等人,2008,2009)。
-
基于轮廓(Kass et al., 1988)的肿瘤分割依赖于形状和表面信息,并利用概率模型(Ben-Dan和Shenhav,2008)或直方图分析(Linguraru等,2012)自动创建分割地图。水平集(Osher和Sethian,1988)方法允许对肿瘤形状进行数值计算,而无需参数化。
- 水平集方法与2D(Smeets等,2008)和3D(Jiménez Carretero等,2011)中的受监督的像素/体素分类相结合,用于肝肿瘤分割。
-
使用局部特征和学习算法的方法。
- 聚类方法包括k-means(Massoptier和Casciaro,2008)和模糊c-means聚类,使用可变形模型进行分割细化(Häme,2008)。
- 在监督分类方法中,有基于模糊分类的水平集方法(Smeets等,2008),支持向量机与基于纹理的可变形表面模型相结合进行分割细化(Vorontsov等,2014),基于纹理特征的AdaBoost(Shimizu等,2008)和图像强度剖面(Li等,2006),逻辑回归(Wen等,2009)以及递归分类和分解超像素的随机森林(Conze等,2017)。
深度学习
在LiTS之前,深度学习方法很少用于肝肿瘤分割任务。
Christ等人(2016)首次使用3D U-Net进行肝和肝肿瘤分割,提出了级联分割策略,并结合3D条件随机场进行细化。随后的许多深度学习方法都是与LiTS数据集一起开发和测试的。
得益于LiTS公共数据集的可用性,许多关于肝脏和肝脏分割的新深度学习解决方案被提出。U-Net架构被广泛使用和修改以提高分割性能。
- 例如,nn-UNet(Isensee等人,2020)首次在MICCAI 2018的LiTS中展示,被证明是在3D图像分割任务中表现最好的方法之一。相关工作将在结果部分讨论。
然而,总体挑战还要求参与者解决其他九个任务,包括脑肿瘤、心脏、海马、肺、胰腺、前列腺、肝血管、脾和结肠分割。因此,算法不一定只针对肝脏CT分割进行了优化。
- 研究的队列涵盖了多种类型的肝肿瘤疾病,包括原发性肿瘤疾病(如肝细胞癌和胆管癌)和继发性肝肿瘤(如来自结肠直肠癌、乳腺癌和肺癌的转移)。
肿瘤具有不同的病变与背景比(高密度或低密度)。图像代表了混合的术前和术后腹部CT扫描,并使用不同的CT扫描仪和采集协议进行获取,包括常见于真实世界临床数据的成像伪影(例如金属伪影)。
- 因此,它在分辨率和图像质量方面被认为是非常多样化的。平面图像分辨率范围从0.56毫米到1.0毫米,切片厚度范围从0.45毫米到6.0毫米。此外,轴向切片的数量在42到1026之间变化。肿瘤数量在0到12之间变化。肿瘤的大小在38 mm3到1231 mm3之间变化。测试集中的肿瘤发生率比训练集高。统计检验(p值=0.6)表明,训练集和测试集中的肝脏体积没有显著差异。训练集和测试集中的平均肿瘤HU值分别为65和59。LiTS数据统计摘要如表3所示。训练集和测试集的比例为2:1,并且训练集和测试集在中心分布方面相似。因此,对未见过的中心的普适性在LiTS中未经过测试。
评估指标
- Dice score
- d(v,S(A)):Average symmetric surface distance(ASSD)是一种评估医学图像分割性能的指标,用于测量分割结果与真实标注之间的表面距离。它计算了两个表面之间的距离,并取其平均值。它可以用于评估分割结果的边界精度和分割错误的程度。它是一种常用的分割评估指标之一。
- "Average symmetric surface distance"是指两个分割表面之间的平均距离,即将两个分割表面之间的距离值相加,然后除以表面点的数量得到的平均值。
- "Maximum symmetric surface distance"是指两个分割表面之间的最大距离,即在所有表面点之间计算距离,然后选择最大的距离作为结果。
- “Relative volume difference”(相对体积差)是一种用于衡量体积测量误差的指标,通常用于比较两个体积测量结果之间的差异。它计算的是两个体积测量结果之间的相对差异,即将两个体积值之差除以它们的平均值。这个指标可以用来评估医学图像分割算法的准确性,以及在不同数据集或不同算法之间进行比较。
鉴于病变检测的临床相关性,我们在附加分析中引入了三个检测度量。
为避免在患者没有肿瘤的情况下出现潜在问题,指标是全局计算的。为了评估病变级别的指标,必须存在预测和参考病变之间的已知对应关系。由于所有病变都被定义为单个二进制图,因此必须确定预测和参考掩模的连接组件之间的对应关系。
每个病变被定义为图像中的3D连通组件。如果预测的病变与其相应的参考病变有足够的重叠,即它们各自的分割掩模的交集与并集之比足够大,则认为病变已被检测到。这允许计算真阳性、假阳性和假阴性检测的数量,从而计算病变检测的精度和召回率。这些指标的定义如下:

Algorithm and architecture
本次比赛中,73个参赛方案采用了完全自动化的方法,而仅有一种方案是半监督的(J. Ma等人)。
在本次比赛中,U-Net衍生的架构被广泛采用,只有两种自动化方法采用了修改后的VGG-net(J. Qi等人)和k-CNN(J. Lipkova等人)。大多数参赛方案采用了从粗到细的方法,在不同的阶段上级联多个U-Net来执行肝脏和肝脏分割。
- 额外的残差连接和调整输入分辨率是对基本U-Net架构最常见的改进。
- 三种方案将单独的模型组合成集成技术。
- 在2017年,由于高计算复杂度,没有任何参赛方案直接使用原始图像分辨率的3D方法。然而,一些方案仅针对肿瘤分割任务使用小输入补丁的3D卷积神经网络。
- 其他方法则尝试通过使用2.5D模型架构来捕捉三维优势,即将一堆图像作为多通道输入提供给网络,并将该堆栈的中心切片的分割掩模作为网络输出。
Main Components of Segement Model
- 在大多数方法中,使用HU值剪裁、归一化和标准化的数据预处理是最常见的技术。
- 数据增强也被广泛使用,主要集中在标准几何变换,如翻转、移动、缩放或旋转。
- 个别提交实施了更高级的技术,如直方图均衡化和随机对比度归一化。
- 最常见的优化器在ADAM和带动量的随机梯度下降之间变化,其中一种方法依赖于RMSProp。
- 多个损失函数用于训练,包括标准和加权交叉熵、Dice损失、Jaccard损失、Tversky损失、L2损失
- 集合损失技术,将多个单个损失函数组合成一个。
Post-processing.
一些后处理方法也被大多数算法使用。
- 常见的后处理步骤是形成连接的肿瘤组件,并将肝脏掩模叠加在肿瘤分割上,以丢弃肝脏区域之外的肿瘤。
- 更先进的方法包括随机森林分类器、形态学滤波、特定的浅层神经网络以消除误报或用于填补肿瘤空洞的自定义算法。
Features of top-performing methods.
The best-performing methods at ISBI 2017 used cascaded U-Net approaches with short and long skip connections and 2.5D input images
- weighted cross-entropy loss functions
- a few ensemble learning techniques were employed by most of the top-performing methods,
- some common pre- and post-processing steps such as HU-value clipping and connected component labeling
一些在MICCAI 2017表现出色的参赛者(例如,J. Zou)整合了ISBI 2017的见解,包括集成学习的想法,添加残差连接和更复杂的基于规则的后处理或经典机器学习算法。
- 因此,与ISBI提交相比,主要的架构差异是更高的集成学习方法的使用率,更高的残差连接发生率以及更多的更复杂的后处理步骤。
- X. Li等人提出的另一种表现出色的方法提出了一个混合的见解,将2D和3D网络的优点集成到3D肝肿瘤分割任务中。
Technique trend and recent advances
- A significant advance was on the 3D deep learning model besides the 2D approaches.
- self-supervised pre-training frameworks to initialize 3D models for better representation than training them from scratch
- self-configuring pipeline to facilitate the model training and the automated design of network architecture
- added a 3D attention module for 3D segmentation models.
- focused on the special trait of liver and liver tumor segmentation and proposed a novel active contour-based loss function to preserve the segmentation boundary
- enhance edge information and cross-feature fusion for liver and tumor segmentation
- considered the varying lesion sizes and proposed a loss reweighting strategy to deal with size imbalance in tumor segmentation.
- attempted to deal with the heterogeneous image resolution with a multi-branch decoder
- 一种新兴趋势是利用现有的稀疏标记图像进行多器官分割。
- Huang等人(2020)尝试对单器官数据集(肝脏、肾脏和胰腺)进行联合训练。
- Fang和Yan(2020)提出了一个金字塔输入和金字塔输出网络,以压缩多尺度特征以减少语义差距。
- 最后,Yan等人(2020)开发了一种通用的病变检测算法,以多任务方式检测CT图像中的各种病变,并提出了从部分标记数据集中挖掘缺失注释的策略。
对于病变大小的分割表现。总的来说,提交的方法在大型肝癌的分割方面表现非常好,但是对于小型肿瘤的分割却很困难(见图8)。
许多小肿瘤只有几个体素的直径;此外,轴向切片的图像分辨率相对较高,为512×512像素。
因此,由于潜在的不同周围像素数量较少,很难检测到这些小结构,这些像素可能表明潜在的肿瘤边界(见图8)。这种情况加剧了医学成像中的噪声和伪影,这些伪影来自大小相似性;与周围肝组织的纹理差异以及它们的任意形状很难与实际的肝癌区分开来。
**总的来说,最先进的方法在具有大型肿瘤的体积上表现良好,在具有小型肿瘤的体积上表现较差。**在单个小肿瘤(<10mm3)出现的检查中,最差的结果被实现。在体积显示少于六个肿瘤且总肿瘤体积超过40mm3的情况下,最好的结果被实现(见图8)。在附录中,我们展示了三个LiTS挑战赛的所有提交方法的性能,针对每个测试体积进行了比较,按病变出现的数量和病变大小进行了聚类,见图A.10。
对比度对分割质量的影响
方法的分割质量受到肿瘤和肝脏HU值之间差异的影响。当前最先进的方法在显示肝脏和肿瘤之间更高对比度的体积中表现最佳。特别是在密度比背景肝脏高40-60 HU的病灶的情况下(见图8)。最差的结果是在对比度低于20 HU的情况下(见图8),包括密度低于肝脏的肿瘤。
- HU值的平均差异使网络更容易区分肝脏和肿瘤,因为可以将简单的阈值推导规则作为决策过程的一部分。有趣的是,更大的差异值并没有导致更好的分割结果。
HU值(Hounsfield Units)是计算机断层扫描(CT)成像中用于描述组织密度的单位。HU值是通过将组织密度值与水的密度进行比较而得出的,水的密度被定义为0 HU,而空气的密度为-1000 HU。因此,HU值可以用来区分不同密度的组织,如肿瘤和肝脏。在医学图像分割中,HU值通常用来确定肝脏和肿瘤的边界。
Future Work
此外,我们建议提供来自多个标注者的多个参考标注的肝肿瘤。这是因为肝肿瘤的分割由于小结构和模糊的边界而存在高度的不确定性(Schoppe等人,2020)。虽然现有基准测试中的大多数分割任务都被制定为一对一映射问题,但它并不能完全解决数据不确定性自然存在的图像分割问题。建模分割任务中的不确定性是一种趋势(Mehta等人,2020;Zhang等人,2020b),它将允许模型生成不仅一个而是多个合理的输出。因此,它将增强自动化方法在临床实践中的适用性。发布的注释数据集不仅限于基准测试分割任务,还可用作最近的形状建模方法(如隐式神经函数)的数据(Yang等人,2022;Kuang等人,2022;Amiranashvili等人,2021)。
考虑到参与LiTS基准数据集的七个机构的患者人口的规模和重要性多样性,我们认为它在医学图像分析方面的价值和贡献将在众多方向上得到高度赞赏。
-
一个例子是在域自适应研究方向中,LiTS数据集可用于考虑数据分布由于域变化(例如采集设置)而产生的明显差异/偏移(Glocker等人,2019;Castro等人,2020)。
-
另一个最近和引人注目的用例是联邦学习的研究方向,其中LiTS基准数据集的多机构性质可以进一步为联邦学习模拟研究和基准测试作出贡献(Sheller等人,2018、2020;Rieke等人,2020;Pati等人,2021)。它将针对LiTS相关任务的潜在解决方案,而不跨机构共享患者数据。我们认为联邦学习尤其重要,因为这一领域的科学成熟可能会导致多机构协作的范式转变。此外,它正在克服技术、法律和文化数据共享方面的关切,因为涉及的患者将始终留在其所属机构内。