一、前言
面向对象分类其实在学界统称基于对象的图像分析(OBIA),而在遥感等地学细分领域中,其称为基于地理对象的图像分析(GEOBIA),这种算法优势非常明显。与普通的像元暴力迭代分类不同,对象的概念体现在同质像元的集合,这样能够很大程度去除“椒盐效应”,区别于模糊分类的效果,其对象边界明显。
面向对象的处理方法中最重要的一部分是图像分割。现有图像分割算法主要有分水岭、SLIC、quickshift以及经典图割(Graphcut)等,按用途的不同,前三者会区分到细分领域中使用,比如医学中最多使用的往往是分水岭和SLIC,遥感中使用的最多是quickshift,其次SLIC。
实际上,ecognition已经很好地满足了现有研究前言的需求,但是关键它是商业软件。

图1 研究区
区域内有水有植被、裸地、建筑用地,基本大类都全了,应该是不差的实验数据。
二、分割算法
先介绍常见的两种分割算法吧。
SLIC分割是基于种子数的分割结果,按照设定的超像素个数,在图像内均匀的分配种子点。所以种子数量是影像分割的效果和速度的关键,数量越多,分割越精细,分割时间越长。不过在最近的实验中发现,种子数对时间的影响程度并不算大,而在像素数量的影响程度远大于前几个指标。在做10000*10000的像元分割时,种子数10000的分割时间达到了5min。当然这也不算很严谨的实验数据,还需要多做几个实验才能得出规律。在scikit-imagepython包中,SLIC已经被集成,当然自己写也不是什么问题,但是造轮不可取。该算法中的参数很多,但是主要还是分割种子数和紧凑性,其中紧凑性指示可选平衡色彩接近度和空间接近度,数值越高,空间接近度越重,使得超像素形状更加平方/立方,这个参数在一般情况的调参中,一般以10倍的大小浮动分割,查看情况,例如0.01,0.1,1,10,100。其他参数就按需设置了,这里不做赘述。
quickshift即快速位移聚类算法,也是非常强大的一种分割算法,基于核平均移动的近似值,可以在多尺度上计算分层分段,一定程度上对遥感尺度效应有奇效。quickshift最重要的参数有三两个,一个是ratio,是介于0和1之间平衡色彩空间接近度和图像空间接近,较高的值会增加色彩空间的重量。kernel_size是可选用于平滑样本密度的高斯内核宽度。越高意味着越少的群集。max_dist是可选数据距离的切点。越高意味着越少的群集。
现在先来看下用scikit-image包处理遥感tif影像的效果,代码如下:
from skimage.segmentation import mark_boundaries
from osgeo import gdal
from skimage.segmentation import slic, quickshift
def read_img(filename):
dataset = gdal.Open(filename)
im_width = dataset.RasterXSize
im_height = dataset.RasterYSize
im_geotrans = dataset.GetGeoTransform()
im_proj = dataset.GetProjection()
im_data = dataset.ReadAsArray(0, 0, im_width, im_height) #0, 0, im_width, im_height
del dataset
return im_width, im_height, im_proj, im_geotrans, im_data
def write_img(filename, im_proj, im_geotrans, im_data):
if 'int8' in im_data.dtype.name:
datatype = gdal.GDT_Byte
elif 'int16' in im_data.dtype.name:
datatype = gdal.GDT_UInt16
else:
datatype = gdal.GDT_Float32
if len(im_data.shape) == 3:
im_bands, im_height, im_width = im_data.shape
else:
im_bands, (im_height, im_width) = 1, im_data.shape
driver = gdal.GetDriverByName("GTiff")
dataset = driver.Create(filename, im_width, im_height, im_bands, datatype)
dataset.SetGeoTransform(im_geotrans)
dataset.SetProjection(im_proj)
if im_bands == 1:
dataset.GetRasterBand(1).WriteArray(im_data)
else:
for i in range(im_bands):
dataset.GetRasterBand(i + 1).WriteArray(im_data[i])
del dataset
img_path = r".\test.tif"
temp_path = r".\classification"
im_width, im_height, im_proj, im_geotrans, im_data = read_img(img_path)
im_data = im_data[0:3]
temp = im_data.transpose((2, 1, 0))
#segments_quick = quickshift(temp, kernel_size=3, max_dist=6, ratio=0.5)
segments_quick = slic(temp, n_segments=25000, compactness=10.)
mark0 = mark_boundaries(temp, segments_quick)
save_path = temp_path + "temp.tif"
re0 = mark0.transpose((2, 1, 0))
write_img(save_path, im_proj, im_geotrans, re0)返回的是一个二维数组,每个聚类的块的像元值都是一样的,而这个值仅代表这个块的顺序,不代表任何遥感或者物理意义。可以把这些值叫做ID。这些ID值,也是后续作为二维空间定位的重要数值。简单看一下这些分割以后的图像长什么样:
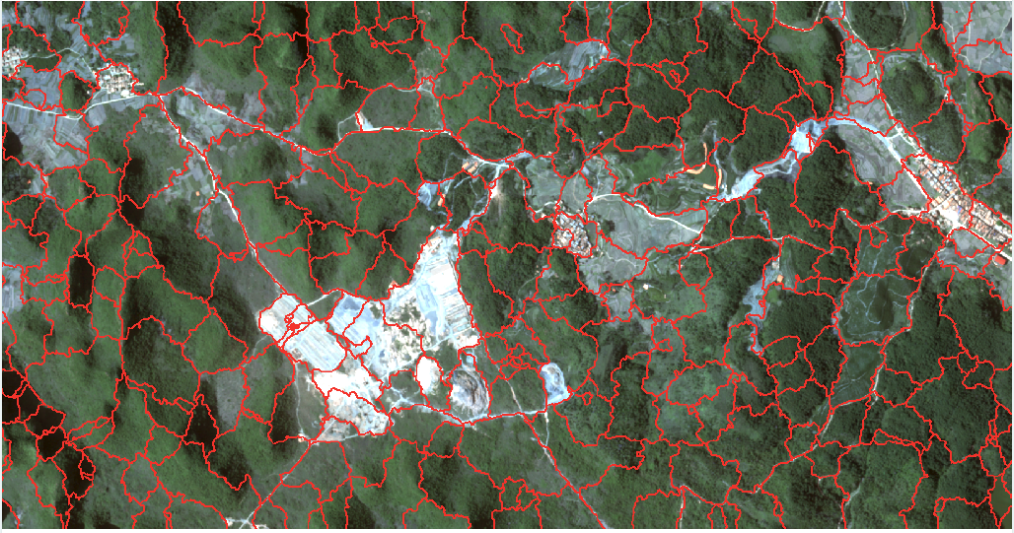
图2 quickshift分割结果

图3 slic分割结果
当然,代码执行完之后输出的其实是栅格tif,但是栅格文件不方便查看,所以我把栅格转成了矢量线,叠合在原来的多光谱遥感影像上,这样读者可以大致感受分割的情况。
三、地类数据
地物类别的数据是我自己用arcmap制作的,影像被我裁剪成了700*500的像元数以后,在该影像上根据目视简单分了几个大类:
林地
裸地
建筑
水域
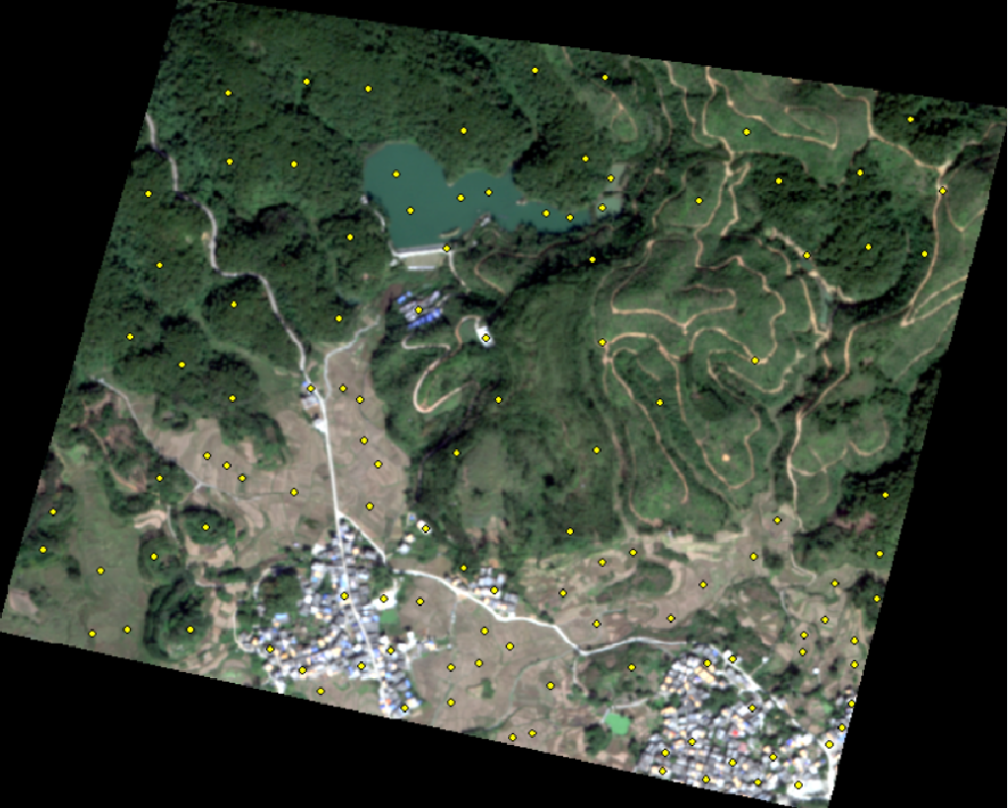
图4 分类样本
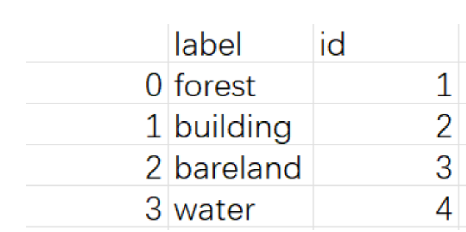
图5 训练样本表
按照机器学习惯例,需要把label数据拆分为测试集和验证集,比率是7:3,这里利用geopandas能够轻松实现,代码如下:
gdf = gpd.read_file(r".\sub_sample.shp")
class_names = gdf['label'].unique()
class_ids = np.arange(class_names.size) + 1
df = pd.DataFrame({'label': class_names, 'type': class_ids})
df.to_csv(r".\temp.csv")
gdf['type'] = gdf['label'].map(dict(zip(class_names, class_ids)))
gdf_train = gdf.sample(frac=0.7)
gdf_test = gdf.drop(gdf_train.index)
gdf_train.to_file(r".\train_data1.shp")
gdf_test.to_file(r".\test_data1.shp")四、构建特征
还是那句老话: 创造新的特征是一件十分困难的事情,需要丰富的专业知识和大量的时间。机器学习应用的本质基本上就是特征工程。——Andrew Ng
作为遥感的面向对象分类,可选的特征数可达上百个,一般选用的特征包括各种植被指数、均值、方差、标准差、灰度共生矩阵等,还有研究者利用其他如物候、温度等数据参与特征构建,然后进行特征优选,最后把特征放入随机森林训练器进行分类,本次实验重点在于python的实现,所以并没有构建过多的特征,而少量的特征意味着不需要进行特征优选,省了很多步骤。
值得一提的是,在特征构建这一步中,所花费的时长是分割的好几十倍,因此如果影像很大,还需要写多线程去加速特征构建过程。在10000*10000像元数的影像中,仅有三个特征的训练时长达到了1.5小时。
def segment_features(segment_pixels):
features = []
npixels, nbands = segment_pixels.shape
for b in range(nbands):
stats = scipy.stats.describe(segment_pixels[:, b])
band_stats = list(stats.minmax) + list(stats)[2:]
if npixels == 1:
band_stats[3] = 0.0
features += band_stats
return features
segment_ids = np.unique(segments)
objects = []
object_ids = []
for id in segment_ids:
segment_pixels = temp[segments == id]
object_features = segment_features(segment_pixels)
objects.append(object_features)
object_ids.append(id)五、后续
后续部分继续整理~~