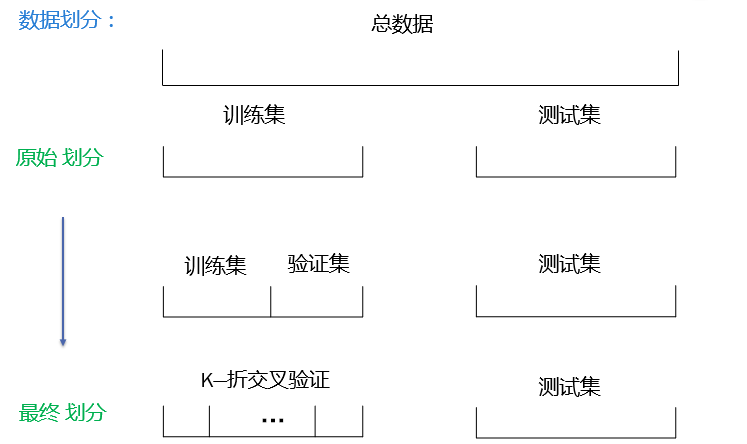
因为一个模型仅仅重复了刚刚训练过的样本的标签,这种情况下得分会很高,但是遇到没有训练过的样本就无法预测了。这种情况叫做过拟合。为了避免过拟合,一个常见的做法就是在进行一个(有监督的)机器学习实验时,保留一部分样本作为测试集(X_test, y_test)。为了提高训练数据的泛化能力,我们把训练集和测试集对半划分,即:X_test:y_test=1:1,test_size=0.5。但是仅仅如此,再训练时,训练集可能自身会出现过拟合现象,从而使测试集的验证效果降低。为了解决这个问题,另一部分数据被取出来作为“验证集”:在训练集上训练,验证集上评估,当实验看起开比较成功的时候,在测试集上进行最终的评估。但是把获得的数据分成三部分,会大量减少训练模型的样本数量,并且结果很依赖于随机选择的训练和验证集。解决这个问题的一个方法就是交叉验证(简写CV)。如采用10次交叉验证(或者留出法)。
下面看一篇博客,已经详细叙述了下他们的联系与区别
https://blog.csdn.net/laolu1573/article/details/51160019
在一个数据集上学习预测函数的参数并且在相同的数据集上测试是一种错误的方法:因为一个模型仅仅重复了刚刚训练过的样本的标签,这种情况下得分会很高,但是遇到没有训练过的样本就无法预测了。这种情况叫做过拟合。为了避免过拟合,一个常见的做法就是在进行一个(有监督的)机器学习实验时,保留一部分样本作为测试集(X_test, y_test)。需要注意的是,“实验”这个词并不是为了表示只是学术上使用,即使是商用场景,机器学习通常也是从实验着手。
在scikit- learn中,使用train_test_split这个帮助函数可以很快的将样本随机的切分成训练集和测试集。我们现在载入iris数据集来训练一
个线性支持向量机:
>>> import numpy as np >>> from sklearn import cross_validation >>> from sklearn import datasets >>> from sklearn import svm >>> iris = datasets.load_iris() >>> iris.data.shape, iris.target.shape ((150, 4), (150,))
我们现在可以很快的采样出一个训练集,并且拿出40%的数据来测试(评估)我们的估计器:
>>> X_train, X_test, y_train, y_test = cross_validation.train_test_split( ... iris.data, iris.target, test_size=0.4, random_state=0) >>> X_train.shape, y_train.shape ((90, 4), (90,)) >>> X_test.shape, y_test.shape ((60, 4), (60,)) >>> clf = svm.SVC(kernel='linear', C=1).fit(X_train, y_train) >>> clf.score(X_test, y_test) 0.96...
在评估不同设置(超参)对分类器的影响时(比如 SVM的需要人工设置的C 这个参数),因为需要不断调整参数直到分类器性能最好,仍然存在在测试数据集上的过拟合现象。这种情况下,模型会漏掉测试集数据的知识,评估矩阵不能反应其泛化性能。为了解决这个问题,另一部分数据被取出来作为“验证集”:在训练集上训练,验证集上评估,当实验看起开比较成功的时候,在测试集上进行最终的评估。
但是把获得的数据分成三部分,会大量减少训练模型的样本数量,并且结果很依赖于随机选择的训练和验证集。
解决这个问题的一个方法就是交叉验证(简写CV)。依然需要保留一个测试集来做最后评估,但是不需要验证集。最基本的方法,就是k-折交叉验证,训练集被分成K个小集合(其他方法将在后面描述,但是一般遵守相同的准则)。这K“折”的每个都依照下面的过程:
- k-1部分作为训练样本来训练模型;
- 用剩余1部分数据对训练好的模型进行验证。
下面的例子示例了怎样通过切分数据,拟合模型和连续5次计算得分(每次使用不同的切片)来估计线性核函数
SVM在iris数据上准确率。
>>> clf = svm.SVC(kernel='linear', C=1) >>> scores = cross_validation.cross_val_score( ... clf, iris.data, iris.target, cv=5) ... >>> scores array([ 0.96..., 1. ..., 0.96..., 0.96..., 1. ])
>>> print("Accuracy: %0.2f (+/- %0.2f)" % (scores.mean(), scores.std() * 2)) Accuracy: 0.98 (+/- 0.03)默认每次CV迭代计算的得分是估计器的score方法得分,可以使用得分参数改变默认配置。
>>> from sklearn import metrics >>> scores = cross_validation.cross_val_score(clf, iris.data, iris.target, ... cv=5, scoring='f1_weighted') >>> scores array([ 0.96..., 1. ..., 0.96..., 0.96..., 1. ])详情见The scoring parameter: defining model evaluation rules章节。在Iris数据集的例子中,每个类别的样本数目比较均衡,所以准确率和F1-score几乎是相等的。
当cv参数是一个整型时,cross_val_score默认使用KFold 或StratifiedKFold的方法,