[生物信息学] 寻找开放阅读框(Open Reading Frame)
前情提要:
并不是生物的所有 DNA 都是有意义并且可以直接转换成氨基酸,对于一串基因来说,它需要找到 起始密码子(start codon) 和 终止密码子(end codon) 才可以找到有效的 编码区(coding region),并将对应的 DNA 翻译成氨基酸。
ORF 示意如下:
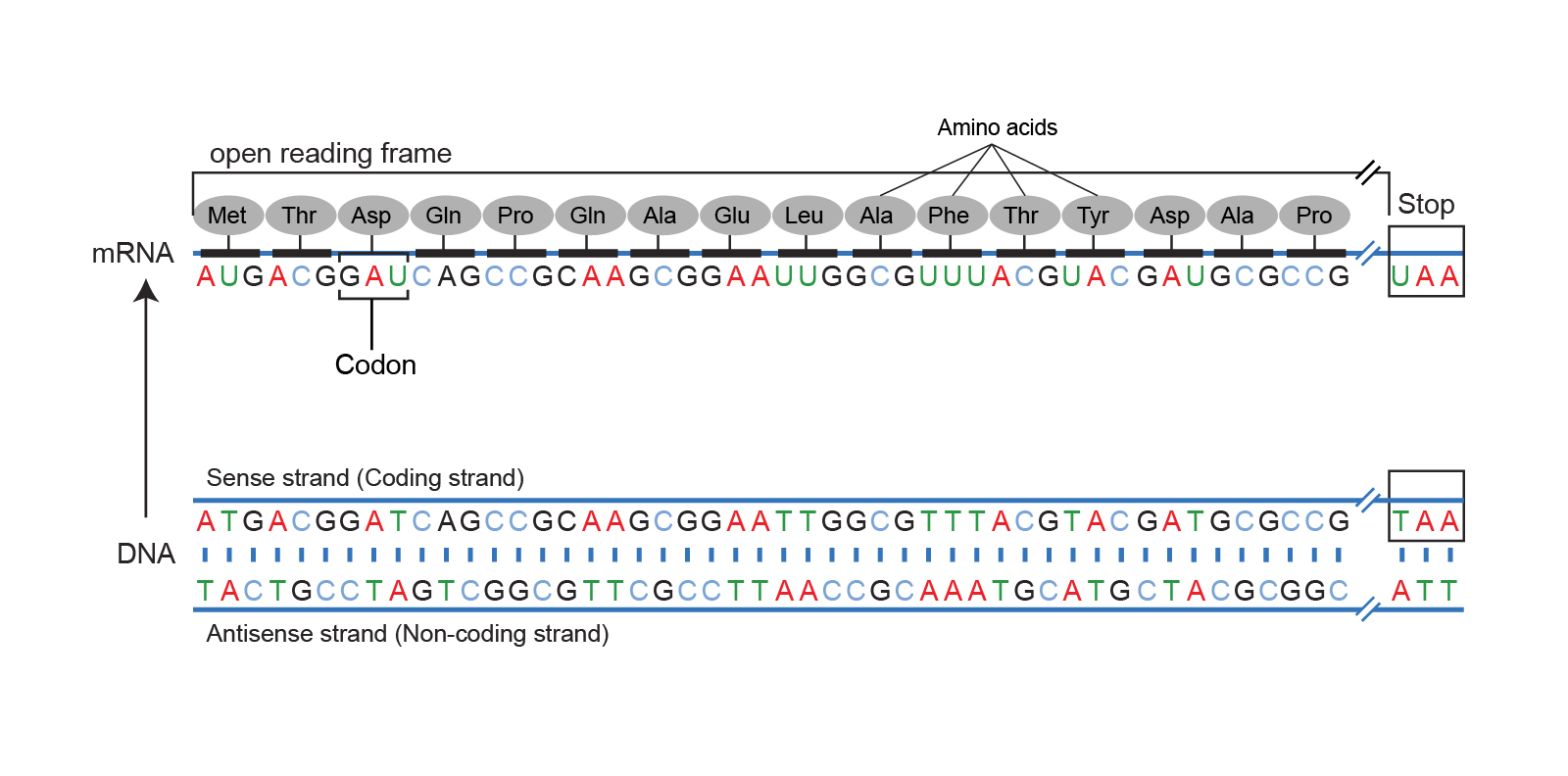
图像资料来源于: https://www.genome.gov/genetics-glossary/Open-Reading-Frame
上面这一段是 ORF,是完整的 DNA 链条中的一小部分,但是该片段有 起始密码子(start codon) 和 终止密码子(end codon),因此可以被转录成对应的氨基酸。
ORF 的阅读方法有六种,以 CGCTACGTCTTACGCTGGAGCTCTCATGGATCGGTTCGGTAGGGCTCGATCACATCGCTAGCCAT 为例:
原本的序列为: GCGATGCAGAATGCGACCTCGAGAGTACCTAGCCAAGCCATCCCGAGCTAGTGTAGCGATCGGTA,对应的反向互补为:ATGGCTAGCGATGTGATCGAGCCCTACCGAACCGATCCATGAGAGCTCCAGCGTAAGACGTAGCG。
-
+1 的阅读方式为:
CGC TAC GTC TTA CGC TGG AGC TCT CAT GGA TCG GTT CGG TAG GGC TCG ATC ACA TCG CTA GCC AT
-
+2 的阅读方式为:
C GCT ACG TCT TAC GCT GGA GCT CTC ATG GAT CGG TTC GGT AGG GCT CGA TCA CAT CGC TAG CCA T
-
+3 的阅读方式为:
CG CTA CGT CTT ACG CTG GAG CTC TCA TGG ATC GGT TCG GTA GGG CTC GAT CAC ATC GCT AGC CAT
-
-1 的阅读方式为:
ATG GCT AGC GAT GTG ATC GAG CCC TAC CGA ACC GAT CCA TGA GAG CTC CAG CGT AAG ACG TAG CG
-
-2 的阅读方式为:
A TGG CTA GCG ATG TGA TCG AGC CCT ACC GAA CCG ATC CAT GAG AGC TCC AGC GTA AGA CGT AGC G
-
-3 的阅读方式为:
AT GGC TAG CGA TGT GAT CGA GCC CTA CCG AAC CGA TCC ATG AGA GCT CCA GCG TAA GAC GTA GCG
DNA 资料来源于: https://vlab.amrita.edu/?sub=3&brch=273&sim=1432&cnt=1
+ 和 - 指的是方向,+ 为原本的 RNA 方向,- 为 DNA 的反向互补阅读方向。1,2,3 则是分别对应所取 DNA 阅读的起始点——氨基酸以 3 个 DNA 为一组,所以开始点可以从索引 0,索引 1,索引 2 开始。
随后只需要找到对应的 起始密码子(start codon) 和 终止密码子(end codon),就可以找到对应的 ORF。