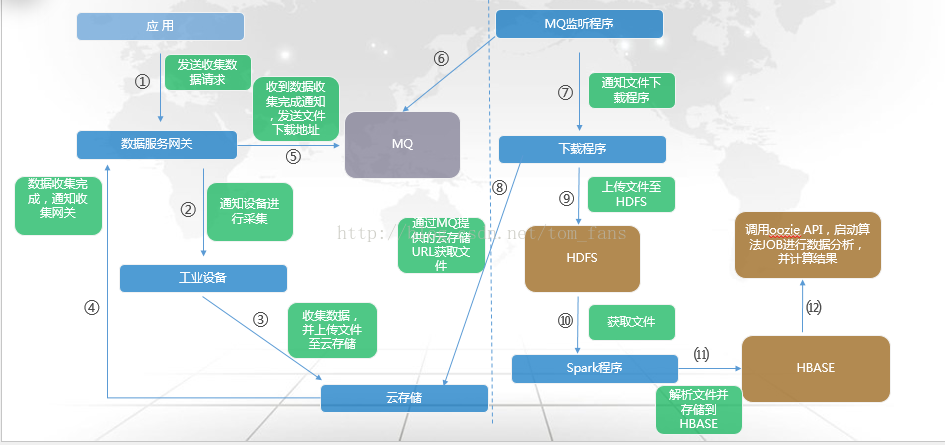
物联网,顾名思义,所有的数据采集是从设备采集的。设备有多种,有些通过传感器来采集,有些设备属于智能设备,本身就是一台小型计算机,能够自己采集,不管是传感器,还是智能设备本身,采集方式一般包含2种,一种是报文方式,所谓报文就是根据你设置的采集频率,比如1分钟一次,1秒一次进行数据传输,传输到哪里?一般放到MQ中。还有一种采集是以文件的方式采集,在做数据分析的时候,工业设备的数据希望是连续不断的,我们可以理解为毫秒级采集,就是设备不停的发送数据,然后形成一个文件或者多个文件。
以下是我的工业设备毫秒级采集及基本处理的流程图:
报文采集这里就不提了,因为它的方式和互联网的日志生成极为相同,就好比日志是每条每条进入,报文的概念是一样的。 那么毫秒级采集由于数据量比较大,所以整个方式处理会有些不同,但是整体和互联网实际也没有区别,毕竟互联网也有很多是以文件方式来处理的。
既然要采集,那么必须有一个策略,策略主要包含以下2个方面:
1. 采集时间
这个很容易理解,你需要采集5分钟还是10分钟
2. 采集参数
每个设备有上千个甚至几千个参数,你需要下发策略,告诉设备你要采集哪些参数
设备开始采集之后,然后以文件的方式保存,然后通过网络传送到云存储。 由于数据量大,这里通常要做系列化以及压缩处理, 避免给磁盘带来太大开销,另外就是网络,毕竟我们从设备直接把文件传输到云存储。
通过以上步骤,我们的采集基本就搞定了,然后就是数据的处理。数据采集完成如果直接调用后台的Spark或者其他程序来处理文件呢? 由于设备毕竟不是计算机,不能像互联网那样直接通知甚至直接调用,所以我们使用了MQ消息服务,每次采集完一个文件,并上传到云存储,就是用云存储的API去写一条数据到MQ,表示有一个文件已经完成了,监听程序发现新文件,并下载然后上传到HDFS,并通过API直接调用oozie的JOB,传输相关文件名,地址等参数。 这个时候后台挂在oozie上的JOB就开始处理文件。
数据分析的逻辑和处理逻辑是一样的,我们所有的后台JOB挂在oozie,只要需要就通过rest API直接触发调用。 分析主要还是算法,主要的流程从采集,处理,分析这一系列的路打通之后,我们所要做的就是优化算法。
另外,物联网的数据分析对时间的顺序有相当大的依赖,一批数据,就算因为几条数据时间乱了,也会导致所有数据无效。更简单理解,实际就是时间序列数据,和监控数据概念类似。
而HADOOP平台,包括 spark , storm等组件属于分布式组件,在处理的时候要注意到,分布式很多时候不适合时间序列数据,因为分布式的插入已经处理,不能保证数据完全按照时间的顺序来处理。目前我使用了一个极其简单的方案来解决,那就是spark只设置一个partition , 另外存储到HBASE的rowkey也是根据时间顺序的。
我知道,上面的做法会导致分布式没有起到作用,比如一次处理的插入或者查询全部在一个hbase region, 包括spark只有一个partition,也就意味着只有一个task.
相关代码:
1. 当文件上传至HDFS,需要先解压缩,并处理系列化的文件
package com.isesol.mapreduce;
import java.io.IOException;
import java.util.ArrayList;
import java.util.zip.GZIPInputStream;
import org.apache.hadoop.conf.Configuration;
import org.apache.hadoop.fs.FSDataInputStream;
import org.apache.hadoop.fs.FSDataOutputStream;
import org.apache.hadoop.fs.FileSystem;
import org.apache.hadoop.fs.Path;
import org.json.JSONObject;
import com.sun.imageio.spi.RAFImageInputStreamSpi;
public class binFileRead_forscala {
public static ArrayList<String> binFileOut(String fileName, int cols) throws ClassNotFoundException {
ArrayList resultset = new ArrayList();
try {
// 解压缩gz文件,并写入新的文件
unGzipFile(fileName);
Configuration conf = new Configuration();
FileSystem fs = FileSystem.get(conf);
FSDataInputStream inputStream = fs.open(new Path(fileName.substring(0, fileName.lastIndexOf('.'))));
byte[] itemBuf = new byte[8];
int i = 0;
int j = 0;
String result = "";
while (true) {
int readlength = inputStream.read(itemBuf, 0, 8);
if (readlength <= 0)
break;
double resultDouble = arr2double(itemBuf, 0);
i++;
j++;
if (j == 1) {
result = result + resultDouble;
} else {
result = result + "," + resultDouble;
}
if (i % cols == 0) {
resultset.add(result);
result = "";
j = 0;
}
}
inputStream.close();
} catch (Exception e) {
e.printStackTrace();
}
cleanFile(fileName);
return resultset;
}
public static double arr2double(byte[] arr, int start) {
int i = 0;
int len = 8;
int cnt = 0;
byte[] tmp = new byte[len];
for (i = start; i < (start + len); i++) {
tmp[cnt] = arr[i];
cnt++;
}
long accum = 0;
i = 0;
for (int shiftBy = 0; shiftBy < 64; shiftBy += 8) {
accum |= ((long) (tmp[i] & 0xff)) << shiftBy;
i++;
}
return Double.longBitsToDouble(accum);
}
/*
* 通过APP ID获取要查询的ROWKEY,再查询字段
*
*
*
*
*/
public static ArrayList<String> getHaseCols(String bizData) throws ClassNotFoundException, IOException {
ArrayList colList = new ArrayList<String>();
int i;
JSONObject obj = new JSONObject(bizData);
JSONObject obj2 = new JSONObject(obj.getString("ipx_bigData_cmd-isesol"));
String param = obj2.getString("collectParam");
String val[] = param.split("\\|");
for (i = 0; i < val.length; i++) {
colList.add(val[i]);
}
return colList;
}
// 解压缩 gzip文件,然后生成新的非GIZP文件。
public static void unGzipFile(String sourcedir) {
String ouputfile = "";
try {
// 建立gzip压缩文件输入流
Configuration conf = new Configuration();
FileSystem fs = FileSystem.get(conf);
if(! fs.exists(new Path(sourcedir))){
System.out.println("the file is not exists");
}
FSDataInputStream inputStream = fs.open(new Path(sourcedir));
// 建立gzip解压工作流
GZIPInputStream gzin = new GZIPInputStream(inputStream);
// 建立解压文件输出流
ouputfile = sourcedir.substring(0, sourcedir.lastIndexOf('.'));
FSDataOutputStream fout = fs.create(new Path(ouputfile));
int num;
byte[] buf = new byte[1024];
while ((num = gzin.read(buf, 0, buf.length)) > 0) {
fout.write(buf, 0, num);
}
System.out.println("ungzip is successful,the file name is " + ouputfile);
gzin.close();
fout.close();
} catch (Exception ex) {
System.out.println("ungip process errors");
System.err.println(ex.toString());
}
return;
}
public static void cleanFile(String filename) {
String ouputfile = "";
try {
Configuration conf = new Configuration();
FileSystem fs = FileSystem.get(conf);
ouputfile = filename.substring(0, filename.lastIndexOf('.'));
fs.delete(new Path(ouputfile));
System.out.println("clean file successful!");
} catch (Exception ex) {
System.out.println("clean file failed!");
System.err.println(ex.toString());
}
}
}
2. 解压并处理二进制之后,使用spark解析,并存储到HBASE
package com.iesol.high_frequency
import java.io.BufferedInputStream;
import java.io.BufferedReader;
import java.io.DataInputStream;
import java.io.DataOutputStream;
import java.io.File;
import java.io.FileInputStream;
import java.io.FileNotFoundException;
import java.io.FileOutputStream;
import java.io.FileReader;
import java.io.IOException;
import java.io.ObjectInputStream;
import java.io.ObjectOutputStream;
import java.io.Serializable;
import scala.util.control._;
import java.nio.file.Path;
import java.nio.file.Paths;
import com.isesol.mapreduce.binFileRead_forscala
import java.util.List;
import org.apache.spark.SparkContext
import org.apache.spark.SparkConf
import org.apache.hadoop.hbase.HBaseConfiguration
import org.apache.hadoop.mapred.JobConf
import org.apache.hadoop.hbase.mapred.TableOutputFormat
import org.apache.hadoop.hbase.util.Bytes
import org.apache.spark.rdd.RDD.rddToPairRDDFunctions
import org.apache.hadoop.hbase.client.Put
import org.apache.hadoop.hbase.io.ImmutableBytesWritable
import org.apache.hadoop.hbase.spark._
import org.apache.hadoop.hbase.client.Scan
import org.apache.hadoop.hbase.TableName
import org.apache.hadoop.hbase.filter._
import org.apache.hadoop.hbase.filter.CompareFilter.CompareOp
import org.apache.hadoop.hbase.client.HTable
import scala.util.Random
object parseFile {
def main(args: Array[String]) {
/*
* fileName 文件名称
*
* appId App请求发出者
*
* bizId 业务唯一号码
*
* bizData 收集信息,包括字段,机床号
*
* collectType 采集类型: collect/healthcheckCollect
*
*/
val fileName = args(0)
val appId = args(1)
val machine_tool = args(2)
val bizId = args(3)
val bizData = args(4)
val collectType = args(5)
var table = "" //default collect type is None
println("fileName is " + fileName)
println("bizId is " + bizId)
println("machine_tool is " + machine_tool)
println("collectType is " + collectType)
if(collectType == "collect") {
table = "t_high_frequently"
} else if (collectType == "healthcheckcollect") {
table = "t_high_frequently_healthcheck"
} else {
table = "t_high_frequently"
}
println("table name is " + table)
val conf = new SparkConf()
conf.setMaster("local").setAppName("high frequency collection " + appId)
val sc = new SparkContext(conf)
val hbaseCols = binFileRead_forscala.getHaseCols(bizData)
val total_colNums = hbaseCols.size()
println("total cols number is " + total_colNums)
val getFile = binFileRead_forscala.binFileOut(fileName, total_colNums)
val getData = new Array[String](getFile.size())
for (num <- 0 to getFile.size() - 1) {
getData(num) = getFile.get(num)
}
val hbaseCols_scala = new Array[String](hbaseCols.size())
for (num <- 0 to hbaseCols.size() - 1) {
hbaseCols_scala(num) = hbaseCols.get(num)
println("hbase cols is " + hbaseCols_scala(num))
}
val bankRDD = sc.parallelize(getData,1).map { x => x.split(",") }
/* start to parse filename, and get the file suffix number */
var fileNum = 0
var name = Array[String]()
var cur_time = Array[String]()
if (fileName.endsWith(".gz")) {
name = fileName.split("\\.")
cur_time = fileName.split("_").take(4)
}
for (i <- name if i != "gz") {
fileNum = i.split("_").last.toInt
}
fileNum += 1000;
/* start to parse RDD ,and put data into hbase, the row key rule is : machine_tool#bizId#fileNum#time */
try {
bankRDD.foreachPartition { x =>
var count = 0
val hbaseconf = HBaseConfiguration.create()
hbaseconf.set("hbase.zookeeper.quorum", "datanode01.isesol.com,datanode02.isesol.com,datanode03.isesol.com,datanode04.isesol.com,cmserver.isesol.com")
hbaseconf.set("hbase.zookeeper.property.clientPort", "2181")
hbaseconf.set("maxSessionTimeout", "6")
val myTable = new HTable(hbaseconf, TableName.valueOf(table))
// myTable.setAutoFlush(true)
myTable.setWriteBufferSize(3 * 1024 * 1024)
x.foreach { y =>
{
val rowkey = System.currentTimeMillis().toString()
val p = new Put(Bytes.toBytes(machine_tool + "#" + cur_time(3) + "#" + fileNum + "#" + rowkey))
for (i <- 0 to hbaseCols_scala.size - 1) {
p.add(Bytes.toBytes("cf"), Bytes.toBytes(hbaseCols_scala(i)), Bytes.toBytes(y(i)))
}
myTable.put(p)
}
}
myTable.flushCommits()
myTable.close()
}
} catch {
case ex: Exception => println("can not connect hbase")
}
}
}