1.中午和下午一直在做高软开发的PPT,晕死。
2.这里由于sv用到了贝叶斯因子,并且有对感兴趣的基因得到:
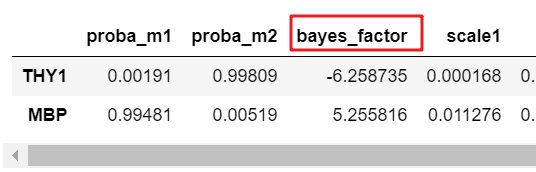 图1
图1
贝叶斯因子的计算公式:


从书中给出的例子可以看出,要接受原假设,就要贝叶斯因子>1的。

sv中取log也是为了防止贝叶斯因子过小而产生溢出吧。 那么就目前的理解,在图1中,THY1是拒绝原假设,而选择备择假设,即在第二种细胞类型中表达量较高,MBP是接受原假设,在第一种类型中表达量较高。不差异表达的情况:log贝叶斯因子在0附近,不能支持原假设。
3.MMD损失
https://blog.csdn.net/a529975125/article/details/81176029(待看)
4.再次注意损失函数包括似然损失、及降维取样KL散度。
我觉得对于这个BP算法我还需要手动地推导一遍才能更好地理解吧,我学了这么久,但是我还是不明白为什么能够拟合函数。。。
#隐隐觉得它找DE基因是一次性的进行批处理,德出不同的细胞类型之间的差异分数???
#其实还有不明白的地方,它是如何和其他方法对比的?DEseq,DCA等,它们进行方法比较所使用的指标是什么?
找DE的过程,先用原来的train_set训练一遍,然后选取两种celltype的细胞进行编码获取到result。
5.由于它重复了:

搜了一下重复采样,并没有什么结果。
6.np.vstack-在垂直方向上合并。
https://www.jianshu.com/p/2469e0e2a1cf
7.np.mean-有axis参数,求哪个轴上的。
8.
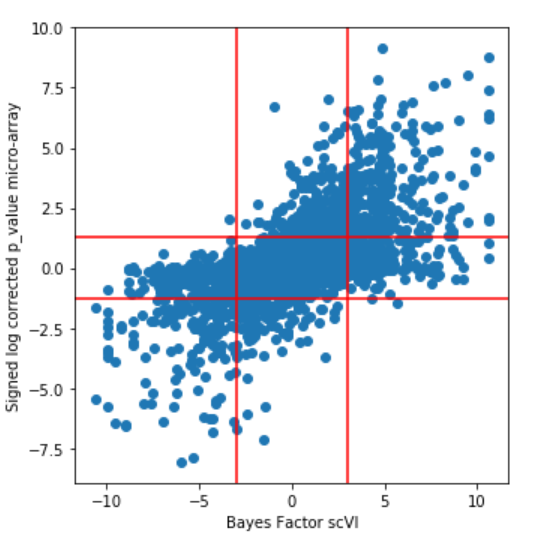
我很想知道这张图代表着什么呢?横轴贝叶斯因子在±3之外的范围是差异表达的,纵轴是标注答案吗?那可以看出来sv效果一般般啊,怎么就好了?
9.library size用在什么地方了?就那个单独对它编码的网络输出的结果?
对的,对前半部分编码网络中的输出library的结果,又输入到了解码网络中,去和px_scale计算px_rate。
10.在拼接函数中:
import numpy as np a=np.concatenate((np.repeat(0, 6), np.repeat(1, 6)), axis=0) print(a)
axis=0应该是按照行进行粘贴,但是其中粘贴的是两个一维的,它的shape是(6,),所以并不是通常的按照行来粘贴。运行结果如下:
[0 0 0 0 0 0 1 1 1 1 1 1]