激活qiime2的执行环境:source activate qiime2-2019.4
如何查看conda已有的环境:conda info -e
以下分析流程参考:https://docs.qiime2.org/2019.4/tutorials/qiime2-for-experienced-microbiome-researchers/
1、数据准备
现在我们常用的就是这种格式的数据,每个样品一对数据文件
wget \ -O "casava-18-paired-end-demultiplexed.zip" \ "https://data.qiime2.org/2019.4/tutorials/importing/casava-18-paired-end-demultiplexed.zip"
下载解压后,文件夹中文件如下:
2、将数据转换为qza格式(qiime新定义的自己的格式类型,有点编程中对象的含义)
qiime tools import \ --type 'SampleData[PairedEndSequencesWithQuality]' \ --input-path casava-18-paired-end-demultiplexed \ --input-format CasavaOneEightSingleLanePerSampleDirFmt \ --output-path demux-paired-end.qza
3、查看数据质量
qiime demux summarize --i-data demux-paired-end.qza --o-visualization demux-summary-1.qzv
用以下命令查看结果:
qiime tools view demux-summary-1.qzv
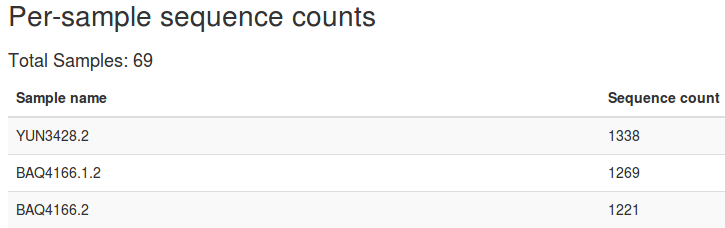

4、双端序列合并成单端
qiime dada2 denoise-paired --p-trim-left-f 9 --p-trim-left-r 9 --p-trunc-len-f 10 --p-trunc-len-r 10 --i-demultiplexed-seqs demux-paired-end.qza --o-representative-sequences demux-paired-end-denoise.qza --o-table table-1.qza --o-denoising-stats stats-1.qza

扫描二维码关注公众号,回复:
6517614 查看本文章


4、对数据进行剪切
qiime dada2 denoise-paired --p-trim-left-f 9 --p-trim-left-r 9 --p-trunc-len-f 10 --p-trunc-len-r 10 --i-demultiplexed-seqs demux-paired-end.qza --o-representative-sequences demux-paired-end-denoise.qza --o-table table-1.qza --o-denoising-stats stats-1.qza
https://forum.qiime2.org/t/demultiplexing-and-trimming-adapters-from-reads-with-q2-cutadapt/2313