之所以需要重采样是由于不同的病人体型不同,但最后数字成像的分辨率是一样的,这就导致了一定程度的失真变形。
但医学图像例如dcm或nii格式,都会带有SliceThickness,PixelSpacing类似的属性,可以利用这些属性去尽量还原真实物体。
例如:(下图第二个物体是星星,忘记改过来了)
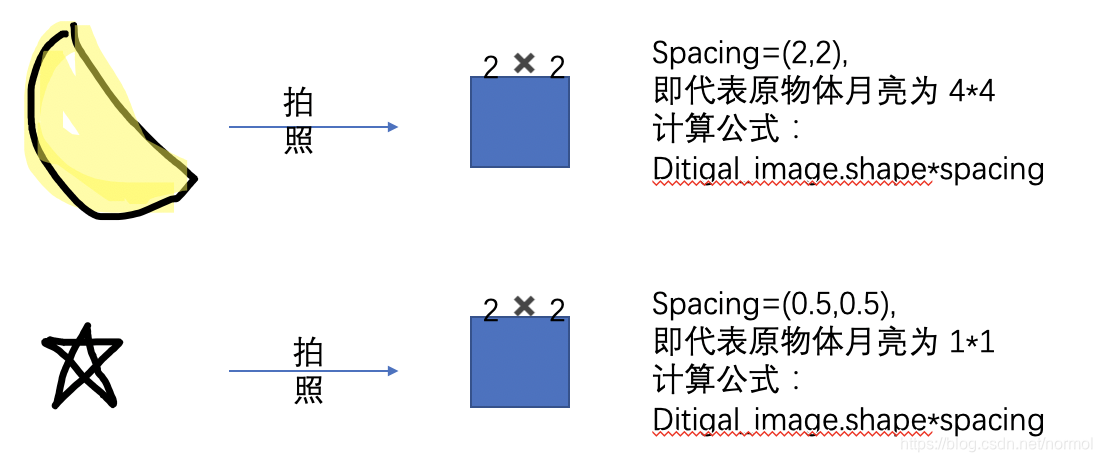
现在希望spacing变为(1,1),意思是希望数字成像的图片大小就代表原始物体大小。
若spacing=(2,2)则表示原始物体大小是数字成像大小的两倍

因此,
# ct_array 是 z,y,x
# refactor_size = ct.GetSpacing()[-1] / new_spacing_z,...
ct_array = ndimage.zoom(ct_array, (ct.GetSpacing()[-1] / new_spacing_z, ct.GetSpacing()[1] / new_spacing_y, ct.GetSpacing()[2] / new_spacing_x), order=3)
但是如果更细致的话,会发现spacing很多时候都是浮点数,意味着我们数字图像还原成原始图像大小后也会是浮点数,但数字图像的分辨率是没有浮点数的,这时就需要round处理,最终的需求还是要计算出refacor_size提供给插值函数,只不过计算refactor_size的过程绕了一下。

def resample(image, scan, new_spacing=[1,1,1]):
image = imgs_to_process
scan = patient
# Determine current pixel spacing
spacing = map(float, ([scan[0].SliceThickness] + list(scan[0].PixelSpacing)))
spacing = np.array(list(spacing))
resize_factor = spacing / new_spacing
new_real_shape = image.shape * resize_factor
new_shape = np.round(new_real_shape)
real_resize_factor = new_shape / image.shape
new_spacing = spacing / real_resize_factor
image = scipy.ndimage.interpolation.zoom(image, real_resize_factor)
return image, new_spacing
print("Shape before resampling\t", imgs_to_process.shape) #Shape before resampling (129, 512, 512)
imgs_after_resamp, spacing = resample(imgs_to_process, patient, [1,1,1])
print("Shape after resampling\t", imgs_after_resamp.shape) #Shape after resampling (206, 292, 292)
当然,也可以不进行插值处理,因为插值后会导致图片间的大小不同,无法输入网络,我见到的一个处理是这样的:
只对轴向(z)进行差值,y,x固定大小缩放,
这样对于每一个数据,就只会有切片数量不同。
在输入网络时,每个数据随机取样n个slice数目相同的三维图片,输入网络即可。