Django中的模型(model)本质是数据库中的表(table),当作为开发者时,总会涉及到数据库的操作,而Django中也提供了一些函数,实现sql语句中的增删改查:
惯例,先看模型:

1 # coding:utf-8 2 import django.utils.timezone as timezone 3 from django.db import models 4 from blast_service.models import DiskInfo 5 # Create your models here. 6 class GenomicsFileInfo(models.Model): 7 COMPUTE_STATE = ( 8 (0, 'computing'), 9 (1, 'computed'), 10 (2, 'idle'), 11 ) 12 ACHIVE_STATUS = ( 13 (0, 'Not archive'), 14 (1, 'Archive failed'), 15 (2, 'Archive success'), 16 (3, 'Ignore'), 17 (4, 'Archiving') 18 ) 19 20 SEND_STATUS = ( 21 (0, 'idle'), 22 (1, 'sended'), 23 ) 24 userid = models.CharField(max_length=32,blank=True) 25 cluster_account = models.CharField(max_length=32) 26 filename = models.CharField(max_length=100) 27 md5 = models.CharField(max_length=32, blank=True) 28 file_path = models.CharField(max_length=500) 29 #updatetime = models.DateTimeField(auto_now_add=True) 30 diskinfo = models.ForeignKey(DiskInfo) 31 size = models.BigIntegerField(blank=True) 32 compute_state = models.IntegerField(choices=COMPUTE_STATE, default=2) 33 achive_status = models.IntegerField(choices=ACHIVE_STATUS, default=0) 34 achive_path = models.CharField(max_length=500, blank=True) 35 updatetime = models.DateTimeField(default = timezone.now) 36 is_deleted = models.BooleanField(default=False, verbose_name=u'delete tag') 37 class Meta: 38 unique_together = ('cluster_account','file_path', 'filename') 39 db_table = 'GenomicsFileInfo' 40 def __unicode__(self): 41 return self.file_path
1 添加新记录
数据库操作第一个,在Django也最常用的一个操作就是添加新记录,向表中添加记录有两种方式:
a. create

1 ... 2 from cnsa_service.models import GenomicsFileInfo #导入需要使用到的两个表 3 from blast_service.models import DiskInfo #在GenomicsFileInfo表中disk_info设置的是连接了外键的,所以也需要导入这个DiskInfo表格 4 ... 5 6 def add_line(): #使用create来添加一个新纪录 7 GenomicsFileInfo.objects.create(cluster_account='luhuifang', filename='test2.fa.gz', file_path='/Users/xiaohui/work/ETL/09.CNSA/SRC/cngb_bak/bakup/cnsa/upload/luhuifang', size='5273', diskinfo=DiskInfo.objects.get(disk_path='/Users/xiaohui/work/ETL/09.CNSA/SRC/cngb_bak/bakup')) 8 9 if __name__=='__main__': 10 add_line()
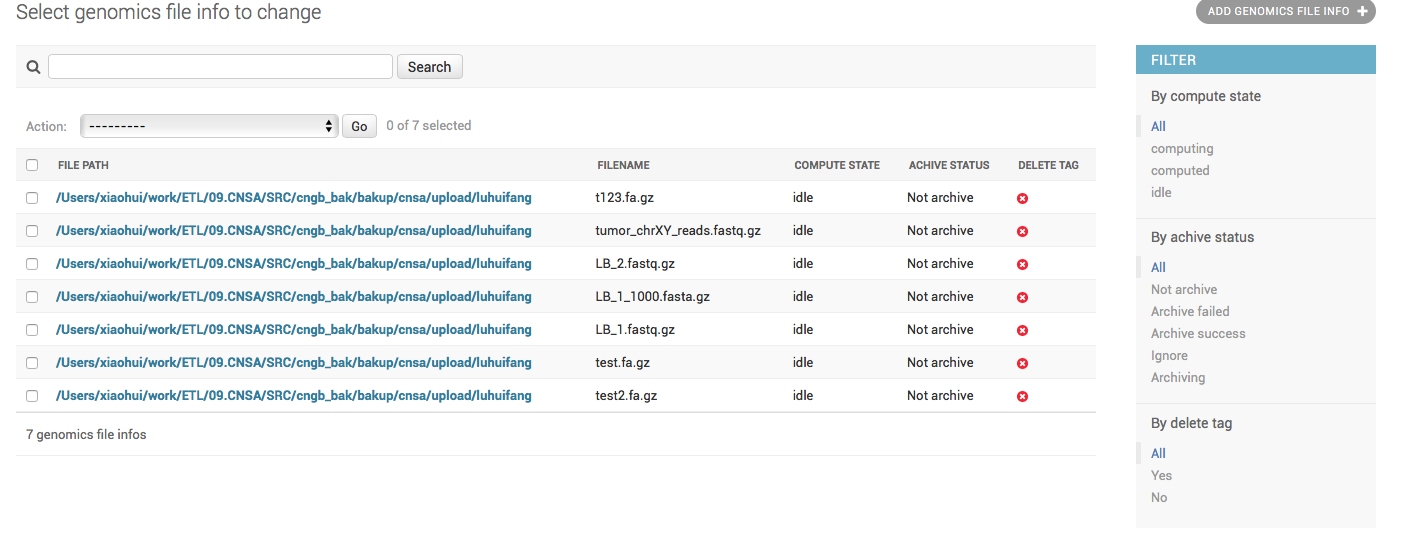
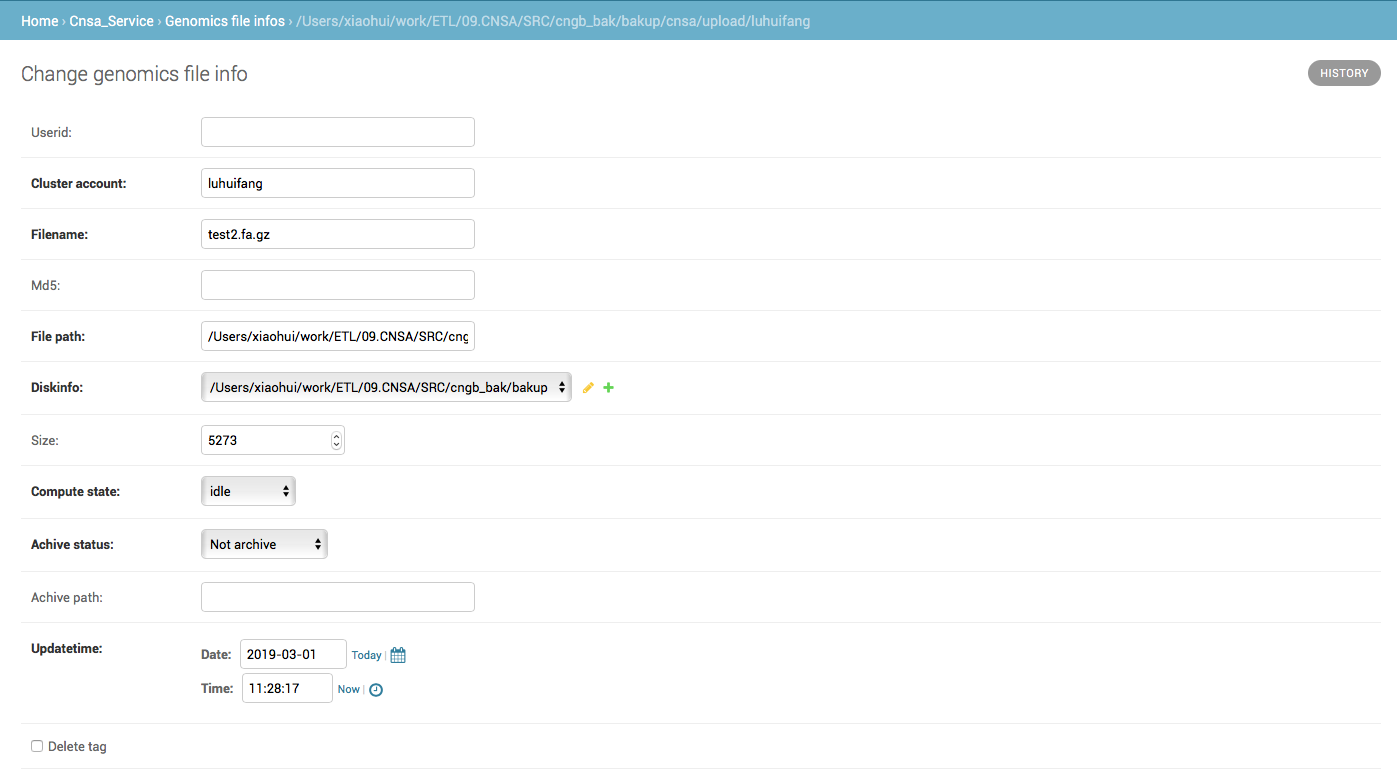
可以在Django管理后台查询到刚刚添加的新纪录:
b. save

... from cnsa_service.models import GenomicsFileInfo from blast_service.models import DiskInfo ... def save_line(): #使用save方式添加新记录 obj = GenomicsFileInfo(cluster_account='luhuifang', filename='test3.fa.gz', file_path='/Users/xiaohui/work/ETL/09.CNSA/SRC/cngb_bak/bakup/cnsa/upload/luhuifang', size='5273', diskinfo=DiskInfo.objects.get(disk_path='/Users/xiaohui/work/ETL/09.CNSA/SRC/cngb_bak/bakup')) obj.save() #一定要调用save()函数才能真正写入数据库中 if __name__=='__main__': save_line()
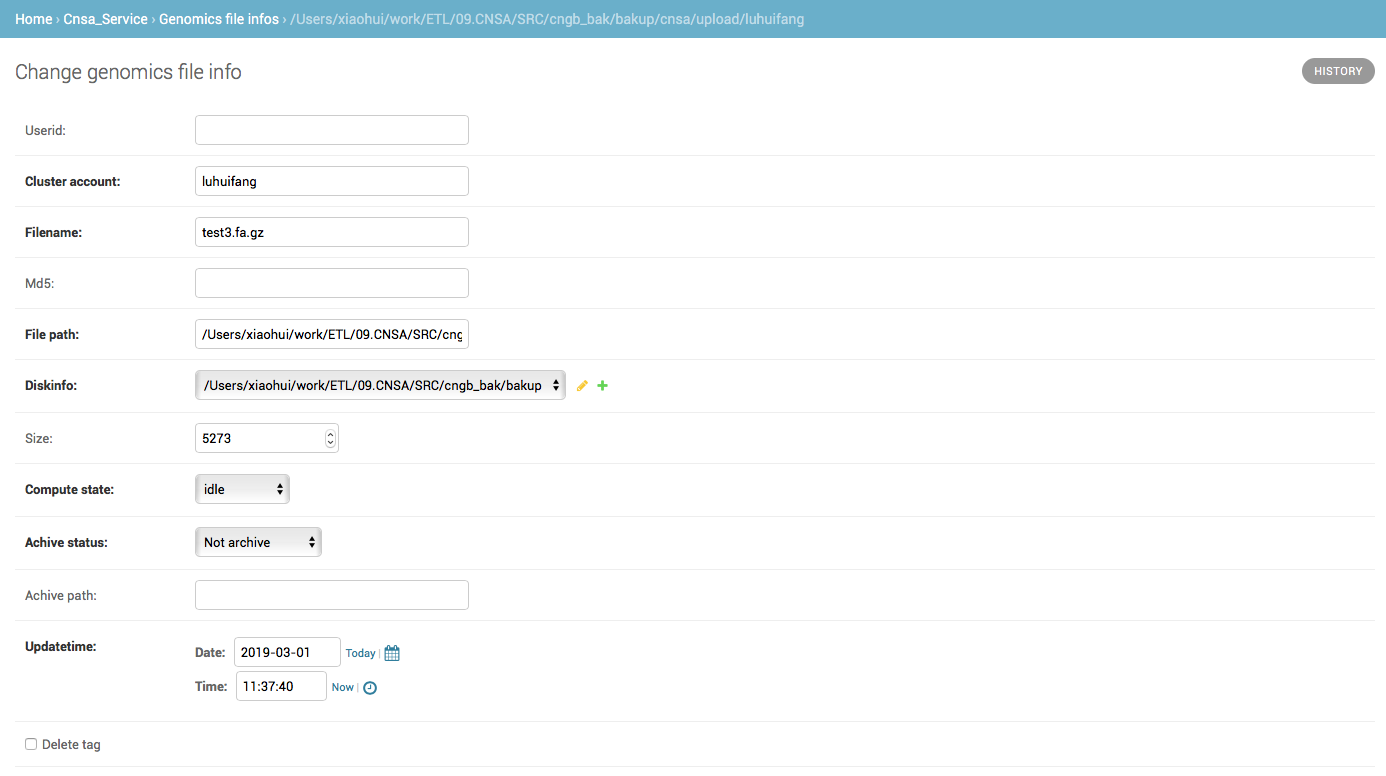
同样可以在后台查询到新加的记录:
2 基础查询
基础查询就如同sql语句中的select,可以查询满足条件的记录:
a. 查询单一结果: get
这个函数只能查询数据库中满足条件记录数为1的记录,1)如果使用此函数查询结果记录数大于1,报MultipleObjectsReturned错误;2)如果使用此函数查询不存在的记录,报DoesNotExist错误。

... from cnsa_service.models import GenomicsFileInfo ... def get_line(): #objs = GenomicsFileInfo.objects.get(filename='LB_3.fastq.gz') #这条记录不存在,报错 DoesNotExist #objs = GenomicsFileInfo.objects.get(cluster_account='tianliu') #用户名为tianliu的记录有两条,会报错MultipleObjectsReturned objs = GenomicsFileInfo.objects.get(filename='LB_2.fastq.gz') #这次查询刚好返回数据库中只有一个记录的结果,正确运行 if objs: print objs if __name__=='__main__': get_line()
b. 查询所有记录:all
这个函数与get不同,使用all函数,可以不带参数,数据库表中所有记录都会被返回。

... from cnsa_service.models import GenomicsFileInfo ... def all_line(): objs = GenomicsFileInfo.objects.all() #不带参数, 返回数据库中所有记录 if objs: print objs if __name__ == '__main__': all_line()
c. 查询结果计数:count
count函数是对查询的结果进行计数的函数。

... from cnsa_service.models import GenomicsFileInfo ... def count_line(): objs = GenomicsFileInfo.objects.count() #统计数据库中所有记录的条数 if objs: print objs if __name__=='__main__': count_line()
3 条件过滤查询
以上get和all函数要么只能查询一条记录,要么就查询所有记录,如果需求是查询一部分满足条件的记录怎么办呢?在Django中使用filter函数可以对查询结果进行过滤。
过滤条件的语法: 属性名称__比较符=值 ##因为语法中使用了两个下划线(__)来分割属性名称和比较符,所以属性名称中不可以包含两个下划线。
语法中的比较符可以包括以下:
... from cnsa_service.models import GenomicsFileInfo ... #exact:表示判等。 #查询用户名为 luhuifang 的文件记录 GenomicsFileInfo.objects.filter(cluster_account__exact='luhuifang') #也可简写为: GenomicsFileInfo.objects.filter(cluster_account='luhuifang') #contains:是否包含,模糊查询。(说明:如果要包含%无需转义,直接写即可) #例1:查询文件名中包含test的文件记录 GenomicsFileInfo.objects.filter(filename__contains='test') #例2:startswith、endswith:以指定值开头或结尾。 GenomicsFileInfo.objects.filter(filename__endswith='fq.gz') #`以上运算符都区分大小写,在这些运算符前加上i表示不区分大小写,如iexact、icontains、istartswith、iendswith.` #isnull:是否为null。 #例:查询md5值不为空的记录 GenomicsFileInfo.objects.filter(md5__isnull=False) #in:是否包含在范围内。 #例:查询文件大小是0和1024的记录 GenomicsFileInfo.objects.filter(size__in=[0,1024]) #比较查询 # gt 大于 (greater then) # gte 大于等于 (greater then equal) # lt 小于 (less then) #lte 小于等于 (less then equal) #例:查询文件大小大于0的记录 GenomicsFileInfo.objects.filter(size__gt=0) #日期查询,可以将datetime类型的字段分开年月日等等来进行比较运算。 #year、month、day、week_day、hour、minute、second:对日期时间类型的属性进行运算。 #例:查询更新时间为2018年的文件记录。 GenomicsFileInfo.objects.filter(updatetime__year=2018) #例:查询2019年3月1号之前更新的记录。 GenomicsFileInfo.objects.filter(updatetime__lt='2019-03-01')
4 查询排序
数据库查询排序是希望按照需求将查询到的结果进行排序,这个问题有两个方法解决:
a. 写模型的时候,在models.py中设置
... from django.db import models ... class GenomicsFileInfo(models.Model): ... class Meta: ordering = ['updatetime'] #这句话主要用作默认排序,这里是按照更新日期进行排序,以后查询的时候,结果都会默认按照更新日期从小到大进行排序,除非使用了显示的order_by函数
一个小问题:按照更新时间(updatetime字段)来排序,发现数据库保存的时间和现在的时间不一致,这个主要原因是Django默认的时间是格林尼治时间,与中国时间有8小时时差,要解决这个问题可以在Django的setting.py中修改配置:
... LANGUAGE_CODE = 'en-us' USE_I18N = True USE_L10N = True #USE_TZ = True # 默认值为True,当这个值为True时表示使用默认时间(格林尼治时间),不管下面TIME_ZONE如何设置都没有用 USE_TZ = False #自定义修改为False,表示使用以下TIME_ZONE设置的时区 TIME_ZONE = 'Asia/Shanghai' #将TIME_ZONE设置为'Asia/Shanghai'表示使用上海的UTS时间 ...
b. 使用order_by函数进行显示排序
使用filter函数过滤查询之后想对结果进行排序,则可以使用order_by函数:
... from cnsa_service.models import GenomicsFileInfo ... def order_line(): objs = GenomicsFileInfo.objects.filter(cluster_account='luhuifang').order_by('updatetime') #按照updatetime排序
#objs = GenomicsFileInfo.objects.filter(cluster_account='luhuifang').order_by('-updatetime') #按照updatetime倒叙排序, 带多个参数就是按照多个字段排序 if objs: for obj in objs: print '{0} {1}'.format(obj.updatetime, obj.filename) if __name__=='__main__': order_line()
5 删除记录
删除记录使用delete函数
... from cnsa_service.models import GenomicsFileInfo ... def delete_line(): objs = GenomicsFileInfo.objects.filter(cluster_account='tianliu').delete() #删除记录 if objs: print objs if __name__=='__main__': delete_line()
6 修改记录
修改记录使用update函数
... from cnsa_service.models import GenomicsFileInfo ... def update_line(): objs = GenomicsFileInfo.objects.filter(filename='test2.fa.gz').update(cluster_account='tianliu') #修改文件名为test2.fa.gz的记录的用户名为 tianliu if objs: print objs if __name__=='__main__': update_line()
