大家好,我们经常会发现GO、kegg、gsea等富集分析的结果会产生很多term,如果top10之内有我们的目的term,那很好!但是如果我们想要研究的term不在top10里面怎么办呢?
甚至我们想要研究的term在top200之后,这时候咋办啊
其实我们在进行富集分析之前,可能已经对自己想要研究的方向有了大概了解。
那么问题就变成了:如何实现 从富集分析的结果里找到支撑我们想法的所有term?
-
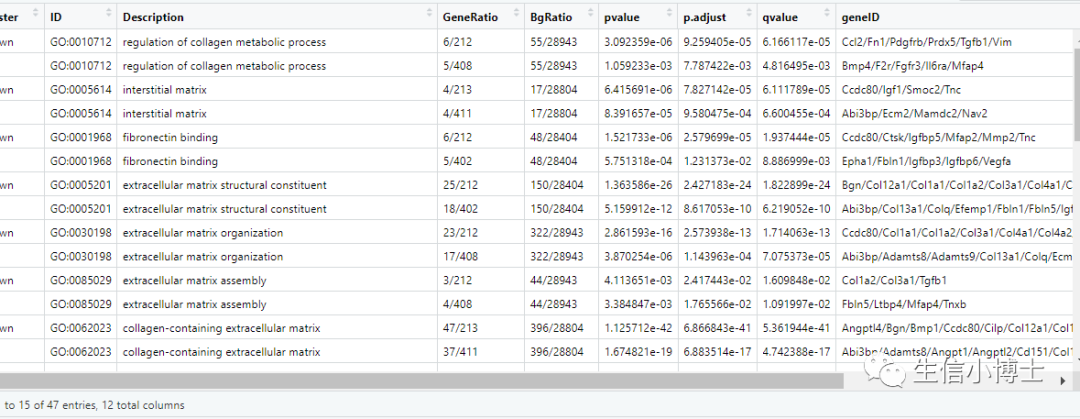
1 比如我们有了这样的富集结果,总共有2000多条:
-

-
2 假如我对纤维化感兴趣,那我可以用这几个词去检索:coll matrix fibro
如果你想研究其他的通路,换个关键词就行了
#代码
selected_lung_terms=xx.all %>%
dplyr::filter(stringr::str_detect(pattern = "coll|matrix|fibro",Description)) %>%
group_by(Description) %>%
add_count() %>%
dplyr::arrange(dplyr::desc(n),dplyr::desc(Description)) %>%
mutate(Description =forcats:: fct_inorder(Description))这样找到的都是和纤维化相关的term
-
3 对这些富集分析结果进行可视化
ggplot(xx.all %>%
dplyr::filter(stringr::str_detect(pattern = "coll|matrix",Description)) %>%
group_by(Description) %>%
add_count() %>%
dplyr::arrange(dplyr::desc(n),dplyr::desc(Description)) %>%
mutate(Description =forcats:: fct_inorder(Description))
, #fibri|matrix|colla
aes(Cluster, Description)) +
geom_point(aes(fill=p.adjust, size=Count), shape=21)+
theme_bw()+
theme(axis.text.x=element_text(angle=90,hjust = 1,vjust=0.5),
axis.text.y=element_text(size = 12),
axis.text = element_text(color = 'black', size = 12)
)+
scale_fill_gradient(low="red",high="blue")+
labs(x=NULL,y=NULL)
# coord_flip()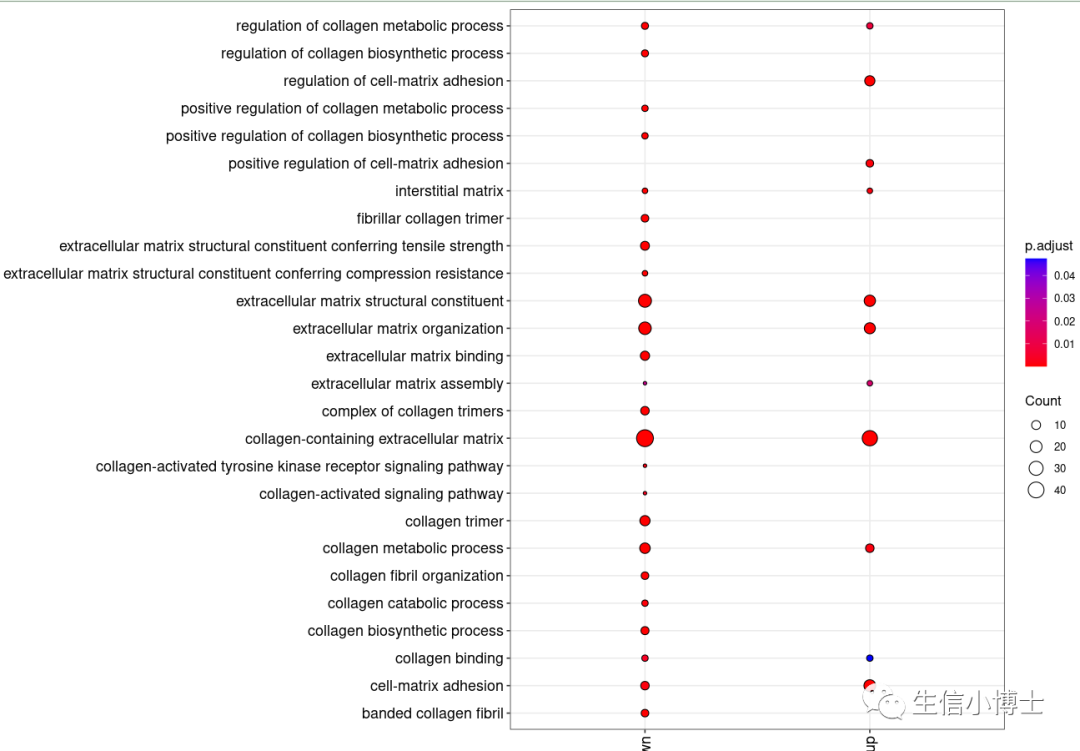
-
4 如果你想把目的term的所有基因都取出来:
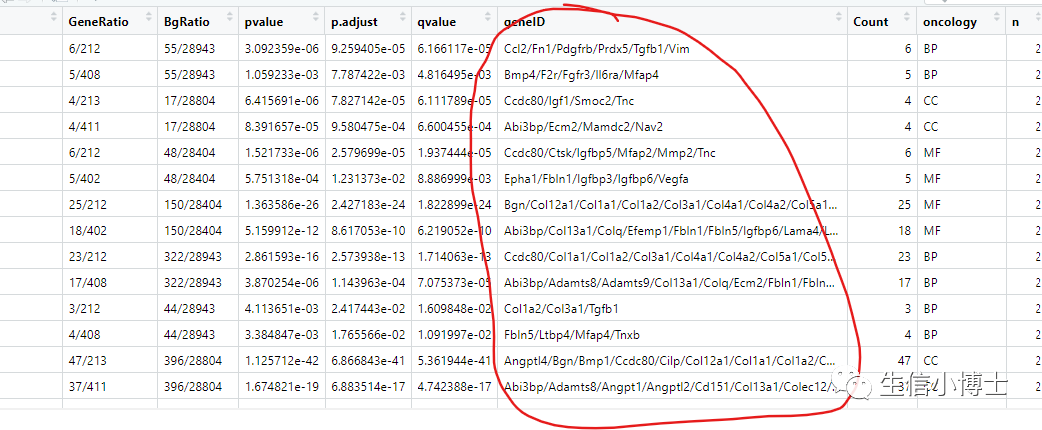
代码如下:
stringr::str_split(string = selected_lung_terms$geneID,pattern = "/",simplify = TRUE) %>%as.vector()所有的基因:
-
5 如果你想看基因在这些term中出现的频率:
stringr::str_split(string = selected_lung_terms$geneID,pattern = "/",simplify = TRUE) %>%as.vector() %>%table() %>%sort()可以看到col1a1出现的次数最多
最后其实对结果的可视化有很多种,我上述展示的有点丑,如果大家感兴趣,下次写写可视化的方法
最后,祝各位都能得偿所愿