美国国家健康与营养调查( NHANES, National Health and Nutrition Examination Survey)是一项基于人群的横断面调查,旨在收集有关美国家庭人口健康和营养的信息。
地址为:https://wwwn.cdc.gov/nchs/nhanes/Default.aspx
《在nhanes数据库挖掘教程5》中我们已经介绍了如何绘制如何对插补后的5个数据进行效应值合并,然后绘制多元性线性回归的限制立方样条图(RCS).不少粉丝后台私信问,为什么只用了a1数据,能不能用上全部数据,后面我想了一下,我们可以使用全部的插补数据做一个敏感性分析增强我们文章结果的可信度。
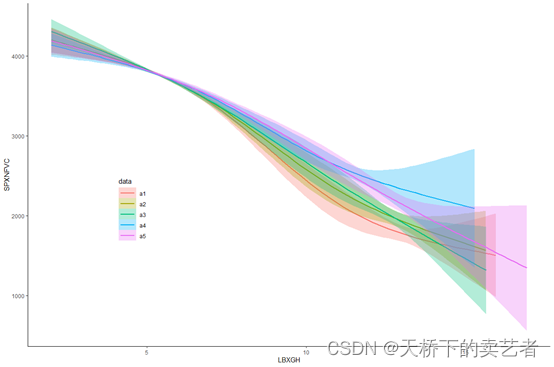
上图中有5条曲线,代表了5个插补数据,线条趋势很接近,表明了我们插补数据的结论很可靠,这样有助于增强我们文章结论的可靠性。
好了下面废话不多说,马上开始,假设你看过上一篇文章《nhanes数据库挖掘教程3–对数据进行多重插补》,并已经通过插补生了5个数据(a1-a5)并进行了分析,我们直接进入绘图环节。
我们先来画a1的,导入数据和R包
library(survey)
library(rms)
bc<-read.csv("E:/nhanes/a1.csv",sep=',',header=TRUE)
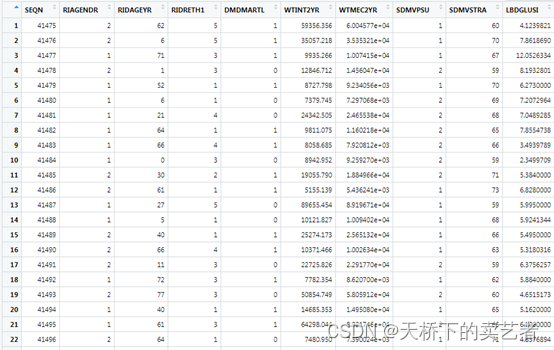
我介绍一下数据,SEQN:序列号,RIAGENDR, # 性别, RIDAGEYR, # 年龄,RIDRETH1, # 种族,DMDMARTL, # 婚姻状况,WTINT2YR,WTMEC2YR, # 权重,SDMVPSU, # psu,SDMVSTRA,# strata,LBDGLUSI, #血糖mmol表示,LBDINSI, #胰岛素( pmmol/L),PHAFSTHR #餐后血糖,LBXGH #糖化血红蛋白,SPXNFEV1, #FEV1:第一秒用力呼气量,SPXNFVC #FVC:用力肺活量,ml(估计肺容量),LBDGLTSI #餐后2小时血糖。
我们自选1-16行,后面是缺失数据分析,我们不需要
转分类变量为因子
bc$RIAGENDR<-as.factor(bc$RIAGENDR)
bc$RIDRETH1<-as.factor(bc$RIDRETH1)
bc$DMDMARTL<-as.factor(bc$DMDMARTL)
本文为转载文章,原文地址如下:https://mp.weixin.qq.com/s?__biz=MzI1NjM3NTE1NQ==&mid=2247488693&idx=1&sn=965b582465c489f5e86812397a35bf0c&chksm=ea26f4a9dd517dbf6342f7e0909fe4b9436bf8ba87d83673691e6c6cb9d9831a983a4e6ab15f#rd