1.简述
最近在把paddleocr 中cls分类模型通过ncnn部署框架部署时,发现onnx -> ncnn 模型的转换过程中出现问题。因为之前的项目都是使用ncnn框架部署的,只能去解决模型转换问题了。
2. 问题描述与分析
模型在onnx推理代码上正常,当把模型转换为ncnn模型之后,发现模型最后的softmax输出的维度不对。模型的为5个分类,onnx的输出为[1, 5] 是正确的,但是跑ncnn模型发现输出为 [1, 8, 5] ,经过打印log分析,是 softmax层前面的 fc层导致的问题。
分析:
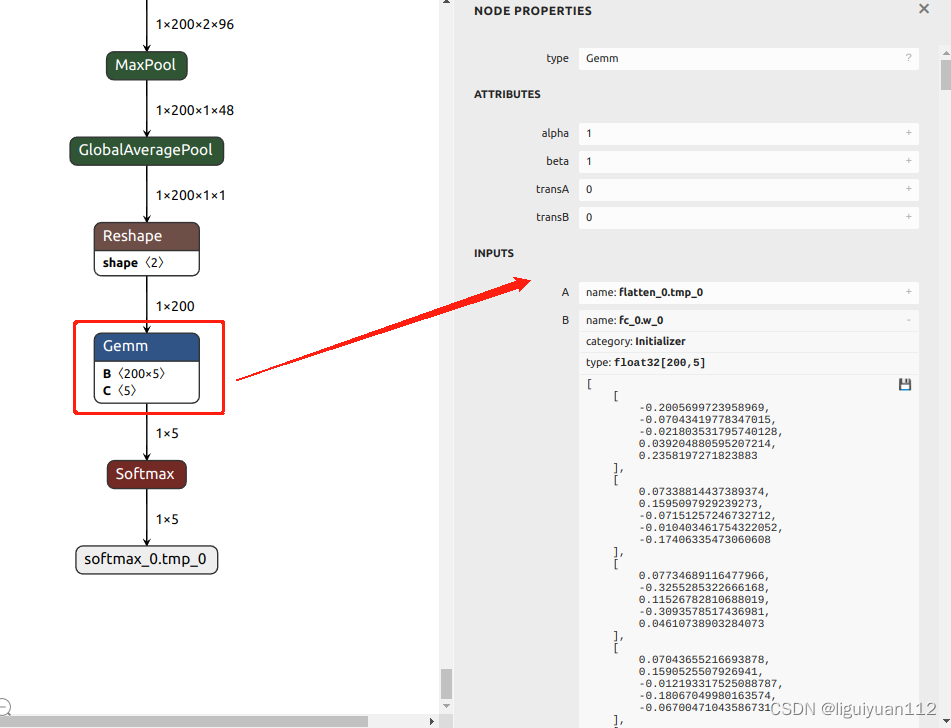
通过Netron工具可视化模型,fc层前面的输入维度为 A: [1, 200],权重B的维度为 B: [200, 5],偏置 C为 C: [5]。我们再看到Gemm层的属性,transA = 0, transB = 0,表示矩阵A和B都不需要转置操作。此时全连接操作是可以直接计算的:
A * B + C,对应的维度为 [1, 200] * [200, 5] + [5] => [1, 5] 输出维度正确
当把模型转换为ncnn模型时:
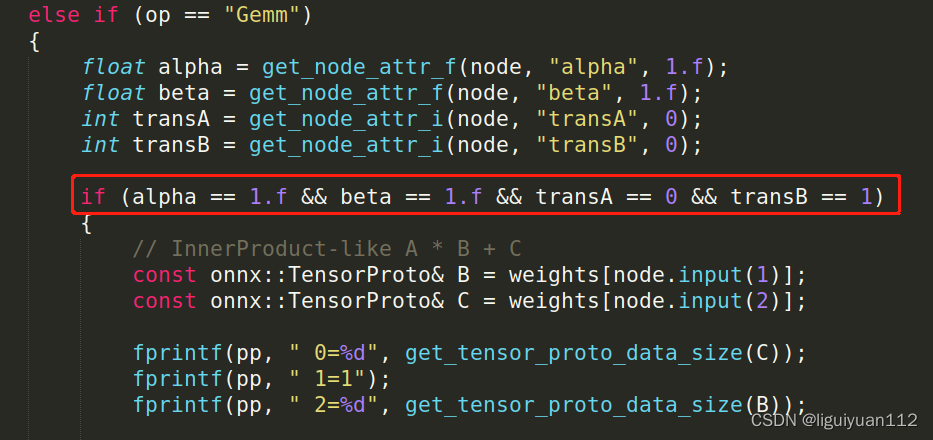
看到onnx2ncnn.cpp文件中关于Gemm 这个op的判断,代码中
(alpha == 1.f && beta == 1.f && transA == 0 && transB == 1) 的值为true时,才会跑 InnerProduct-like A * B + C 的流程,否则会进入 gemm矩阵乘法的流程。这样看来我的ncnn模型推理结果不正确 是因为fc层中的transB = 0,导致跑到了gemm的执行流程,没有跑到InnerProduct的执行流程。
跑到gemm矩阵乘法流程为什么输出的维度就不正确呢,我想原因还是矩阵乘法的计算问题,计算的维度:
A * B = [1, 200] * [5, 200] ,这个矩阵是不能正常计算的,由于维度不同不能直接计算,但是在执行代码的时候没出错的原因,可能是由于gemm分块计算的原因,然后最后输出为 [1, 8, 5]。
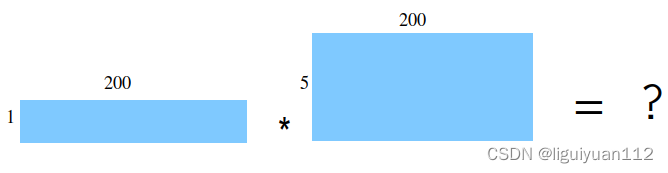
3. 解决方法
在onnx2ncnn.cpp代码中加入 transB = 0 时,也跑InnerProduct的流程,然后在代码中把矩阵 B 的数据进行转置一下就可以了。
else if (alpha == 1.f && beta == 1.f && transA == 0 && transB == 0)
{
// InnerProduct-like A * B + C
const onnx::TensorProto& B = weights[node.input(1)];
const onnx::TensorProto& C = weights[node.input(2)];
fprintf(pp, " 0=%d", get_tensor_proto_data_size(C));
fprintf(pp, " 1=1");
fprintf(pp, " 2=%d", get_tensor_proto_data_size(B));
int weight_data_size = get_tensor_proto_data_size(B);
int num_output = B.dims(B.dims_size() - 1);
int num_input = weight_data_size / num_output;
int quantize_tag = 0;
fwrite(&quantize_tag, sizeof(int), 1, bp);
// reorder num_input-num_output to num_output-num_input
{
const float* bptr = B.has_raw_data() ? (const float*)B.raw_data().data() : B.float_data().data();
for (int j = 0; j < num_output; j++)
{
for (int k = 0; k < num_input; k++)
{
float vb = bptr[k * num_output + j];
fwrite(&vb, sizeof(float), 1, bp);
}
}
}
fwrite_tensor_proto_data(C, bp);
}然后再重新编译一下代码,重新转换一下模型就正常了。
至此,问题终于得以解决,弄了好多天才解决问题,实属不易啊。