文章《手把手教你受用R语言建立COX回归后画立列线图》发布后,不少人发信息给我说,说我的calibration curve校正曲线图做得很难看,让我老脸微微一红。

今天重新来修正一下,原来的代码过一下,具体含义看原来文章
library(survival)
library(rms)
data(package="survival")
bc<-cancer
bc <- na.omit(bc)
dd <- datadist(bc)
options(datadist="dd")
f <- cph(Surv(time, status) ~ age + sex + ph.ecog + pat.karno +wt.loss,
x=T, y=T, surv=T, data=bc, time.inc=36)
surv <- Survival(f)
nom <- nomogram(f, fun=list(function(x) surv(36, x), function(x) surv(60, x),
function(x) surv(120, x)), lp=F, funlabel=c("3-year survival", "5-year survival", "10-year survival"),
maxscale=10, fun.at=c(0.95, 0.9, 0.85, 0.8, 0.75, 0.7, 0.6, 0.5))
plot(nom)
validate(f, method="boot", B=1000, dxy=T)
rcorrcens(Surv(time, status) ~ predict(f), data = bc)
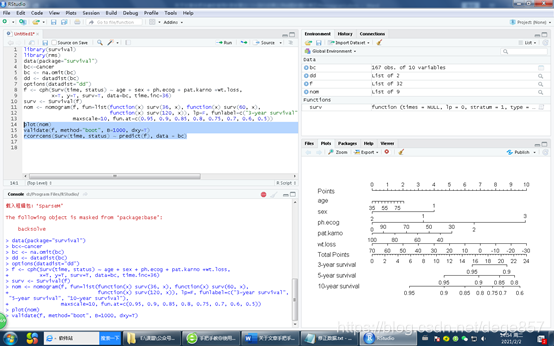
做到这一步已经做到内部验证做完区分度了,准备做校准度,原来代码是这样的,其实对图片修正很容易,我们需要根据原来数据把u,和m这两个参数调整一下,u代表你想看到多久的生存时间,注意f3的time.inc也要同步修改,m代表抽样的样本量
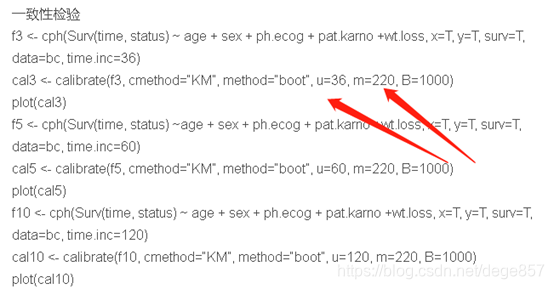
新改代码如下
f3 <- cph(Surv(time, status) ~ age + sex + ph.ecog + pat.karno +wt.loss, x=T, y=T, surv=T,
data=bc, time.inc=720)
cal3 <- calibrate(f3, cmethod="KM", method="boot", u=720, m=50, B=1000)
plot(cal3)
f5<- cph(Surv(time, status) ~ age + sex + ph.ecog + pat.karno +wt.loss, x=T, y=T, surv=T,
data=bc, time.inc=360)
cal5 <- calibrate(f5, cmethod="KM", method="boot", u=360, m=50, B=1000)
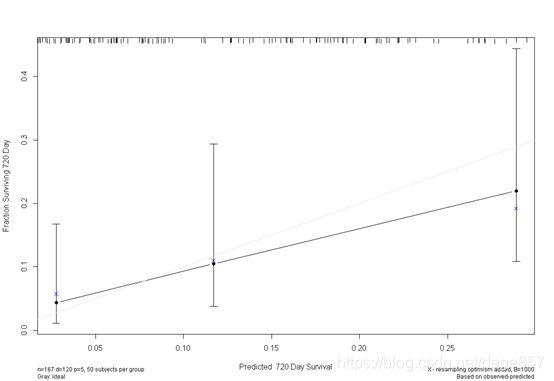
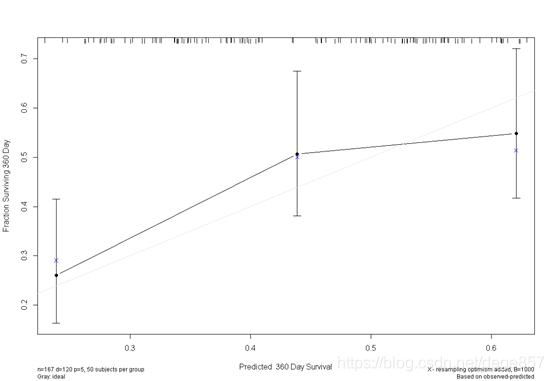
还可以美化一下,加点颜色
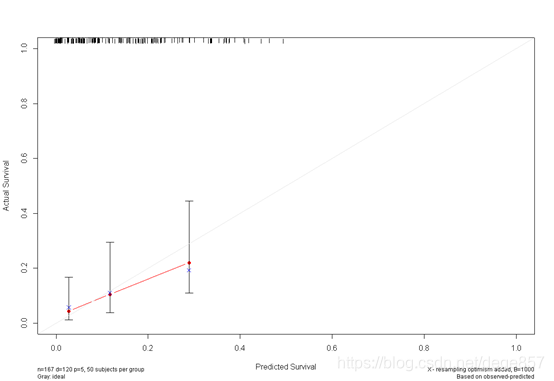
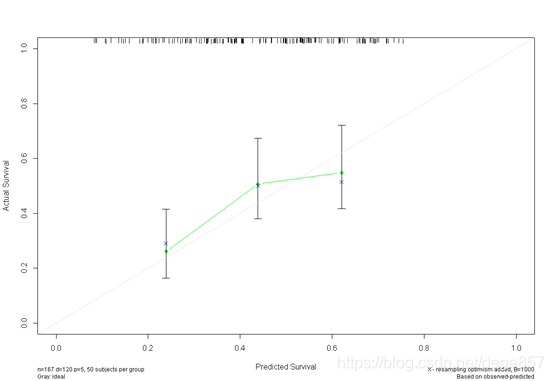
也可以把两张图合在一起
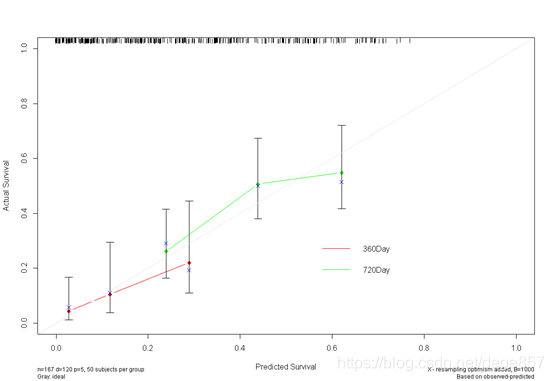
更多精彩文章请关注公众号:零基础说科研
