最近一个偶然的机会,我发现了一个可以在短短几秒内处理几十亿数据的python工具包:Vaex, 处于好奇我研究了一下Vaex,下面给大家简单介绍一下Vaex及其基本使用方法。
Vaex是什么
Vaex是用于惰性核心数据框架(类似于Pandas)的python库,用于可视化和探索大型表格数据集。它可以在N维表格上计算统计数据,例如均值,总和,计数,标准差等,最大可达十亿(109109)每秒的对象/行数。可视化使用直方图,密度图和3d体积渲染完成,从而可以交互式探索大数据。Vaex使用内存映射,零内存复制策略和惰性计算来获得最佳性能(不浪费内存)。
为什么选择vaex?
- 性能:适用于海量表格数据,处理速度>10^9 行/秒
- 惰性/虚拟列:动态计算,不浪费内存
- 内存效率:在执行过滤(选择)数据集的子集时没有内存副本。
- 可视化:直接支持,单线通常就足够了。
- 用户友好的API:您只需要处理DataFrame对象,而制表符补全和docstring可以帮助您:
ds.mean<tab>,感觉与Pandas非常相似。
总结一下:Vaex是一个高性能Python库,基于懒惰的Out-of-Core DataFrame(类似于Pandas),可以以可视化方式浏览大型表格数据集。它可以计算每秒超过十亿行的基本统计信息。它支持多种可视化,允许交互式探索大数据。
安装Vaex
Vaex的安装和别的python包的安装没什么两样:
pip install vaex
测试Vaex
下面我们来实际测试一下vaex吧,不过因为我们手上没有几十亿的大数据集,所以我们先让python生成一个10亿 行x 2列的随机数数据集,然后我们把这个数据集另存为一个csv文件,我们就用它来测试Vaex.
import vaex
import pandas as pd
import numpy as np
n_rows = 1000000000
n_cols = 2
df = pd.DataFrame(np.random.randint(0, 100, size=(n_rows, n_cols)), columns=['col%d' % i for i in range(n_cols)])
df.shape![]()
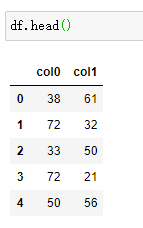
我们创建了一个只有两列的随机数dataframe,随机数的范围是100以内的整数。接下来我们要查询一下这个df占用了多少内存,因为我们是在单机上运行,随时监控一下内存的变化很重要,不然的话很有可能造成内存泄漏而死机。
df.info(memory_usage='deep')
我们发现这个超大的数据集已经消耗了14.9G的内存空间,还好我用机器是google的虚拟主机(内存30G)看来还够用。接下来我们把这个df另存为一个csv文件。
file_path = './data/big_file.csv'
df.to_csv(file_path, index=False)
我们发现这个csv文件占用了5.8G的磁盘空间。下面我们就用这个csv文件来测试我们的Vaex,这里需要说明的是由于Vaex能访问超大数据集是因为它采用的是内存映射和惰性的动态计算的工作方式,这和pandas的 dataframe 不一样,所以目前Vaex所支持的数据集文件的格式主要是 Apache Arrow 和 HDF5 两种数据格式,这里我们使用hdf5格式,因此我们需要将csv格式转化成hdf5格式:
file_path = './data/big_file.csv'
dv = vaex.from_csv(file_path, convert=True, chunk_size=10_000_000)执行上述代码后Vaex将分块读取CSV,并将每个块转换为磁盘上的临时HDF5文件。最后将所有临时文件合并成一个大的HDF5文件,然后删除所有临时文件。这里通过chunk_size参数指定要读取的单个块的大小。我这里将chunk_size设置为1千万表示生成的每个临时文件包含1千万条数据,不过最终这些临时文件都会被删除只保留一个经过合并后最终的HDF5文件。

我们发现最终生成的hdf5文件居然有16G超过原来的csv文件2倍多,并且在我的机器上耗时18分钟才完成转化。接下来我们来探索一下Vaex的基本功能,首先我们读取这个hdf5文件并生成一个df:
%%time
df = vaex.open('./data/big_file.csv.hdf5')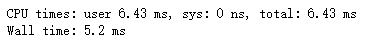
我们发现读取这个hdf5文件只花了5个毫秒,下面我们查看一下df的类型:
type(df)![]()
下面我们对第一列的10亿条数据求和:
%%time
col0_sum = df.col0.sum()
col0_sum
我们发现对第一列的10亿条数据求和只用了2秒。
轻松实现可视化
vaex可以项pandas的dataframe那样轻松实现可视化只要指定vaex的plot1d或者plot2d方法就可以了:
df.plot1d(df.col0, figsize=(14, 7))
数据过滤
Vaex具有零内存复制策略。 这意味着过滤DataFrame时只会占用很少的内存,并且不会复制数据。 看一下下面的列子:
%%time
df_filtered = df[(df.col0> 50) & ((df.col1<80))]
df_filtered
我们在这里创建的df_filtered 是不需要额外的内存!这是因为df_filtered只是原来df的一个浅层复制的副本。创建过滤的df_filtered时,Vaex将创建二元掩码(binary mask),然后将其应用于原始数据,而无需进行数据复制。这些过滤器的内存成本很低:一个过滤器需要约1.2 GB内存来过滤10亿行DataFrame。如果让pandas来过来10亿行的数据可能需要消耗100GB内存。
虚拟列
将Vaex DataFrame的现有列转换为新列会创建虚拟列。虚拟列就像普通的列一样,但是它们不占用任何内存。这是因为Vaex仅记住定义它们的表达式,而不预先计算值。仅在必要时才对这些列进行动态计算,这样以保持较低的内存使用率。来看下面的例子:
%%time
df['col2'] = df.col0 + df.col1
df
创建一个虚拟列耗时还不到1秒。
聚合操作
Vaex的聚合操作和pandas的略有不同, 更重要的是它的速度非常快。下面我们先创建一个虚拟列,然后再做聚合操作:
%%time
df ['col1_50'] = df.col1>= 50
df
Vaex在单个命令中组合了分组和聚合。下面的代码按“ col1_50”列对数据进行分组,然后计算col0列的总和。
%%time
df_group = df.groupby(df['col1_50'],agg = vaex.agg.sum(df['col0']))
df_group 
连接操作(join)
Vaex在不创建内存副本的情况连接(join)接数据集,这可以节省主内存。熟悉pandas的朋友应该熟悉join操作:
%%time
df_join = df.join(df_group,on ='col1_50')
df_join
总结
Vaex的大多数操作都类似于pandas,只不过vaex更适合处理大数据集(亿级),而且占用更少的内存,并且是在单机上操作。我使用的机器是google云的虚拟主机配置是8vCPU,30G内存,50G硬盘。如果你的内存只有16G或更小,那可以在创建测试数据集时适当的调整数据的维度,比如我的测试数据的维度是10亿行x2列,你可以创建100万行x1000列等,根据你自己机器的内存来调整测试数据的维度。