本文不对标准化和归一化相关原理和公式作阐述
标准化(均值移除)
由于一个样本的不同特征值差异较大,不利于使用现有机器学习算法进行样本处理。均值移除可以让样本矩阵中的每一列的平均值为0,标准差为1。
转换公式如下:
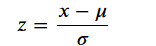
标准化的意义:
想象一下,我们经常通过梯度下降来进行优化求解,公式一般如下,如果特征之间的数值差异太大,那么更新的结果肯定也会产生较大的差异,这是我们所不希望的。在最开始的时候,我们认为特征之间的重要程度的是一样,并不想偏袒哪个特征,所以这部预处理工作必做!
import pandas as pd
import numpy as np
df = pd.read_csv(
'../data/wine_data.csv', #葡萄酒数据集
header=None, #用哪行当做列名,我们自己来指定
usecols=[0,1,2] #返回一个子集,我们拿部分特征举例就可以了
)
df.columns=['Class label', 'Alcohol', 'Malic acid']
df.head()
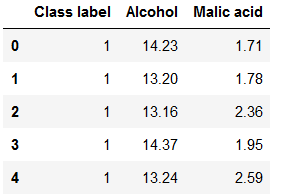
在数据中,Alcohol和Malic acid 衡量的标准应该是不同的,特征之间数值差异较大
from sklearn import preprocessing
std_scale = preprocessing.StandardScaler().fit(df[['Alcohol', 'Malic acid']])
df_std = std_scale.transform(df[['Alcohol', 'Malic acid']])
print('Mean after standardization:\nAlcohol={:.2f}, Malic acid={:.2f}'
.format(df_std[:,0].mean(), df_std[:,1].mean()))
print('\nStandard deviation after standardization:\nAlcohol={:.2f}, Malic acid={:.2f}'
.format(df_std[:,0].std(), df_std[:,1].std()))
#输出结果:
Mean after standardization:
Alcohol=-0.00, Malic acid=-0.00
Standard deviation after standardization:
Alcohol=1.00, Malic acid=1.00
归一化
另一种方法叫做 Min-Max scaling (或者叫做 "normalization"也就是我们常说的0-1归一化)。
有些情况每个样本的每个特征值具体的值并不重要,但是每个样本特征值的占比更加重要。
处理后的所有特征的值都会被压缩到 0到1区间上.这样做还可以抑制离群值对结果的影响. 归一化公式如下:
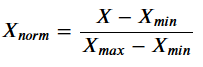
from sklearn import preprocessing
minmax_scale = preprocessing.MinMaxScaler().fit(df[['Alcohol', 'Malic acid']])
df_minmax = minmax_scale.transform(df[['Alcohol', 'Malic acid']])
print('Min-value after min-max scaling:\nAlcohol={:.2f}, Malic acid={:.2f}'
.format(df_minmax[:,0].min(), df_minmax[:,1].min()))
print('\nMax-value after min-max scaling:\nAlcohol={:.2f}, Malic acid={:.2f}'
.format(df_minmax[:,0].max(), df_minmax[:,1].max()))
#输出结果:
Min-value after min-max scaling:
Alcohol=0.00, Malic acid=0.00
Max-value after min-max scaling:
Alcohol=1.00, Malic acid=1.00
标准化处理对PCA主成分分析的影响
import pandas as pd
df = pd.read_csv(
'../data/wine_data.csv',
header=None,
)
df.head()
 ①将数据分为测试集和训练集
①将数据分为测试集和训练集
from sklearn.model_selection import train_test_split
X_wine = df.values[:,1:]
y_wine = df.values[:,0]
X_train, X_test, y_train, y_test = train_test_split(X_wine, y_wine,
test_size=0.30, random_state=12345)
②特征标准化
import sklearn.preprocessing as sp
std_scale = sp.StandardScaler().fit(X_train)
X_train_std = std_scale.transform(X_train)
X_test_std = std_scale.transform(X_test)
③使用PCA进行降维
分别在标准化和非标准化数据集上执行PCA,将数据集转换成二维特征子空间。
from sklearn.decomposition import PCA
# on non-standardized data
pca = PCA(n_components=2).fit(X_train)
X_train = pca.transform(X_train)
X_test = pca.transform(X_test)
# om standardized data
pca_std = PCA(n_components=2).fit(X_train_std)
X_train_std = pca_std.transform(X_train_std)
X_test_std = pca_std.transform(X_test_std)
④图像对比
import matplotlib.pyplot as plt
fig, (ax1, ax2) = plt.subplots(ncols=2, figsize=(10,4))
for l,c,m in zip(range(1,4), ('blue', 'red', 'green'), ('^', 's', 'o')):
ax1.scatter(X_train[y_train==l, 0], X_train[y_train==l, 1],
color=c,
label='class %s' %l,
alpha=0.5,
marker=m
)
for l,c,m in zip(range(1,4), ('blue', 'red', 'green'), ('^', 's', 'o')):
ax2.scatter(X_train_std[y_train==l, 0], X_train_std[y_train==l, 1],
color=c,
label='class %s' %l,
alpha=0.5,
marker=m
)
ax1.set_title('Transformed NON-standardized training dataset after PCA')
ax2.set_title('Transformed standardized training dataset after PCA')
for ax in (ax1, ax2):
ax.set_xlabel('1st principal component')
ax.set_ylabel('2nd principal component')
ax.legend(loc='upper right')
ax.grid()
plt.tight_layout()
plt.show()
 很明显,可以清晰的看到经过标准化的数据可分性更强的。
很明显,可以清晰的看到经过标准化的数据可分性更强的。