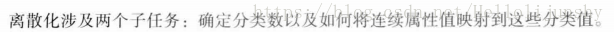数据集成:是将储存咋两个数据框内的数据,以“关键词”为依据,以行为单位做列向合并,是通过merge()函数实现,基本形式:merge(数据框1,数据框2,by="关键字")。
但是在数据集成中可能会有这些问题出现:
(1)同名异意:数据集A中的ID描述的是菜品单号,数据集B中的ID描述的是订单编号,则合并的话会出现问题。
(2)异名同意:数据集A中的data描述的是日期,数据集B中的dt描述的也是日期,则合并的话会出现问题。
(3)单位不统一:两个数据集描述的同一个实体,但是单位不同。
另外,各个变量之间的单位的量纲不同(单位不同),或者数值差别过大,若不进行处理,分析的结果可能会出错,所以,对数据做标准化很重要:
数据规范化代码实现:
# 设置工作空间
# 把“数据及程序”文件夹拷贝到F盘下,再用setwd设置工作空间
setwd("E:/自己重要的文件/R语言个人分类总结/R语言数据分析与挖掘实战/图书配套数据、代码/chapter4/示例程序")
# 读取数据
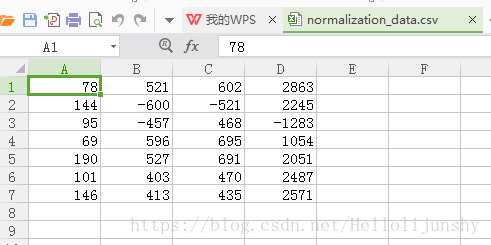
data <- read.csv('./data/normalization_data.csv', header = FALSE)
# 最小-最大规范化
b1 <- (data[, 1] - min(data[, 1])) / (max(data[, 1]) - min(data[, 1]))
b2 <- (data[, 2] - min(data[, 2])) / (max(data[, 2]) - min(data[, 2]))
b3 <- (data[, 3] - min(data[, 3])) / (max(data[, 3]) - min(data[, 3]))
b4 <- (data[, 4] - min(data[, 4])) / (max(data[, 4]) - min(data[, 4]))
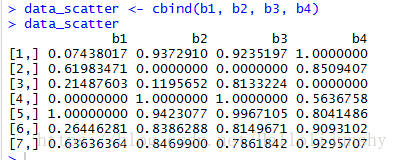
data_scatter <- cbind(b1, b2, b3, b4)
# 零-均值规范化
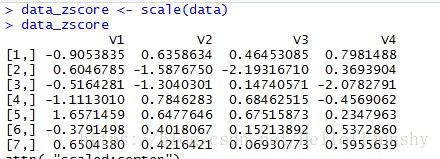
data_zscore <- scale(data)
i1 <- ceiling(log(max(abs(data[, 1])), 10)) # 小数定标的指数
c1 <- data[, 1] / 10 ^ i1
i2 <- ceiling(log(max(abs(data[, 2])), 10))
c2 <- data[, 2] / 10 ^ i2
i3 <- ceiling(log(max(abs(data[, 3])), 10))
c3 <- data[, 3] / 10 ^ i3
i4 <- ceiling(log(max(abs(data[, 4])), 10))
c4 <- data[, 4] / 10 ^ i4
data_dot <- cbind(c1, c2, c3, c4)
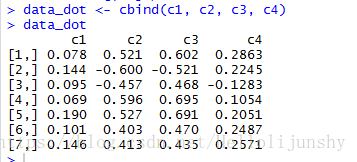
# 打印结果
options(digits = 4) # 控制输出结果的有效位数
data
data_scatter
data_zscore
data_dot
连续属性离散化:
一些数据挖掘算法(特别是分类算法ID3,Apriori算法等)要求数据类型是离散分类的,这时需要将连续属性变换为离散属性,这就是连续属性离散化。
2 常用的离散化方法有等宽法、等频法和聚类等
连续属性离散化的代码实现:
# 设置工作空间
# 把“数据及程序”文件夹拷贝到F盘下,再用setwd设置工作空间
setwd("E:/自己重要的文件/R语言个人分类总结/R语言数据分析与挖掘实战/图书配套数据、代码/chapter4/示例程序")
# 读取数据文件,提取标题行
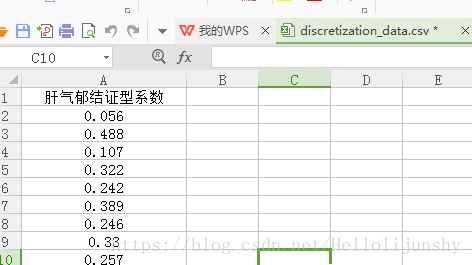
data <- read.csv('./data/discretization_data.csv', header = TRUE)
# 等宽离散化
v1 <- ceiling(data[, 1] * 10)
plot(data[, 1], v1, xlab = '肝气郁结证型系数') # 图示结果
# 等频离散化
names(data) <- 'f' # 变量重命名
attach(data)
seq(0, length(f), length(f) / 6) # 等频划分为6组
v <- sort(f) # 按大小排序作为离散化依据
v2 <- rep(0, 930) # 定义新变量, 930是 一共有930个样本数据。
for (i in 1:930) {
v2[i] <- ifelse (f[i] <= v[155], 1,
ifelse (f[i] <= v[310], 2,
ifelse (f[i] <= v[465], 3,
ifelse (f[i] <= v[620], 4,
ifelse (f[i] <= v[775], 5, 6)))))
}
detach(data)
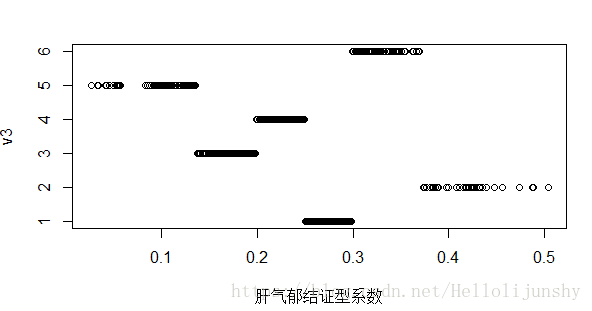
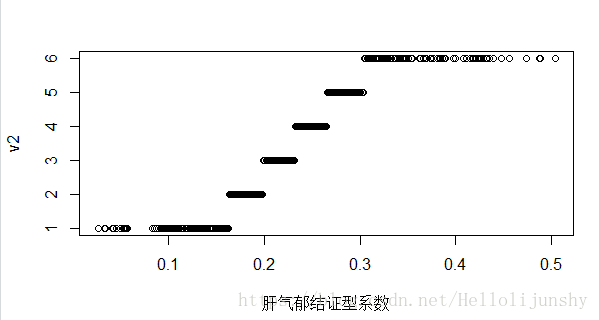
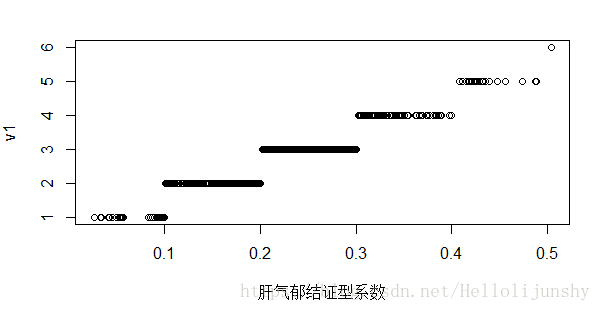
plot(data[, 1], v2, xlab = '肝气郁结证型系数') # 图示结果
上图就是说明:1,2,3,4,5,6类的个数都相等(虽然从图上看不出来,因为黑色部分与黑色部分肉眼比较不了个数)
# 聚类离散化result <- kmeans(data, 6)
v3 <- result$cluster
plot(data[, 1], v3, xlab = '肝气郁结证型系数') # 图示结果