欢迎来到课程二的第三个作业。在这个作业中,我们将使用Python来构建一些统计模型,这些模型是我们上周学到的,用于分析淋巴瘤患者数据集的生存估计。我们还将评估这些模型并解释它们的输出。在此过程中,您将学习以下内容:
- Censored(删失) Data
- Kaplan-Meier Estimates
- Subgroup Analysis
作业解析
作业名:
Survival Estimates that Vary with Time.ipynb
作业地址:
github --> bharathikannann/AI-for-Medicine-Specialization-deeplearning.ai --> AI for Medical Prognosis --> Week 3
导入包
import lifelines
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
from util import load_data
from lifelines import KaplanMeierFitter as KM
from lifelines.statistics import logrank_test
-
lifelines是一个开源的数据分析库,主要用于生存分析和可靠性分析。它提供了一些常见的生存分析工具,如Kaplan-Meier曲线、Cox比例风险模型等,方便用户进行生存数据的处理和分析。 -
numpy是Python科学计算中的基础包,提供了支持多维数组和矩阵运算的工具,是众多科学计算和数据分析库的基础 -
pandas是Python数据分析的核心库之一,主要用于数据清洗、处理、转换和分析。 -
matplotlib是Python中最流行的绘图库之一。
加载数据集
该实验用的数据集是 lifelines 提供的淋巴瘤数据
data = load_data()
print("data shape: {}".format(data.shape))
data.head()

"Time”一栏列出了病人在死亡或删失前活了多久。
"Event"栏显示是否观察到死亡。如果观察到事件(即患者死亡),则事件为1,如果数据删失,则事件为0。
这里的删失意味着没有任何观察到的事件就结束了。例如,让病人最多住院100天。如果患者在44天后死亡,其事件记录为Time = 44, event = 1。如果患者在100天后出院,3天后死亡(共103天),则在我们的流程中没有观察到该事件,对应的行Time = 100, event = 0。如果病人在入院后存活了25年,他们的数据仍然是Time = 100, Event = 0。
删失数据
我们可以绘制一个生存时间的直方图,看看在删失或事件发生之前,案例存活了多长时间。

练习1
编写一个计算删失数据比例的函数
def frac_censored(df):
"""
Return percent of observations which were censored.
Args:
df (dataframe): dataframe which contains column 'Event' which is
1 if an event occurred (death)
0 if the event did not occur (censored)
Returns:
frac_censored (float): fraction of cases which were censored.
"""
result = 0.0
### START CODE HERE ###
result =len(df[df["Event"]==0]["Event"])/len(df)
### END CODE HERE ###
return result
生存估计
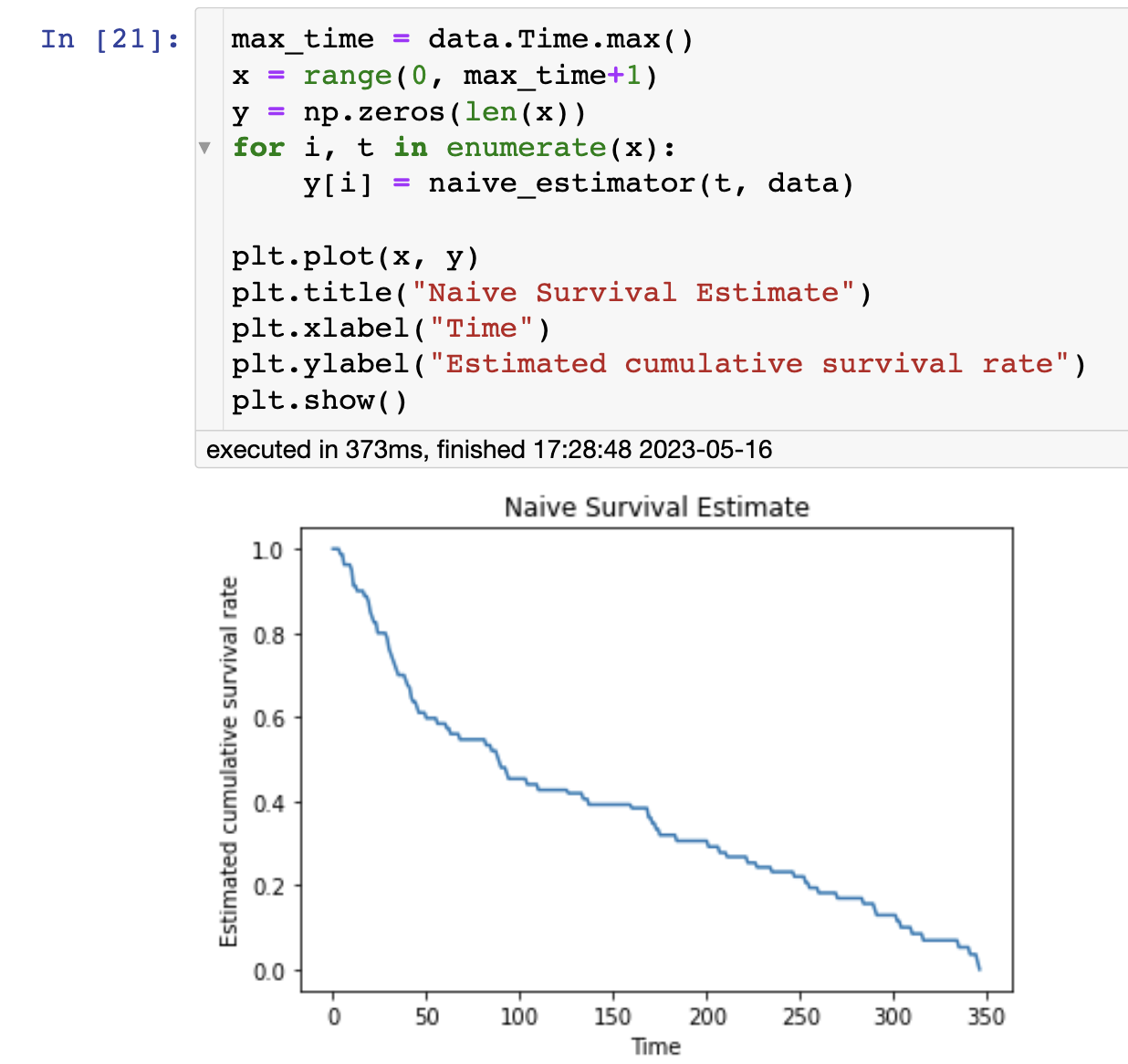
为了说明Kaplan Meier方法的优点,我们将从一个简单的估计器开始,用于估计上述生存函数。为了估计这个量,我们将计算时间 t 之后仍然存活的人数除以在时间 t 之前未删失的人数。
用公式表示为:
S ^ ( t ) = ∣ X t ∣ ( t 时间内活着的人) ∣ M t ∣ ( t 时间内除了删失的所有人) \hat{S}(t) = \frac{|X_t|(t时间内活着的人)}{|M_t|(t时间内除了删失的所有人)} S^(t)=∣Mt∣(t时间内除了删失的所有人)∣Xt∣(t时间内活着的人)
X t = { i : T i > t } X_t = \{i : T_i > t\} Xt={
i:Ti>t},
M t = { i : e i = 1 or T i > t } M_t = \{i : e_i = 1 \text{ or } T_i > t\} Mt={
i:ei=1 or Ti>t}.
M t M_t Mt的计算,需要注意的是,在t时间内删失的人(ei=0 且 Time < t)是不包括在内的,因为不知道它具体生存情况. 这里 M t M_t Mt的计算中, e_i = 1 包含两种情况,一种是在 t 时间前死的,一种是 t 时间后死的。而 T_i > t 也包含两种情况,一种是 t 时间后删失,一种是 t 时间后死亡。 显然二者是有重叠的,但是把它们合并在一起(逻辑: or),就可以得到在 t 时间内没有删失的总人数。
有点绕,自己捋捋~~
理解到了,看代码就很简单了
简单估计函数
def naive_estimator(t, df):
"""
Return naive estimate for S(t), the probability
of surviving past time t. Given by number
of cases who survived past time t divided by the
number of cases who weren't censored before time t.
Args:
t (int): query time
df (dataframe): survival data. Has a Time column,
which says how long until that case
experienced an event or was censored,
and an Event column, which is 1 if an event
was observed and 0 otherwise.
Returns:
S_t (float): estimator for survival function evaluated at t.
"""
S_t = 0.0
### START CODE HERE ###
X_t = len(df[df["Time"]>t])
M_t = len(df[(df["Time"]>t) | (df["Event"]==1)])
S_t = X_t/M_t
### END CODE HERE ###
return S_t
我们知道一个 t 怎么求生存率之后,接下来,使用 for 循环求连续 t 的生存率。
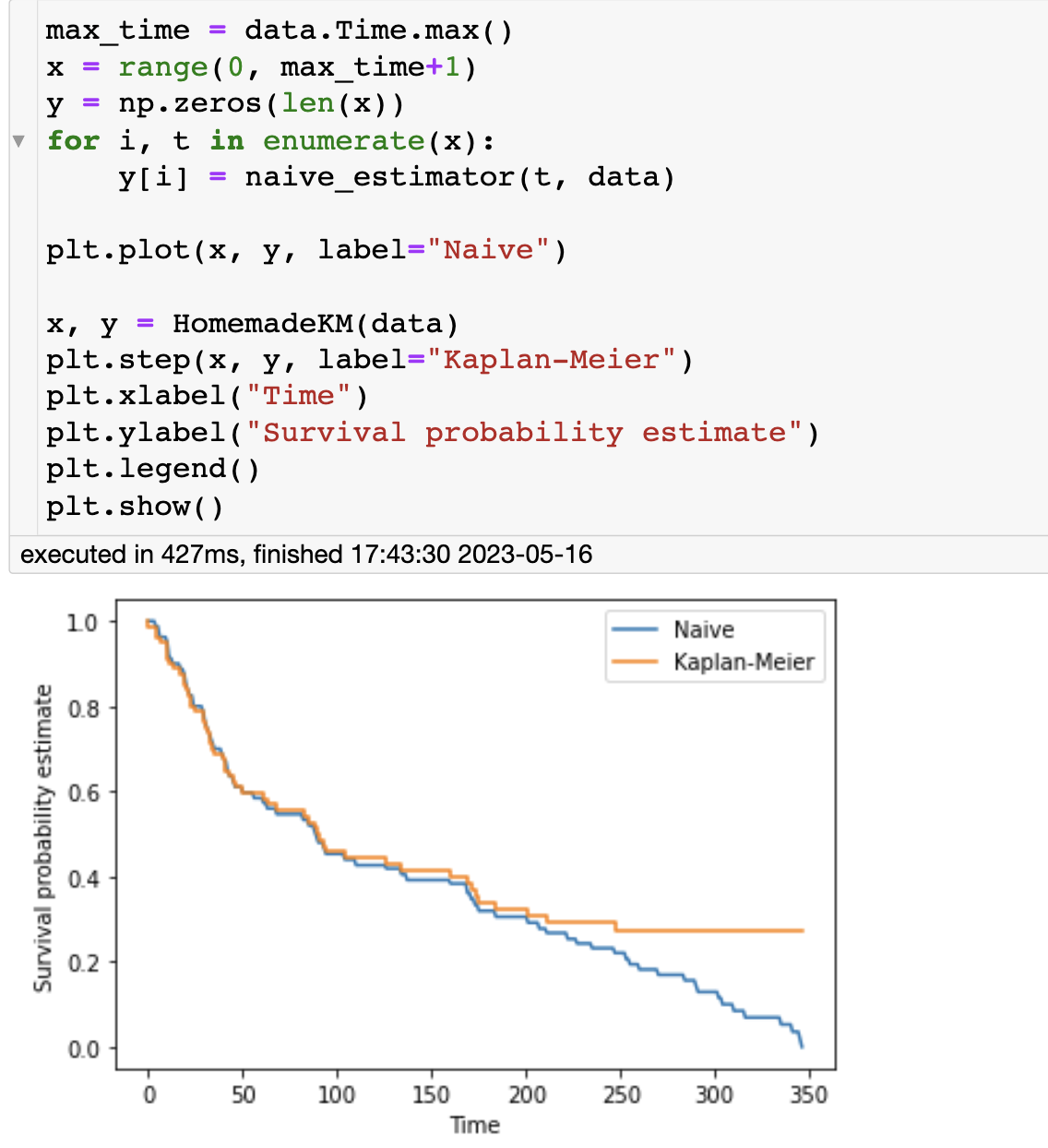
Kaplan Meier 估计
接下来我们将其与Kaplan Meier估计进行比较。在下面的单元格中,编写一个函数,计算数据集中每个不同时间 S ( t ) S(t) S(t)的Kaplan Meier估计。
回想一下Kaplan-Meier估计:
S ( t ) = ∏ t i ≤ t ( 1 − d i n i ) S(t) = \prod_{t_i \leq t} (1 - \frac{d_i}{n_i}) S(t)=ti≤t∏(1−nidi)
其中 t i t_i ti是在数据集中观察到的事件, d i d_i di是时间 t i t_i ti的死亡人数, n i n_i ni是我们知道在时间 t i t_i ti之前幸存下来的人数。
# UNQ_C3 (UNIQUE CELL IDENTIFIER, DO NOT EDIT)
def HomemadeKM(df):
"""
Return KM estimate evaluated at every distinct
time (event or censored) recorded in the dataset.
Event times and probabilities should begin with
time 0 and probability 1.
Example:
input:
Time Censor
0 5 0
1 10 1
2 15 0
correct output:
event_times: [0, 5, 10, 15]
S: [1.0, 1.0, 0.5, 0.5]
Args:
df (dataframe): dataframe which has columns for Time
and Event, defined as usual.
Returns:
event_times (list of ints): array of unique event times
(begins with 0).
S (list of floats): array of survival probabilites, so that
S[i] = P(T > event_times[i]). This
begins with 1.0 (since no one dies at time
0).
"""
# individuals are considered to have survival probability 1
# at time 0
event_times = [0]
p = 1.0
S = [p]
### START CODE HERE (REPLACE INSTANCES OF 'None' with your code) ###
# get collection of unique observed event times
observed_event_times = df["Time"].unique()
# sort event times
observed_event_times = sorted(observed_event_times)
# iterate through event times
for t in observed_event_times:
# compute n_t, number of people who survive to time t
n_t = len(df[df["Time"]>=t])
# compute d_t, number of people who die at time t
d_t = len(df[(df["Time"]==t) & (df["Event"]==1)])
# update p
p = p * (1 - (d_t/n_t))
S.append(p)
event_times.append(t)
# update S and event_times (ADD code below)
# hint: use append
### END CODE HERE ###
return event_times, S
子组分析
我们可以看到在时间和删失信息的列之外,数据集中还有一个名为Stage_group的列。
这个列中的数值1代表患有III期癌症的患者,2代表IV期。我们想比较这两组患者的生存函数。
这一次,我们将使用lifelines中的KaplanMeierFitter类。运行下面的代码块来拟合并绘制每组的Kaplan Meier曲线。
S1 = data[data.Stage_group == 1]
km1 = KM()
km1.fit(S1.loc[:, 'Time'], event_observed = S1.loc[:, 'Event'], label = 'Stage III')
S2 = data[data.Stage_group == 2]
km2 = KM()
km2.fit(S2.loc[:, "Time"], event_observed = S2.loc[:, 'Event'], label = 'Stage IV')
ax = km1.plot(ci_show=False)
km2.plot(ax = ax, ci_show=False)
plt.xlabel('time')
plt.ylabel('Survival probability estimate')
plt.savefig('two_km_curves', dpi=300)

注意: 对 KaplanMeierFitter 的使用不熟悉的,可以查看下面这篇
生存分析利器:Python 中的 Kaplan-Meier Fitter 类详解
比较一下两个组中,在相同时间的生存率有什么不同
survivals = pd.DataFrame([90, 180, 270, 360], columns = ['time'])
survivals.loc[:, 'Group 1'] = km1.survival_function_at_times(survivals['time']).values
survivals.loc[:, 'Group 2'] = km2.survival_function_at_times(survivals['time']).values

Log-Rank检验
为了说明生存曲线之间是否存在统计差异,我们可以进行log-rank检验。这个测试告诉我们,如果两条曲线相同,我们可以观察到这个数据的概率。log-rank检验的推导有点复杂,但幸运的是,lifeline有一个简单的函数来计算它。
def logrank_p_value(group_1_data, group_2_data):
result = logrank_test(group_1_data.Time, group_2_data.Time,
group_1_data.Event, group_2_data.Event)
return result.p_value
logrank_p_value(S1, S2)
结果=0.009588929834755544
众所周知,p<0.05具有统计学意义。
Congratulations!
你已经完成了课程二的第三个作业。你们学过Kaplan Meier估计量,生存分析中一个基本的非参数估计量。下周我们将学习如何在生存估计中考虑患者协变量